Hvad er ChIP?
Kromatinimmunopræcipitation, eller ChIP, er en antistofbaseret teknologi, der anvendes til selektivt at berige specifikke DNA-bindingsproteiner sammen med deres DNA-mål. ChIP anvendes til at undersøge en bestemt protein-DNA-interaktion, flere protein-DNA-interaktioner eller interaktioner på tværs af hele genomet eller en delmængde af gener.
ChIP anvender antistoffer, der selektivt genkender og binder proteiner, herunder histoner, histonmodifikationer, transkriptionsfaktorer og kofaktorer, for at give oplysninger om kromatintilstande og gentranskription. En kombination af proteomanalyse og molekylærbiologiske teknikker, der anvendes i ChIP, giver mulighed for at forstå genekspression og -regulering i celler eller væv af interesse.
Hvornår anvendes ChIP?
Typisk anvendes ChIP til at identificere den relative hyppighed af et specifikt protein eller en specifik proteinmodifikation i et bestemt område i genomet. ChIP kan bruges til at besvare en lang række videnskabelige spørgsmål, der vedrører interaktion mellem proteiner og kromatin. ChIP kan f.eks. bruges til at sammenligne tilstedeværelsen af visse proteiner på forskellige loci, kortlægge de forskellige proteiner på tværs af et genomisk område af interesse eller kvantificere proteinbinding til et inducerbart gen som reaktion på en stimulus over tid.
Hvordan virker ChIP?
Princippet bag ChIP er relativt ligetil og bygger på brugen af et antistof til at isolere eller udfælde et bestemt protein, histon, transkriptionsfaktor eller cofaktor og dets bundne kromatin fra en proteinblanding, der er blevet ekstraheret fra celler eller væv. Heraf navnet på denne teknik: Derfor navnet på teknikken: Chromatin Immunoprecipitation. I ChIP-PCR eller ChIP-seq kan immunberigede DNA-fragmenter derefter identificeres og kvantificeres ved hjælp af almindeligt tilgængelige PCR- eller qPCR-reagenser og Next Generation Sequencing (NGS)-teknologier.
Hvad er native ChIP (N-ChIP) vs. crosslinked ChIP (X-ChIP)?
Der er 2 typer ChIP-teknikker, der kan udføres afhængigt af det eksperimentelle spørgsmål og forsøgets udgangsmateriale: 1) native ChIP (N-ChIP) og 2) crosslinked ChIP (X-ChIP). Begge typer ChIP har fordele og ulemper:
- I N-ChIP anvendes der ikke noget fikseringsmiddel til at tværbinde proteinerne til kromatinet. I stedet isoleres naturligt kromatin fra cellekerner, der fordøjes med en nuklease. Fordi antistoffer opdrættes mod ufikserede antigener, giver N-ChIP den fordel, at antistoffer bedre kan genkende og binde sig til deres målantigener. PCR er muligvis ikke nødvendig til downstream-analyse på grund af den høje forekomst af histonproteiner. Selv om disse fordele gør N-ChIP til en attraktiv metode, kan den kun anvendes til påvisning af histoner. Desuden kan tab af proteinbinding under kromatinfordøjelsen og immunoprecipiteringstrinene forvrænge dataene eller hindre korrekte analyser.
- I X-ChIP anvendes kemiske fiksativer såsom formaldehyd til at krydsbinde det pågældende protein til DNA’et, og fragmentering af kromatin opnås ved sonicering eller nucleasefordøjelse. Fordelen ved X-ChIP er, at det kan anvendes med histon- og non-histonproteiner og generelt kræver mindre cellulært udgangsmateriale end N-ChIP. X-ChIP minimerer også risikoen for tab af kromatinprotein under ekstraktionen, hvilket gør det muligt at påvise forbigående proteininteraktioner. Udfældningstrinnet er dog mindre effektivt, og DNA-amplifikation ved PCR er nødvendig for downstream-analyser.
Hvad er de forskellige typer ChIP-assays?
Når selve kromatinimmunudfældningen er afsluttet, kan der foretages flere downstream-analyser af det oprensede kromatin og de tilknyttede proteiner, histoner, transkriptionsfaktorer og cofaktorer. De mest almindelige metoder til analyse af enkeltgener og analyser af hele genomet er henholdsvis qPCR og ChIP-seq. PCR og ChIP-chip er også muligheder for downstream-analyse.
5.1 Hvad er fordelene ved ChIP-PCR?
ChIP-PCR udføres for at analysere histonmodifikationer og/eller proteinbinding til en kendt delmængde af mål loci i genomet. Ved ChIP-PCR identificeres og kvantificeres immune berigede DNA-fragmenter ved hjælp af almindeligt tilgængelige PCR- eller qPCR-reagenser og -teknologier. Der kan opnås hurtige og kvantitative sammenligninger af specifikke regioner i genomet på tværs af flere prøver ved hjælp af ChIP-qPCR. Dette er billigere og mere tidseffektivt end metoder til sekventering af hele genomet.
5.2 Hvad er fordelene ved ChIP-chip?
ChIP-chip-teknologien henviser til udnyttelsen af en DNA-mikroarray-chip til analyse af ChIP-immunberigede DNA-fragmenter. Ved at anvende genom-tiling-mikroarray-teknologi kan der foretages en helgenom-analyse af proteiner, der er bundet til isoleret DNA, og der genereres et genomisk kort med høj opløsning over proteinbinding og proteinmodifikationer. ChIP-chip har flere anvendelsesmuligheder inden for grundforskning såvel som sygdomsbaseret forskning. Det kan f.eks. bruges til at identificere bindingsstederne for transkriptionsfaktorer, enhancers og repressorer og til at sammenligne disse typer af bundne proteiner i kontrol- og patologiske prøver. Da omkostningerne ved NGS imidlertid er faldet betydeligt, og lignende resultater opnås ved hjælp af ChIP-seq, vælger flere at udføre ChIP-seq i stedet for ChIP-chip.
5.3 Hvad er fordelene ved ChIP-seq?
I lighed med ChIP-chip giver ChIP-seq oplysninger om proteinbinding på tværs af genomet. I modsætning til ChIP-chip anvender ChIP-seq imidlertid NGS-teknologi til at identificere DNA-fragmenter og kortlægge dem i forhold til hele genomet.
Mere moderne DNA-amplifikationsteknologi gør det muligt at foretage robuste analyser på få dage med en lille mængde input-DNA. Når udgangsmaterialet er knapt, har disse teknologiske fremskridt i metoderne til biblioteksfremstilling gjort ChIP-seq-eksperimenter mulige.
Dertil kommer, at ny teknologi, hvor DNA-prøver er entydigt mærket med korte sekvenser, kendt som stregkoder, nu gør det muligt at samle individuelle fragmenter i en enkelt sekventeringsbane til multiplexanalyser. Dette har øget effektiviteten betydeligt og sænket omkostningerne ved DNA-seksekventeringseksperimenter, hvilket yderligere understøtter ChIP-seq-applikationer.
Sammenlagt er fordelen ved ChIP-seq på grund af fremskridt inden for DNA-sekventeringsteknologi, at et stort antal ChIP-berigede DNA-prøver kan sekventeres billigt på relativt kort tid med højere følsomhed og nøjagtighed end ChIP-chip.
Hvad er de forskellige trin i ChIP-assayet?
ChIP-assays følger en generel protokol:
- Krydsbinding af proteiner til DNA kun for X-ChIP
- Cellelysis
- Chromatinfragmentering ved fordøjning (for X-ChIP og N-ChIP) eller sonicering-(kun for X-ChIP)
- Immunopræcipitering ved hjælp af specifikke antistoffer
- DNA-oprensning med henblik på efterfølgende analyser
- DNA-analyse ved hjælp af PCR, qPCR, mikroarray eller NGS
Vigtigt er det, at positive og negative kontroller på hvert trin er afgørende for at afgøre, om et ChIP-forsøg har været vellykket.
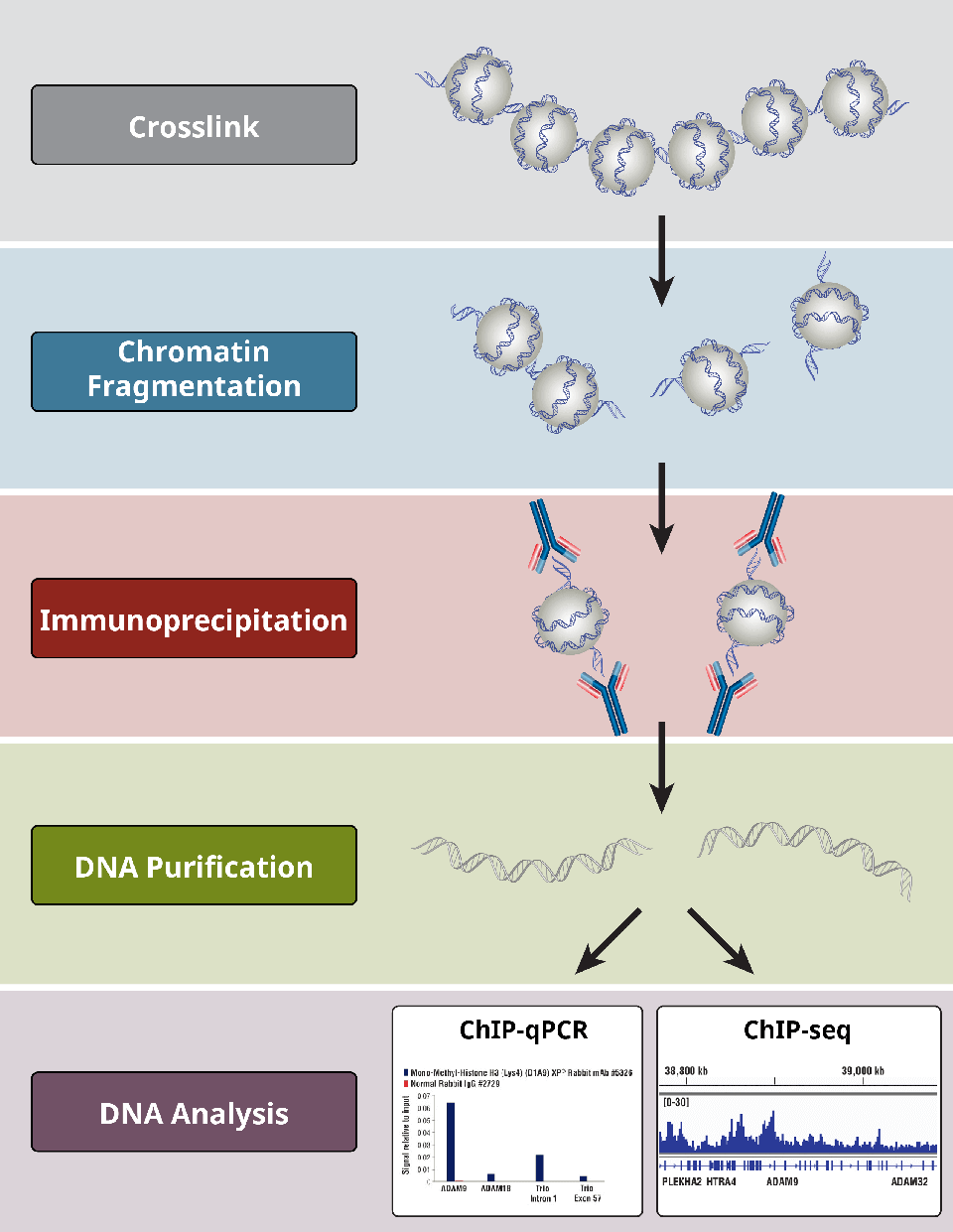
Oversigt over de mest kritiske trin i en ChIP-protokol.
Oversigt over de mest kritiske trin i en ChIP-protokol.
6.1 Hvordan krydskobler man celler og væv til ChIP?
Krydsningsreagenser bruges til at “fiksere” proteiner til det DNA, som de binder. Formaldehydbaserede reagenser anvendes typisk til at opnå denne fiksering. Celler og væv fikseres generelt på samme måde, men væv kræver en længere fikseringstid og en hurtigere fikseringsleverance for hurtigt at trænge igennem målvævet, før det begynder at degenerere.
Overfiksering af kromatin kan reducere effektiviteten af fragmenteringen ved sonicering, ud over at den hæmmer antistoffernes binding til deres proteinmål. Derfor bør fikseringstiden bestemmes empirisk for at muliggøre maksimal antistof-antigenbinding og samtidig opnå ideel krydsbinding af proteiner til deres mål-DNA.
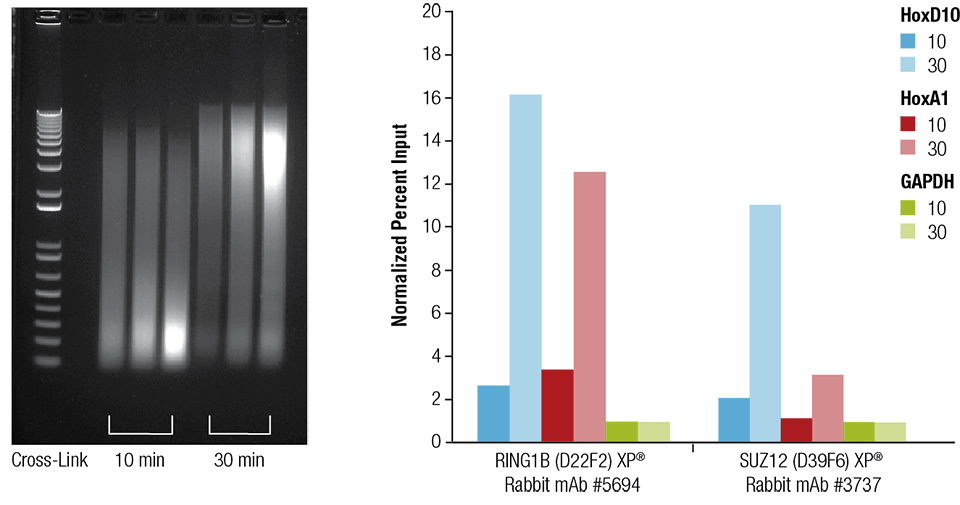
Mus hjerte (H), hjerne (B) og lever (L) blev krydsbundet i 10 eller 30 minutter som angivet (venstre panel). Kromatinet blev forberedt og sonicated i 4 minutter. ChIP blev udført ved hjælp af kromatin fremstillet fra hjertevæv med de angivne antistoffer ved hjælp af SimpleChIP® Plus Sonication Chromatin IP Kit #56383, og det berigede DNA blev kvantificeret ved realtids-PCR ved hjælp af primere til de angivne gener (højre panel). Mængden af immunoprecipiteret DNA i hver prøve er repræsenteret som normaliseret signal i forhold til den negative loci GAPDH, som er lig med 1.
Musehjerte (H), hjerne (B) og lever (L) blev tværbundet i 10 eller 30 minutter som angivet (venstre panel). Kromatinet blev forberedt og sonicated i 4 minutter. ChIP blev udført ved hjælp af kromatin fremstillet fra hjertevæv med de angivne antistoffer ved hjælp af SimpleChIP® Plus Sonication Chromatin IP Kit #56383, og det berigede DNA blev kvantificeret ved realtids-PCR ved hjælp af primere til de angivne gener (højre panel). Mængden af immunoprecipiteret DNA i hver prøve er repræsenteret som normaliseret signal til den negative loci GAPDH, som er lig med 1.
6.2 Hvordan fragmenterer man kromatin?
Kromatinfragmentering er afgørende for, at et ChIP-eksperiment lykkes. Fragmentering af kromatin er nødvendig for at solubilisere kromatinet og muliggøre dets coprecipitation. Desuden afhænger opløsningen af ChIP-assayet af fragmenteringen af kromatinet, da DNA-fragmentstørrelsen bestemmer opløsningen af ChIP-assayet.
Den enzymatiske fordøjelse anvender mikrococcal nuclease (MNase), som kløver dobbeltstrenget DNA mellem nukleosomer for at generere kromatinfragmenter. Mens en fuldstændig MNasefordøjelse genererer DNA-fragmenter på 150 basepar (mononukleosomer), genererer en ufuldstændig fordøjelse DNA-fragmenter på mellem 150 og 750 basepar (mono-, di- og tri-nukleosomer). Ved sonicering anvendes mekanisk kraft til at fragmentere kromatin. Sonicering fragmenterer kromatin mellem og inden for nukleosomer og frembringer en række kromatinfragmenter mellem 150 og 1000 basepar.
Til X-ChIP anvendes enten enzymatisk fordøjelse eller sonicering til at skære kromatinet. Sonikationsbetingelserne i sonikations-ChIP-protokollen bør bestemmes empirisk, da de varierer alt efter celletype og eksperimentel tilstand. Digestionsbetingelserne er mere konsistente på tværs af forskellige celletyper og væv, men kromatinfragmentstørrelsen bør stadig analyseres før IP.
For N-ChIP anvendes nukleaser til at fragmentere kromatinet for at opretholde proteinbindingen i ufikserede prøver. Nukleasefragmenteringen bør også bestemmes empirisk for at minimere overfordøjelsen af kromatinet.
6.3 Hvorfor bruge enzymatisk fordøjelse til ChIP?
Nukleasefordøjelse skal bruges til N-ChIP, da proteinerne ikke er tværbundet til DNA’et, og de barske betingelser, der er forbundet med soniceringsbaseret fragmentering, ville resultere i dissociation af kromatinproteinerne fra DNA’et. N-ChIP er ideel til analyse af histonprotein-DNA-interaktioner, fordi histon-DNA-bindingen er meget stærk og stabil. N-ChIP fungerer imidlertid ikke godt til analyse af chromatinbinding af transkriptionsfaktorer og kofaktorer.
Enten enzymatisk fordøjelse eller sonicering kan bruges til at fragmentere chromatin i X-ChIP. Fordelene ved enzymatisk fordøjelse omfatter konsistensen af fragmenteringen og milde fragmenteringsbetingelser (lavere varme og detergent), der bedre bevarer integriteten af kromatinet og antistofepitoper, hvilket resulterer i øget immunberigelse af transkriptionsfaktor- og kofaktorbundet kromatin.
6.4 Hvorfor bruge sonicering til fragmentering af kromatin til ChIP?
I modsætning til den kromatinfragmentering, der opnås ved enzymatisk fordøjelse, er sonicering afhængig af mekaniske kræfter til at fragmentere kromatin i mindre stykker. Den ideelle størrelse af kromatinfragmenter til immunberigelse er mellem 200 og 1000 basepar. Sonicering er den traditionelle metode til fragmentering af kromatin og kan udføres ved hjælp af en traditionel sonicator eller mere avancerede sonicatorer med vandbad, der giver en mere fokuseret sonicering. Sonicering genererer virkelig randomiserede kromatinfragmenter; det kræver imidlertid omfattende optimering på tværs af forskellige cellelinjer og væv og er vanskeligt at reproducere fra eksperiment til eksperiment. Kravet om høje detergentbuffere og den varme, der genereres under sonicering, kan skade integriteten både af kromatinet og antistofepitoper på kromatinproteiner.
6.5 Optimering af kromatinsonicering til ChIP
Soniceringsbaseret kromatinfragmentering anvender traditionelt høje detergentbuffere og genererer varme, hvilket begge kan skade integriteten af kromatinet og antistofepitoper. Derfor skal mængden af sonicering, der anvendes til fragmentering af kromatin, bestemmes eksperimentelt for forskellige cellelinjer og væv. Man skal identificere og anvende den minimale mængde sonicering, der er nødvendig for at generere 150 til 1000 basepar DNA-fragmenter for at minimere skader på kromatinet.
Hvor man går i gang med en fuld ChIP-analyse med efterfølgende analyse ved qPCR, DNA-chip eller NGS, bør man anvende gelelektroforese til at analysere kromatinprøver, der er soniceret i forskellige tider. Fragmentstørrelsen afhænger af soniceringstiden – fragmentstørrelsen aftager, når soniceringstiden øges. Data tyder dog på, at længere soniceringstider ikke fører til bedre resultater. Derfor er kørsel af renset immunoprecipiteret DNA på en gel og bestemmelse af den ideelle fragmentstørrelse en ukompliceret metode til bestemmelse af den minimale soniceringstid, der er nødvendig for at opnå den ønskede DNA-størrelse, og til at undgå unødig beskadigelse af kromatin.

Enzymfordøjet kromatin blev kørt på en agarosegel. Lane 1 viser kromatin, der er underfordøjet. Lane 2 viser korrekt fordøjet kromatin, og Lane 3 viser kromatin, der er overfordøjet.
Enzymfordøjet kromatin blev kørt på en agarosegel. Lane 1 viser kromatin, der er underfordøjet. Lane 2 viser korrekt fordøjet kromatin, og Lane 3 viser kromatin, der er overfordøjet.
6.6 Hvordan vælger man et antistof til ChIP?
Valg af et passende antistof til et ChIP-eksperiment er afgørende for, om det lykkes. Antistoffer, der anvendes i et ChIP-forsøg, skal være specifikke for det pågældende protein og have en høj affinitet for antigenet. Det bedste valg af antistof til et ChIP- eller ChIP-seq-eksperiment er et ChIP- eller ChIP-seq-valideret antistof. Hvis der ikke er noget ChIP-valideret antistof til rådighed for det pågældende gen, er det næstbedste valg et antistof, der er blevet valideret i IP. Det er vigtigt at bemærke, at ikke alle IP-validerede antistoffer virker i ChIP, og at ikke alle ChIP-validerede antistoffer virker i ChIP-seq. Desuden gælder det, at jo mere et antistof er valideret på tværs af andre anvendelser, f.eks. western, IP, IF, flow og IHC, jo mere tillid kan man have til antistoffets ydeevne og specificitet. Valideringen af et antistof skal følges op af en empirisk bestemmelse af den optimale koncentration af antistoffet sammen med IP-vaskbetingelser.
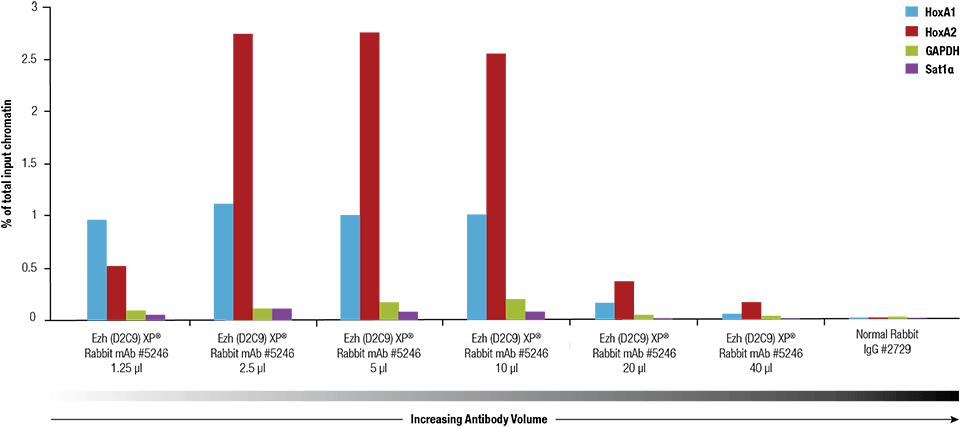
Ezh2 (D2C9) XP® Rabbit mAb #5246 blev titreret ved hjælp af SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 på krydsbundet kromatin fremstillet fra 4 x 106 NCCIT-celler.
Ezh2 (D2C9) XP® kanin mAb #5246 blev titreret ved hjælp af SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 på krydsbundet kromatin fremstillet fra 4 x 106 NCCIT-celler.
6.7 Sådan udføres immunudfældningen
Antistoffer anvendes til at fange et protein af interesse og dets bundne DNA. Antistofkoncentrationen bør bestemmes empirisk, med et generelt udgangspunkt på 0,5-2,0 μg antistof anvendt pr. 10 μg kromatin-DNA (svarer til ca. 4 x 106 celler). Strengheden af buffere og vasketider bør også bestemmes empirisk, da de afhænger af antistoffets affinitet til dets målantigen. Typisk udføres antistof:kromatin-inkuberinger i 2 timer til natten.
Antistof-antigen (+DNA)-komplekser er affinitetsindfanget på en antistofbindende harpiks. I ChIP-forsøg består denne harpiks typisk af magnetiske, sepharose- eller agaroseperler af ChIP-kvalitet, der er konjugeret til protein A og/eller protein B. Antistoffer binder protein A- og/eller protein G-perler med forskellig affinitet afhængigt af den art, de er udviklet i, og IgG-subtypen af deres tunge kæde. Perlerne inkuberes typisk med antistof:kromatin i 2-4 timer.
Der er behov for vasketrin for at fjerne kromatin, der ikke er bundet til et antistof, efterfulgt af omvending af tværbindinger (til X-ChIP) og rensning af DNA. Desuden skal der udføres en IgG-kontrol-IP for at bestemme baggrunden (signal:støj). Positive kontrolantistoffer (dvs. histon H3 i alt) og/eller positive kontrol qPCR-primere (for kendte positive og negative målproteinbindende loci) skal også medtages for at bestemme uspecifik binding. For at opnå optimale resultater bør QC af kromatin-IP’en ved qPCR udføres før nedstrøms NGS-analyse.
6.8 Sådan elueres kromatinet fra protein A/G-perlerne
Kromatinet elueres fra protein A/G-perlerne ved hjælp af detergent og varme. Der kræves “vortexing” eller blanding ved lav hastighed for at holde perlerne i suspension og for at øge elueringen af kromatinet.
6.9 Hvordan kromatinkrydsforbindelserne omvendes
Krydsforbindelserne omvendes ved høj varme og højt salt (begge dele er vitale komponenter). Proteinase K tilsættes også for at fordøje de tilknyttede kromatinproteiner og tilsatte antistoffer, hvilket giver mulighed for mere effektiv nedstrøms DNA-oprensning.
6.10 Sådan oprenses DNA
Når kromatin-krydsforbindelserne er fjernet, oprenses DNA enten ved hjælp af klassiske phenol-chloroform efterfulgt af ethanoludfældningsmetoder eller ved hjælp af kolonnebaserede DNA-oprensningssæt.
Hvordan analyseres beriget DNA
Når DNA er oprenset, kan der udføres flere downstream-analyser, herunder ChIP-PCR, ChIP-qPCR, ChIP-chip og ChIP-seq.
7.1 ChIP-PCR- og ChIP-qPCR-analyse
ChIP-PCR- og ChIP-qPCR-analyser er bedst til enkeltgenanalyse og kan bruges til at amplificere og kvantificere specifikke DNA-fragmenter på en hurtig og omkostningseffektiv måde.
7.2 ChIP-chip-analyse
ChIP-chip-analyse anvender tiling DNA-mikroarray-chips til at skabe et genomdækkende kort med høj opløsning over proteinbinding og proteinmodifikation.
7.3 ChIP-seq-analyse
ChIP-seq-analyse anvender standard NGS-teknologi til at tilpasse renset DNA med tidligere annoterede hele genomer for at identificere proteinbindingsprofiler på tværs af hele genomet.