Genomische Merkmale von A. brassicae und zwei anderen koinfizierenden A. alternata-Isolaten
Wir sequenzierten die Genome von zwei Isolaten von A. alternata (PN1 und PN2), die B. juncea mit A. brassicae koinfizierten. Der Aufbau von A. brassicae wurde bereits beschrieben. Es bestand aus neun vollständigen Chromosomen und einem Chromosom, bei dem an einem Ende telomere Wiederholungen fehlten. Abgesehen von diesen Chromosomen gab es sechs Contigs, von denen eines ~ 1 Mb groß war, die zusammen ein entbehrliches Chromosom darstellen könnten (Abb. 1). Die N50 der A. brassicae-Assembly betrug 2,98 Mb (Tabelle 1). Die beiden Isolate, die B. juncea koinfizieren, wurden auf der Grundlage ihrer ITS- und GAPDH-Sequenzen als A. alternata identifiziert. Die A. alternata-Assemblies Aat_PN1 und Aat_PN2 bestanden aus 14 Contigs mit einer Gesamtgröße von 33,77 Mb bzw. 15 Contigs mit einer Gesamtgröße von 33,53 Mb (Tabelle 1). Sechs Contigs in jeder der beiden Assemblies enthielten telomere Wiederholungen an beiden Enden und repräsentieren daher höchstwahrscheinlich vollständige chromosomale Moleküle. Vier weitere Contigs in beiden Assemblies enthielten telomere Repeats an einem Ende, waren aber von ähnlicher Größe wie vollständige Chromosomenmoleküle, wie sie bei A. solani beschrieben wurden. Daher repräsentieren die Genomassemblies für A. alternata-Isolate jeweils zehn nahezu vollständige Chromosomen der beiden Isolate.
Ganzgenom-Alignments mit verwandten Alternaria spp. zeigten eine allgemeine Syntenie zwischen den Genomen mit geringfügigen Umlagerungen (Abb. 2). Zusätzlich wurden aus den Sequenzierungsdaten für die beiden Isolate von A. alternata auch mitochondriale Sequenzen gewonnen. Die mitochondrialen Genome der A. alternata-Stämme waren etwa 49 783 bp bzw. 50 765 bp groß und wiesen eine hohe Ähnlichkeit mit dem zuvor veröffentlichten mitochondrialen Genom von A. alternata auf.
Die Genvorhersage nach der Maskierung von Wiederholungen führte zur Identifizierung von 11593, 11495 und 11387 Genen in den Genomassemblies von A. brassicae, A. alternata PN1 bzw. PN2. Dies war vergleichbar mit den geschätzten Genzahlen in anderen Alternaria spp. (Tabelle 1). Die BUSCO-Analyse zeigte, dass die in den drei Genomen vorhergesagten Genmodelle 98 % der in einer Kopie konservierten Pilzgene abdeckten, was auf eine annähernde Vollständigkeit der Assemblies hinweist. Die vorhergesagten Gene wurden mit Hilfe einer Kombination von Datenbanken, wie im Abschnitt Methoden beschrieben, umfassend annotiert (Abb. 1). Zusätzlich zu den drei Genomen haben wir auch Gene de novo in den Genomassemblies von drei anderen Alternaria-Arten vorhergesagt, die mit Long-Read-Technologien sequenziert wurden, nämlich A. brassicicola (abra43), A. alternata (ATCC34957) und A. solani (altNL03003) (Tabelle 1). Diese sechs Genome und ihre Genvorhersagen wurden für die vergleichende Analyse von Sekundärmetabolit-kodierenden Genclustern und Effektor-kodierenden Genen verwendet.

Zusammenfassung des A. brassicae-Genoms, (von außen nach innen) a Pseudochromosomen/Gerüste, b Protein-kodierende Gene, c Repeat-Elemente, d Transponierbare Elemente (DNA und LTR), e vorhergesagte Sekundärmetabolit-Cluster, f sezernierte Proteine, g vorhergesagte Effektoren
Die phylogenomische Analyse ordnet eine separate Klade für die Brassica-infizierenden A. brassicae und A. brassicicola innerhalb der Gattung Alternaria
Um die Divergenz und die Verwandtschaft zwischen A. brassicae, den beiden A. alternata-Isolaten (PN1 und PN2) und den anderen Alternaria-Arten genau zu rekonstruieren, führten wir phylogenomische Analysen unter Verwendung von 29 Einzelkopie-Orthologen durch, die das höchste phylogenetische Signal aufwiesen, wie vom Programm Mirlo berechnet. Die Auswahl von Genen mit höheren phylogenetischen Signalen führt zu Phylogenien, die besser mit dem Artenbaum übereinstimmen. Die sich daraus ergebende Phylogenie zeigte, dass die großsporigen Alternaria- und die kleinsporigen Alternaria-Arten getrennt in zwei verschiedenen Kladen geclustert wurden (Abb. 3). Interessanterweise gruppierten sich die beiden Hauptpathogene der Brassicas, nämlich A. brassicae und A. brassicicola, getrennt von allen anderen Alternaria-Arten, was möglicherweise auf einen unterschiedlichen Evolutionspfad hinweist, der auf den gemeinsamen Wirtspräferenzen dieser beiden Arten beruht.
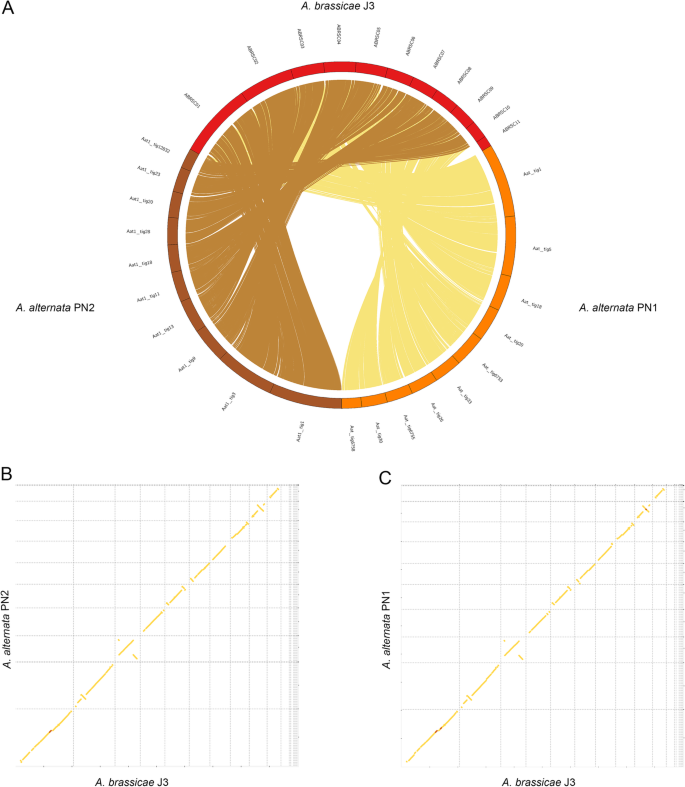
Gesamtgenom-Alignments von A. alternata PN1 und PN2 mit A. brassicae. a Circos-Plot, der die Makrosyntenie von A. alternata PN1 und PN2 mit A. brassicae über alle Contigs außer den entbehrlichen Contigs (ABRSC11, scaffold13,17,18,19), b und c Syntenic Dotplots von A. brassicae mit A. alternata PN1 und PN2
Vergleichende Analysen von A. alternata-Isolate aus verschiedenen Wirten
Wir haben die Genome von A. alternata PN1 und PN2 (isoliert aus B. juncea) mit denen von A. alternata ATCC34957 (isoliert aus Sorghum) verglichen, um etwaige Unterschiede in ihrem Genomgehalt zu ermitteln, die es ihnen ermöglichen könnten, zwei sehr unterschiedliche Arten zu infizieren. Ganzgenom-Alignments von A. alternata PN1 und PN2 mit dem von A. alternata ATCC34957 ergaben ein sehr hohes Maß an Syntenie und das Fehlen von artspezifischen Regionen. Wir identifizierten 719, 152 bzw. 586 isolatspezifische Gene zwischen den drei A. alternata-Isolaten (Zusatzdatei 1: Tabelle S1). Bei mehr als zwei Dritteln der isolatspezifischen Gene in allen drei Isolaten handelte es sich um uncharakterisierte Proteine oder um Gene, die nicht annotiert waren. Bemerkenswert ist, dass alle drei Isolate keine entbehrlichen Chromosomen enthielten, die möglicherweise Pathogenität verleihen, wie dies für A. alternata-Isolate, die viele Obstkulturen wie Zitrusfrüchte, Birnen und Äpfel infizieren, berichtet wurde. Das Genrepertoire der drei Isolate umfasste auch eine ähnliche Anzahl und Art von Effektoren, CAZymen und Sekundärmetaboliten-Clustern (Tabelle 2). Darüber hinaus verursachen die beiden Isolate PN1 und PN2 unter epiphytotischen Bedingungen keine eigenen Infektionssymptome bei B. juncea (Daten nicht gezeigt). Unsere Ergebnisse deuten darauf hin, dass es sich bei diesen Isolaten von A. alternata (PN1 und PN2) um fakultative Pathogene handeln könnte, die eine saprophytische Lebensweise führen und unter bestimmten Umweltbedingungen zu einer pathogenen Lebensweise übergehen können.
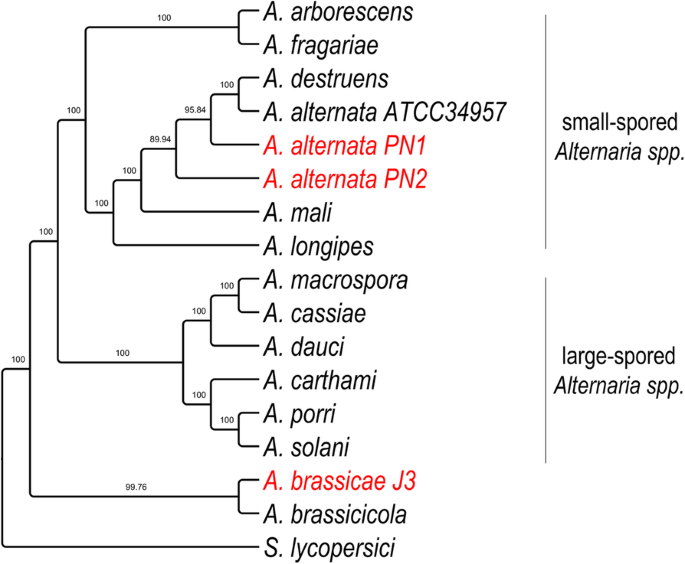
Phylogenetischer Baum der Alternaria-Arten mit S. lycopersici als Außengruppe. Der Baum wurde unter Verwendung von 29 Einzelkopie-Orthologen konstruiert, die das höchste phylogenetische Signal aufweisen, wie in Mirlo berechnet. Die Zweigunterstützungswerte aus 1000 Bootstrap-Wiederholungen werden gezeigt
Eine Fülle von wiederholungsreichen Regionen und transponierbaren Elementen in A. brassicae
Filamentöse Pflanzenpathogene neigen dazu, eine ausgeprägte Genomarchitektur mit höherem Repeat-Gehalt zu haben. Die Schätzung des Repeat-Gehalts und die Maskierung mit RepeatModeler und RepeatMasker ergaben, dass das Genom von A. brassicae aus ~ 9,33 % Repeats besteht, verglichen mit 2,43 und 2,64 % Repeats in den Genomen von A. alternata. Das Genom von A. brassicae weist unter allen bisher sequenzierten Alternaria-Arten den höchsten Repeat-Gehalt (~ 9,33 %) auf. Unsere Analyse ergab, dass sich der Wiederholungsgehalt zwischen den A. alternata-Isolaten und den anderen pathogenen Alternaria-Arten deutlich unterscheidet. Die pathogenen Alternaria-Arten, insbesondere A. brassicae und A. brassicicola, wiesen im Vergleich zu den anderen (pathogenen und nicht-pathogenen) A. alternata-Isolaten ein wesentlich größeres Repertoire an LTR/Gypsy- und LTR/Copia-Elementen auf (> 8X) (Abb. 4). Die Genome von A. brassicae und A. brassicicola wiesen auch eine Überrepräsentation von DNA-Transposons auf, die ~ 5 % des Genoms ausmachten, im Vergleich zu < 1 % bei den anderen Alternaria-Arten (Abb. 4).

Vergleich des Wiederholungsgehalts bei sechs Alternaria-Arten. Die Größe der Blasen entspricht (a) dem prozentualen Anteil transponierbarer Elemente (TEs) im Genom, (b) der Kopienzahl der TE im Genom
Diese Vermehrung repetitiver DNA und die anschließende Evolution von Genen, die diese Regionen überlappen, könnte der Schlüssel zum evolutionären Erfolg sein, durch den es diesen Krankheitserregern gelungen ist, über Generationen von koevolutionären Konflikten mit ihren Wirten zu überleben. Die Nähe zu TEs setzt die Gene potenziell der Gefahr von durch Wiederholungen induzierten Punktmutationen (RIP) aus und beschleunigt somit die Evolution. Ektopische Rekombination zwischen ähnlichen TEs kann auch zu neuen Genkombinationen führen und dadurch die Vielfalt von Proteinen oder Metaboliten erhöhen.
Vorhandensein eines entbehrlichen Chromosoms in der großsporigen A. brassicae
Linienspezifische (LS) Chromosomen oder entbehrliche Chromosomen (DC) wurden von mehreren phytopathogenen Arten einschließlich A. alternata berichtet. DCs in A. alternata sind dafür bekannt, dass sie dem Isolat Virulenz und Wirtsspezifität verleihen. Die Ganzgenom-Alignments von A. brassicae mit anderen Alternaria spp. zeigten, dass ein Contig von ca. 1 Mb zusammen mit anderen kleineren Contigs (66-366 kb) spezifisch für A. brassicae war und keine Syntenie zu irgendeiner Region in anderen Alternaria spp. aufwies. Dies veranlasste uns zu der Hypothese, dass diese Contigs zusammen ein DC von A. brassicae darstellen könnten. Um dies zu bestätigen, suchten wir in den Contigs nach dem Vorhandensein von AaMSAS- und ALT1-Genen, die als Markergene für verzichtbare Chromosomen bei Alternaria spp. bekannt sind. Wir fanden zwei Kopien des AaMSAS-Gens als Teil von zwei Clustern für die Biosynthese von Sekundärmetaboliten auf dem 1-MB-Contig. Wir haben jedoch kein Homolog des ALT1-Gens gefunden. Darüber hinaus wurde der Wiederholungsgehalt der Contigs (ABRSC11, Scaffold 13, 17, 18 und 19) mit dem des gesamten Genoms verglichen. Der Gengehalt der stammbaumspezifischen Contigs war deutlich geringer als der der Kernchromosomen (Tabelle 3). Umgekehrt waren die DC-Contigs im Vergleich zu den Kernchromosomen stark mit TE-Genen angereichert (Tabelle 3).
Obwohl die DC nicht mit Genen angereichert waren, die für sekretierte Proteine kodieren, war der Anteil an sekretierten Effektorgenen im Vergleich zu den Kernchromosomen um 30% höher. Alle oben genannten Beweise deuten darauf hin, dass A. brassicae tatsächlich ein DC beherbergen kann. DCs in Alternaria spp. wurden bisher nur von den kleinsporigen Alternaria spp. berichtet, und von keiner großsporigen Alternaria-Art ist bekannt, dass sie DCs beherbergt. Es bleibt abzuwarten, ob die DC zur Virulenz von A. brassicae beitragen. Zukünftige Studien würden die Charakterisierung des entbehrlichen Chromosoms in A. brassicae und die Korrelation seines Vorhandenseins mit der Pathogenität verschiedener Isolate beinhalten.
Orthologie-Analysen offenbaren artspezifische Gene mit mutmaßlicher Rolle in der Virulenz
Unterschiede im Gengehalt und in der Diversität innerhalb der Gene tragen zu Anpassung, Wachstum und Pathogenität bei. Um die Unterschiede im Gengehalt innerhalb der Gattung Alternaria und der Dothideomycetes zu katalogisieren, führten wir eine Orthologie-Analyse des kombinierten Satzes von 3.60.216 Proteinen aus 30 verschiedenen Arten (einschließlich 16 Alternaria-Arten) durch, die zu den Dothideomycetes gehören (Additional file 2: Table S2), von denen 3.45.321 Proteine mindestens einer der Orthogruppen zugeordnet werden konnten. Wir identifizierten 460 A. brassicae-spezifische Gene, die in A. brassicae vorhanden waren, aber in allen anderen Alternaria-Arten fehlten (Additional file 3: Tabelle S3). Zu diesen artspezifischen Genen gehörten 35 Gene, die für sekretierte Proteine kodieren, von denen 11 als Effektoren vorhergesagt wurden. Zusätzlich waren 20 dieser artspezifischen Gene auf dem DC vorhanden. Eine große Anzahl dieser Proteine gehörte zu der Kategorie der nicht charakterisierten Proteine ohne bekannte Funktion. Um zu prüfen, ob diese artspezifischen Gene das Ergebnis einer adaptiven Evolution sind, die in den wiederholungsreichen Regionen des Genoms stattfindet, führten wir einen Permutationstest durch, um die Überlappung von wiederholungsreichen Regionen und transponierbaren Elementen mit einem zufälligen Gensatz mit der Überlappung dieser artspezifischen Gene zu vergleichen. Wir fanden heraus, dass sich diese artspezifischen Gene signifikant mit wiederholungsreichen Regionen (P-Wert: 9,99e-05; Z-Score: – 4,825) und transponierbaren Elementen (P-Wert: 0,0460; Z-Score: 2,539) im Genom überlappten.
Sekundärmetabolitprofil von A. brassicae und sein Zusammenhang mit transponierbaren Elementen (TEs)
Die Gattungen Alternaria und Cochliobolus sind als Hauptproduzenten von wirtsspezifischen Sekundärmetaboliten-Toxinen bekannt. Insbesondere Alternaria spp. sind für die Produktion von chemisch vielfältigen Sekundärmetaboliten bekannt, zu denen die wirtsspezifischen Toxine (HSTs) und nicht-HSTs gehören. Diese Sekundärmetaboliten werden in der Regel durch nicht-ribosomale Peptidsynthasen (NRPS) und Polyketidsynthasen (PKS) gebildet. Wir identifizierten in A. brassicae fünf NRPS-Gencluster vom Typ SM, 12 Gencluster vom Typ PKS und sieben terpenartige Gencluster (Zusatzdatei 4: Tabelle S4). Von den fünf NRPS-Clustern konnten wir drei Cluster identifizieren, die bekannte Sekundärmetaboliten produzieren, nämlich Destruxin B, HC-Toxin und Dimethylcoprogen (Siderophor), die eine bekannte Rolle bei der Virulenz spielen.
Der Gencluster, der für die Produktion von Dimethylcoprogen (Siderophor) in A. brassicae verantwortlich ist, besteht aus 22 Genen, darunter die wichtigsten Biosynthesegene, Oxidoreduktasen und Siderophor-Transporter. Siderophore sind eisenchelatbildende Verbindungen, die von Pilzen verwendet werden, um extrazelluläres Eisen zu gewinnen, und von denen berichtet wurde, dass sie an der Virulenz von Pilzen beteiligt sind. Die Identifizierung des für die Siderophorsynthese verantwortlichen Genclusters würde die Untersuchung von Siderophoren und ihrer Rolle bei der Pathogenität von A. brassicae ermöglichen. Darüber hinaus wurde auch ein aus 12 Genen bestehendes PKS-Cluster identifiziert, das für die Melaninproduktion verantwortlich ist (Additional file 4: Tabelle S4). Der Melanin-Biosynthese-Cluster wurde bereits für A. alternata beschrieben. Auch der Transkriptionsfaktor Amr1, der die Melaninproduktion induziert, wurde bei A. brassicicola charakterisiert und ist dafür bekannt, dass er die Virulenz unterdrückt. Die Rolle von Melanin bei der Virulenz ist jedoch unklar und artspezifisch.
Die zur Gattung Alternaria gehörenden Pflanzenpathogene scheinen eine dynamische Fähigkeit zum Erwerb neuer Sekundärstoffpotenziale zu besitzen, um neue ökologische Nischen zu besiedeln. Die plausibelste Erklärung für diesen dynamischen Erwerb von Sekundärmetaboliten ist der horizontale Gentransfer innerhalb der Gattung Alternaria und möglicherweise mit anderen Gattungen. In der Literatur gibt es zahlreiche Hinweise darauf, dass ein Großteil der HST von Alternaria auf den entbehrlichen Chromosomen getragen wird und der Austausch dieser Chromosomen die Wirtsspezifität erweitern kann. Wir haben auch einen NRPS-Cluster identifiziert, der möglicherweise für HC-Toxin in einem der DCs kodiert (Gerüst 18) (Additional file 4: Tabelle S4). HC-Toxin ist eine bekannte Virulenzdeterminante des Pflanzenpathogens Cochliobolus carbonum, das Maisgenotypen infiziert, denen eine funktionelle Kopie von HM1 fehlt, einer Carbonylreduktase, die das Toxin entgiftet. In einem kürzlich erschienenen Bericht wurde gezeigt, dass A. jesenskae ebenfalls HC-Toxin produzieren kann und damit der einzige andere Pilz neben C. carbonum ist, der das Toxin produziert. Das Vorhandensein eines HC-Toxin-Genclusters, eines Virulenzfaktors bei C. carbonum, in einem DC von A. brassicae deutet darauf hin, dass der horizontale Gentransfer zwischen den Arten häufiger ist als erwartet.
Neben dem horizontalen Gentransfer können auch die rasche Duplikation, Divergenz und der Verlust der SM-Gene dazu beitragen, dass der Erreger neue Stoffwechselfähigkeiten entwickelt. Diese Duplikations- und Divergenzprozesse werden möglicherweise durch die Nähe der Sekundärmetabolit-Cluster zu den Wiederholungselementen begünstigt, die sie anfällig für RIP-Mutationen machen. Daher haben wir getestet, ob die Sekundärmetabolit-Cluster auch mit wiederholungsreichen Regionen assoziiert sind. Mit Hilfe eines Permutationstests wurde die Überlappung der wiederholungsreichen Regionen mit einem zufälligen Gensatz mit der Überlappung der Sekundärmetabolit-Cluster-Gene verglichen. Die Sekundärmetabolit-Cluster überlappten sich signifikant mit wiederholungsreichen Regionen im Vergleich zum zufälligen Gensatz (P-Wert: 0,0017; Z-Score: – 2,7963). Außerdem überlappten sich diese Cluster signifikant mit transponierbaren Elementen in den wiederholungsreichen Regionen (P-Wert: 0,0087; Z-Score: 2,9871). Dies zeigt, dass beide oben beschriebenen Mechanismen für den Erwerb eines neuen Sekundärstoffpotenzials im Fall von A. brassicae möglich sein könnten. Populationsanalysen auf Art- und Gattungsebene können Aufschluss über die Verbreitung dieser Mechanismen innerhalb der Gattung Alternaria geben.
Synteny-Analysen zeigen die genetische Grundlage der Exklusivität der Destruxin B-Produktion durch A. brassicae innerhalb der Alternaria-Gattung
Destruxin B ist eine Klasse von zyklischen Depsipeptiden, die als einer der wichtigsten Pathogenitätsfaktoren von A. brassicae bekannt ist und als wirtsspezifisches Toxin von A. brassicae beschrieben wurde. Über die Produktion von Destruxin B durch andere Alternaria-Arten wurde bisher nicht berichtet. Hier berichten wir zum ersten Mal über die biosynthetischen Gencluster, die für die Produktion von Destruxin B in A. brassicae verantwortlich sind. Der Cluster besteht aus 10 Genen, einschließlich des wichtigsten biosynthetischen Enzyms, das von einem NRPS-Gen (DtxS1) kodiert wird, und des ratenlimitierenden Enzyms DtxS3 (Aldoketo-Keto-Reduktase) (Zusatzdatei 4: Tabelle S4). Interessanterweise zeigte die Syntenie-Analyse dieses Clusters bei den sechs Alternaria-Arten, dass diese beiden Gene bei keiner der anderen Alternaria-Arten vorhanden waren, obwohl die Gesamtsyntenie des Clusters bei allen diesen Arten erhalten blieb (Abb. 5). Das Fehlen der Schlüsselgene, die für die Enzyme DtxS1 und DtxS3 im Destruxin-B-Cluster bei den anderen Alternaria-Arten kodieren, erklärt das Fehlen von Destruxin B bei diesen Arten.

Syntenie des Destruxin-B-Clusters bei den sechs Alternaria-Arten. DtxS1 und DtxS3 sind rot markiert (beschriftet mit den jeweiligen Gen-IDs) und fehlen bei allen anderen Alternaria-Arten
Destruxin B wurde außer von A. brassicae auch vom Entomopathogen Metarhizium robertsii und von Ophiosphaerella herpotricha , dem Erreger der Frühjahrsfäule des Bermudagras, beschrieben. Wang et al. beschrieben den für die Destruxinproduktion in M. robertsii verantwortlichen Sekundärmetaboliten-Biosynthesecluster. Der Destruxin-Cluster in den Metarhizhium-Arten besteht aus zwei zusätzlichen Schlüsselgenen, nämlich DtxS2 und DtxS4. DtxS2 ist für die Umwandlung von Destruxin B in Destruxin A, C, D und E verantwortlich. Wir konnten kein syntenisches Gen in dem Cluster finden, das DtxS2 oder DtxS4 in A. brassicae entspricht. Es ist bekannt, dass A. brassicae nur Destruxin B und nicht die anderen Formen von Destruxin produziert, was möglicherweise das Fehlen von DtxS2 in A. brassicae erklärt. DtxS4 ist eine Aspartat-Decarboxylase, die für die Produktion von β-Alanin aus Asparaginsäure benötigt wird und für die Herstellung der Grundstruktur von Destruxin wesentlich ist. Außerdem waren Deletionsmutanten von DtxS4 in der Lage, Destruxin zu produzieren, wenn sie mit β-Alanin im Medium ergänzt wurden. Obwohl DtxS4 nicht im Destruxin-Cluster vorhanden ist, verfügt A. brassicae über eine funktionelle Kopie der Aspartat-Decarboxylase, die an anderer Stelle im Genom vorhanden ist, und kann daher möglicherweise die Versorgung mit β-Alanin für die Synthese von Destruxin B sicherstellen. Die Evolutionsgeschichte der Destruxin-B-Produktion innerhalb der Gattung Metarhizhium ähnelt der von Alternaria. Den spezialisierten Krankheitserregern wie M. acridum fehlen die beiden Schlüsselenzyme, so dass sie keine Destruxine produzieren. M. robertsii hingegen, ein Generalist mit einem breiteren Wirtsspektrum, produziert Destruxine. Es wurde daher die Hypothese aufgestellt, dass Destruxine für die Etablierung des erweiterten Wirtsspektrums von M. roberstii verantwortlich sein könnten. Eine ähnliche Hypothese könnte auch auf A. brassicae zutreffen, das ein breites Wirtsspektrum hat und fast alle Brassicaceae infizieren kann. Weitere Experimente zur Bestimmung des Wirtsspektrums von Destruxin-Nullmutanten von A. brassicae könnten dazu beitragen, die Rolle dieses wichtigen Phytotoxins bei der Erweiterung des Wirtsspektrums von A. brassicae zu klären.
Unterschiedliche CAZym-Profile von A. brassicae und A. brassicicola innerhalb der Gattung Alternaria
CAZyme (Carbohydrate-Active enZymes) sind Proteine, die am Abbau, der Umlagerung oder der Synthese von glykosidischen Bindungen beteiligt sind. Pflanzenpathogene sezernieren eine Vielzahl von CAZymen, die die komplexen Polysaccharide in der Pflanzenzellwand abbauen. Sie bestehen aus fünf verschiedenen Klassen, nämlich Glykosidhydrolasen (GH), Glykosyltransferasen (GT), Polysaccharid-Lyasen (PL), Kohlenhydrat-Esterasen (CE) und Kohlenhydrat-bindenden Modulen (CBM). Wir identifizierten > 500 CAZyme in den sechs Alternaria spp. einschließlich A. brassicae (Zusatzdatei 5: Tabelle S5). Die CAZyme von A. brassicae und A. brassicicola unterschieden sich von denen der anderen Alternaria spp. und bildeten somit einen eigenen Cluster (Abb. 6). Die Anzahl der Enzyme mit Hilfsaktivität oder der Enzyme, die am Abbau von Pflanzenzellwänden beteiligt sind, variierte erheblich zwischen den verschiedenen verglichenen Arten. Nahezu 46% der CAZyme in A. brassicae wurden sezerniert, von denen ~ 17% als Effektoren vorhergesagt wurden.

Heatmap der CAZymprofile in 16 Arten der Gattung Alternaria. Die Clusterung der Organismen entspricht weitgehend der Phylogenie der Arten, mit Ausnahme von A. brassicae und A. brassicicola, die separat mit der Außengruppe Stemphylium lycopersici geclustert sind. Die CAZyme wurden gemäß der CAZy-Klassifikation
gruppiert. Die AA9-Familie (früher GH61; kupferabhängige lytische Polysaccharid-Monooxygenasen (LPMO)) ist im Vergleich zu den anderen CAZy-Familien in der Alternaria-Gattung deutlich vergrößert, wobei jede Art > 20 Kopien des Gens enthält. Die Kopienzahlen in Alternaria spp. sind viel höher als die für Botrytis und Fusarium spp. berichteten Kopienzahlen. Die AA9-Familie ist am Abbau von Zellwand-Polysacchariden beteiligt und wirkt bekanntermaßen auf eine Reihe von Polysacchariden wie Stärke, Xyloglucan, Cellodextrine und Glucomannan. Es wurde angenommen, dass LPMOs eine doppelte Funktion haben – sie spalten direkt die Polysaccharide der Zellwand und wirken als ROS-Generator und tragen so zum oxidativen Stress bei, der zur Nekrose in den Pflanzengeweben führt. Auffallend ist, dass 11 der 26 AA9-Proteine, die in A. brassicae vorkommen, als sekretierte Effektoren vorhergesagt werden. Die Charakterisierung dieser CAZyme und ihre Rolle in der Pathogenese könnten Gegenstand weiterer Studien sein.
Überlappende Effektorprofile innerhalb der Alternaria-Gattung
Wir haben das Effektorrepertoire von sechs Alternaria-Arten (Tabelle 2), einschließlich A. brassicae, mit Hilfe der im Abschnitt Methoden beschriebenen Pipeline vorhergesagt. Die meisten der kleinen sekretierten Proteine, die als Effektoren vorhergesagt wurden, waren nicht charakterisierte/vorgesagte Proteine und könnten daher neuartige art- oder gattungsspezifische Effektoren sein. A. brassicae wies den größten Anteil an unbekannten/vorhergesagten/hypothetischen Proteinen in der Gruppe der Effektorkandidaten auf. Wir stellten fest, dass die meisten Effektoren zwischen den sechs Arten gemeinsam sind und sich überschneiden, was darauf hindeutet, dass die weitreichenden Mechanismen der Pathogenese, die proteinhaltige Effektoren beinhalten, innerhalb der Gattung konserviert sein könnten. Allerdings fanden wir zwei Kopien eines Proteins, das eine Beta/Gamma-Kristallin-Faltung enthält, nur in A. brassicae und A. brassicicola und nicht in den anderen vier Alternaria-Arten. Eine weitere Suche in der nr-Datenbank des NCBI ergab, dass dieses Protein in der Gattung Alternaria überhaupt nicht vorkommt und die nächsten Übereinstimmungen in anderen Pflanzenpathogenen, nämlich Macrophomina- und Fusarium-Arten, gefunden wurden. Es liegen jedoch keine Informationen über seine Funktion oder Rolle bei der Pathogenität in einer der Arten vor.
Wir konnten auch feststellen, dass einige der Effektoren in A. brassicae das Potenzial haben, sich adaptiv zu entwickeln, da sie auch signifikant mit den wiederholungsreichen Regionen des Genoms assoziiert waren (P-Wert: 0,0003; Z-Score: – 2,8778). Analysen auf Populationsebene sind daher erforderlich, um die Effektoren zu identifizieren, die einer positiven Selektion unterliegen, was Aufschluss über die Evolution der Pathogenität von A. brassicae geben könnte. Die in dieser Studie identifizierten Effektoren zeigen das breite Spektrum an Proteinen, die an der Pathogenese von A. brassicae beteiligt sein könnten. 39 dieser Effektoren wurden als CAZyme vorhergesagt, die verschiedene Aufgaben beim Abbau der Zellwand und der damit verbundenen Polysaccharide haben. Das Genom von A. brassicae enthielt zwei Nekrose- und Ethylen-induzierende Peptidproteine, die mit der Pathogenese verschiedener Pathogene, einschließlich Oomyceten und Nekrotrophen, in Verbindung gebracht wurden. Kürzlich wurde gezeigt, dass Hrip1 (Hypersensitive response inducing protein 1) von A. alternata von MdNLR16 in einer klassischen Gen-für-Gen-Weise erkannt wird, und die Deletion von Hrip1 aus A. alternata steigert seine Virulenz. Ein Hrip1-Homolog ist auch in A. brassicae vorhanden, aber es wird nicht angenommen, dass es außerhalb der Zelle sezerniert wird, obwohl dies experimentell überprüft werden muss. Das Vorhandensein von Effektoren, die Gen für Gen erkannt werden, eröffnet die Möglichkeit, komplementäre R-Gene im Wirt zu identifizieren, die für die Entwicklung resistenter Sorten oder Kultivare genutzt werden können.