Características genómicas de A. brassicae y otros dos aislados de A. alternata coinfectados
Secuenciamos los genomas de dos aislados de A. alternata (PN1 y PN2) que estaban coinfectando B. juncea con A. brassicae. El ensamblaje de A. brassicae se ha descrito previamente. Brevemente, el ensamblaje consistía en nueve cromosomas completos y un cromosoma al que le faltaban repeticiones teloméricas en uno de los extremos. Además de estos cromosomas, había seis contigs, uno de los cuales tenía un tamaño de ~ 1 Mb, que podrían constituir un cromosoma prescindible (Fig. 1). El N50 del ensamblaje de A. brassicae fue de 2,98 Mb (Tabla 1). Los dos aislados que co-infectan B. juncea fueron identificados como A. alternata en base a sus secuencias ITS y GAPDH. Los ensamblajes de A. alternata Aat_PN1 y Aat_PN2 constaban de 14 contigs con un total de 33,77 Mb, y 15 contigs con un total de 33,53 Mb, respectivamente (Tabla 1). Seis contigs en cada uno de los dos conjuntos contenían repeticiones teloméricas en ambos extremos y, por tanto, es muy probable que representen moléculas cromosómicas completas. Otros cuatro contigs en ambos ensamblajes contenían repeticiones teloméricas en un extremo, pero tenían un tamaño similar al de las moléculas cromosómicas completas descritas en A. solani. Por lo tanto, los ensamblajes del genoma de los aislados de A. alternata representaban diez cromosomas casi completos de cada uno de los dos aislados.
Los alineamientos del genoma completo con Alternaria spp. relacionadas mostraron una sintenia general entre los genomas con reordenamientos menores (Fig. 2). Además, se obtuvieron secuencias mitocondriales a partir de los datos de secuenciación de los dos aislados de A. alternata. Los genomas mitocondriales de las cepas de A. alternata tenían un tamaño aproximado de 49.783 pb y 50.765 pb, respectivamente, y mostraban una gran similitud con el genoma mitocondrial de A. alternata publicado anteriormente.
La predicción de genes tras el enmascaramiento de repeticiones dio como resultado la identificación de 11593, 11495 y 11387 genes en los ensamblajes de los genomas de A. brassicae, A. alternata PN1 y PN2, respectivamente. Esto es comparable a los números de genes estimados en otras especies de Alternaria (Tabla 1). El análisis BUSCO mostró que los modelos de genes predichos en los tres genomas cubrían el 98% de los genes fúngicos conservados en una sola copia, lo que indica que los ensamblajes están casi completos. Los genes predichos fueron anotados exhaustivamente utilizando una combinación de bases de datos como se describe en la sección de Métodos (Fig. 1). Además de los tres genomas, también predijimos genes de novo en los ensamblajes del genoma de otras tres especies de Alternaria que fueron secuenciadas utilizando tecnologías de lectura larga, a saber, A. brassicicola (abra43), A. alternata (ATCC34957) y A. solani (altNL03003) (Tabla 1). Estos seis genomas y sus predicciones genéticas se utilizaron para los análisis comparativos de los grupos de genes que codifican metabolitos secundarios y los genes que codifican efectores.

Resumen del genoma de A. brassicae, (De las pistas circulares exteriores a las interiores) a pseudocromosomas/esquemas, b Genes codificadores de proteínas, c Elementos repetidos, d Elementos transponibles (ADN y LTR), e Grupos de metabolitos secundarios predichos, f Proteínas secretadas, g Efectores predichos
El análisis filogenómico asigna un clado separado para las especies A. brassicae y A. brassicicola dentro del género Alternaria
Para reconstruir con precisión la divergencia y la relación entre A. brassicae, los dos aislados de A. alternata (PN1 y PN2), y las otras especies de Alternaria, realizamos análisis filogenómicos utilizando 29 ortólogos de una sola copia que tenían la mayor señal filogenética calculada por el programa Mirlo. La selección de genes con señales filogenéticas más altas conduce a filogenias que son más congruentes con el árbol de especies . La filogenia resultante mostró que las especies de Alternaria de esporas grandes y de esporas pequeñas se agrupan por separado en dos clados diferentes (Fig. 3). Curiosamente, los dos principales patógenos de las Brassicas, es decir, A. brassicae y A. brassicicola, se agruparon por separado de todas las demás especies de Alternaria, lo que posiblemente indica una trayectoria evolutiva diferente basada en las preferencias comunes de los huéspedes de estas dos especies.
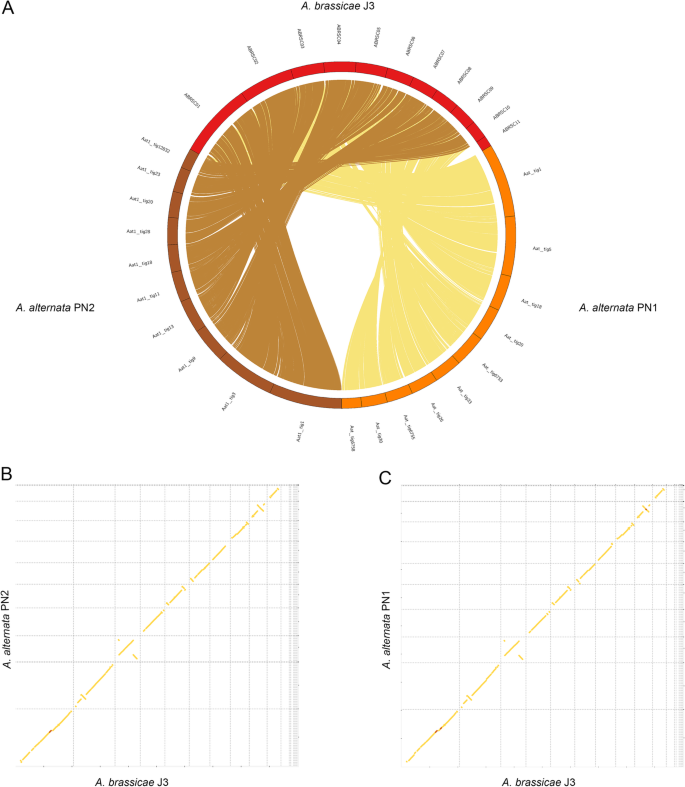
Alineaciones del genoma completo de A. alternata PN1 y PN2 con A. brassicae. a Diagrama de Circos que muestra la macrosintonía de A. alternata PN1 y PN2 con A. brassicae a través de todos los contigs excepto los contigs prescindibles (ABRSC11, scaffold13,17,18,19), b y c Syntenic dotplots de A. brassicae con A. alternata PN1 y PN2
Análisis comparativos de aislados de A. alternata obtenidos de diferentes hospedadores
Comparamos los genomas de A. alternata PN1 y PN2 (aislados de B. juncea) con el de A. alternata ATCC34957 (aislado de sorgo) para identificar cualquier diferencia en su contenido genómico que pudiera permitirles infectar a dos especies muy diferentes. Los alineamientos del genoma completo de A. alternata PN1 y PN2 con el de A. alternata ATCC34957 revelaron niveles muy altos de sintenia y la ausencia de regiones específicas de la especie. Identificamos 719, 152 y 586 genes específicos del aislamiento entre los tres aislados de A. alternata, respectivamente (Archivo adicional 1: Tabla S1). Más de dos tercios de los genes específicos de los tres aislados eran proteínas no caracterizadas o no tenían anotaciones. En particular, los tres aislados no contenían ningún cromosoma prescindible que pudiera conferir patogenicidad, como se ha informado en el caso de los aislados de A. alternata que infectan muchos de los cultivos frutales, como los cítricos, las peras y las manzanas. Los repertorios de genes de los tres aislados también consistían en un número y tipo similar de efectores, CAZymes y grupos de metabolitos secundarios (Tabla 2). Además, los dos aislados PN1 y PN2 no causan síntomas de infección por sí mismos en B. juncea en condiciones epifíticas (datos no mostrados). Nuestros resultados sugieren que estos aislados de A. alternata (PN1 y PN2) pueden ser patógenos facultativos que llevan un estilo de vida saprofito y pueden cambiar a un estilo de vida patógeno bajo ciertas condiciones ambientales.
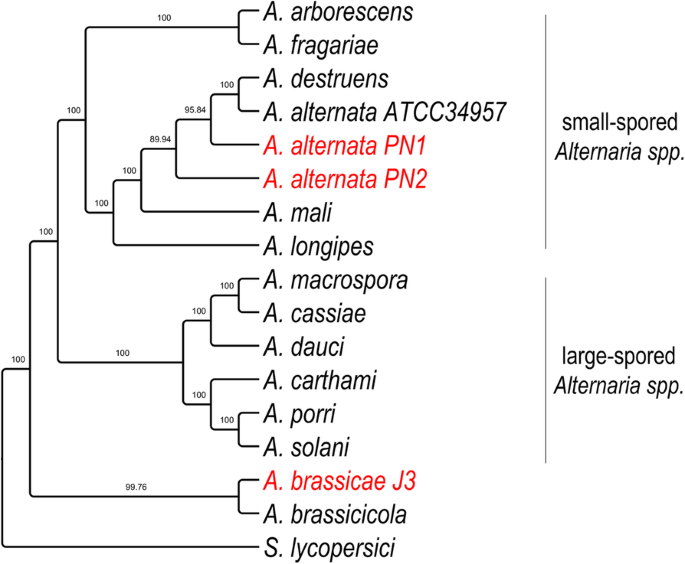
Árbol filogenético de las especies de Alternaria con S. lycopersici como grupo externo. El árbol se construyó utilizando 29 ortólogos de una sola copia, que tenían la mayor señal filogenética calculada en Mirlo. Se muestran los valores de soporte de la rama a partir de 1000 réplicas bootstrap
Una abundancia de regiones ricas en repeticiones y elementos transponibles en A. brassicae
Los patógenos de plantas filamentosas tienden a tener una arquitectura del genoma distinta con mayor contenido de repeticiones. La estimación del contenido de repeticiones y el enmascaramiento mediante RepeatModeler y RepeatMasker revelaron que el genoma de A. brassicae constaba de ~ 9,33% de repeticiones en comparación con el 2,43 y el 2,64% de repeticiones en los genomas de A. alternata. El genoma de A. brassicae alberga el mayor contenido de repeticiones (~ 9,33%) entre todas las especies de Alternaria secuenciadas hasta la fecha. Nuestro análisis demostró que el contenido de repeticiones difiere significativamente entre los aislados de A. alternata y las demás especies patógenas de Alternaria. Las especies patógenas de Alternaria, especialmente A. brassicae y A. brassicicola, tenían un repertorio considerablemente mayor de elementos LTR/Gypsy y LTR/Copia (> 8X) en comparación con los otros aislados de A. alternata (patógenos y no patógenos) (Fig. 4). Los genomas de A. brassicae y A. brassicicola también tenían una sobrerrepresentación de transposones de ADN, que ascendía a ~ 5% del genoma, en comparación con < 1% en las otras especies de Alternaria (Fig. 4).

Comparación del contenido de repeticiones en seis especies de Alternaria. El tamaño de las burbujas corresponde al a) porcentaje de elementos transponibles (TEs) en el genoma, b número de copias del TE en el genoma
Esta proliferación de ADN repetitivo y la posterior evolución de los genes que se superponen a estas regiones puede ser la clave del éxito evolutivo en el que estos patógenos han logrado persistir a lo largo de generaciones de conflicto coevolutivo con sus huéspedes. La proximidad a los TEs expone potencialmente a los genes a Mutaciones Puntuales Inducidas por Repetición (RIP) y por tanto a una evolución acelerada . La recombinación ectópica entre TEs similares también puede dar lugar a nuevas combinaciones de genes y, por lo tanto, aumentar la diversidad de proteínas o metabolitos.
La presencia de un cromosoma prescindible en el A. brassicae de esporas grandes
Cromosomas específicos de linaje (LS) o cromosomas prescindibles (DC) se han reportado en varias especies fitopatógenas incluyendo A. alternata. Se sabe que los CC de A. alternata confieren virulencia y especificidad de hospedador al aislado. Los alineamientos del genoma completo de A. brassicae con otras Alternaria spp. revelaron que un contig de aproximadamente 1 Mb junto con otros contigs más pequeños (66-366 kb) era específico de A. brassicae y no mostraba sintenia con ninguna región de las otras Alternaria spp. Sin embargo, se observó una sintenia parcial cuando el contig se alineó con las secuencias de otros cromosomas prescindibles reportados en Alternaria spp. Esto nos llevó a plantear la hipótesis de que estos contigs juntos podrían representar un CD de A. brassicae. Para confirmarlo, buscamos en los contigs la presencia de los genes AaMSAS y ALT1, que son genes marcadores conocidos de cromosomas prescindibles en Alternaria spp. Encontramos dos copias del gen AaMSAS como parte de dos grupos de biosíntesis de metabolitos secundarios en el contig de 1 Mb. Sin embargo, no encontramos ningún homólogo del gen ALT1. Además, el contenido de repeticiones de los contigs (ABRSC11, scaffold 13, 17, 18 y 19) se comparó con el genoma completo. El contenido génico de los contigs específicos del linaje fue significativamente menor que el de los cromosomas centrales (Tabla 3). Por el contrario, los contigs del CD estaban altamente enriquecidos en contenido de TE en comparación con los cromosomas centrales (Tabla 3).
Aunque, el CD no estaba enriquecido con genes que codifican proteínas secretadas, la proporción de genes efectores secretados era un 30% mayor en comparación con los cromosomas centrales. Todas las pruebas anteriores apuntan al hecho de que A. brassicae puede albergar un CD. Hasta ahora, sólo se ha informado de la existencia de DC en Alternaria spp. de esporas pequeñas, y no se conoce ninguna especie de Alternaria de esporas grandes que albergue DC. Queda por ver si el DC contribuye a la virulencia de A. brassicae. Los estudios futuros implicarían la caracterización del cromosoma prescindible en A. brassicae y la correlación de su presencia con la patogenicidad de diferentes aislados.
El análisis ortológico revela genes específicos de la especie con funciones putativas en la virulencia
Las diferencias en el contenido y la diversidad de los genes contribuyen a la adaptación, el crecimiento y la patogenicidad. Para catalogar las diferencias en el contenido génico dentro del género Alternaria y los Dothideomycetes, llevamos a cabo un análisis de ortología en el conjunto combinado de 3.60.216 proteínas de 30 especies diferentes (incluyendo 16 especies de Alternaria) pertenecientes a los Dothideomycetes (Archivo adicional 2: Tabla S2) de las cuales 3.45.321 proteínas pudieron ser asignadas a al menos uno de los ortogrupos. Identificamos 460 genes específicos de A. brassicae que estaban presentes en A. brassicae pero ausentes en todas las demás especies de Alternaria (archivo adicional 3: tabla S3). Estos genes específicos de la especie incluían 35 genes codificadores de proteínas secretadas, de los cuales 11 se predijeron como efectores. Además, 20 de estos genes específicos de la especie estaban presentes en el CD. Un gran número de estas proteínas pertenecían a la categoría de proteínas no caracterizadas sin función conocida. Para comprobar si estos genes específicos de especies son el resultado de la evolución adaptativa que tiene lugar en las regiones ricas en repeticiones del genoma, llevamos a cabo una prueba de permutación para comparar el solapamiento de las regiones ricas en repeticiones y los elementos transponibles con un conjunto de genes al azar con el solapamiento de estos genes específicos de especies. Descubrimos que estos genes específicos de la especie se solapaban significativamente con las regiones ricas en repeticiones (valor P: 9,99e-05; puntuación Z: – 4,825) y los elementos transponibles (valor P: 0,0460; puntuación Z: 2,539) en el genoma.
Perfil de metabolitos secundarios de A. brassicae y su asociación con elementos transponibles (TEs)
Se sabe que los géneros de Alternaria y Cochliobolus son los principales productores de toxinas de metabolitos secundarios específicos del huésped. Alternaria spp. es especialmente conocida por la producción de metabolitos secundarios químicamente diversos, que incluyen las toxinas específicas del hospedador (HST) y las no HST. Estos metabolitos secundarios suelen ser generados por sintasas peptídicas no ribosomales (NRPS) y sintasas policétidas (PKS). Identificamos cinco clústeres de genes SM de tipo NRPS, 12 clústeres de genes de tipo PKS y siete clústeres de genes de tipo terpeno en A. brassicae (Archivo adicional 4: Tabla S4). De los cinco clusters de NRPS, pudimos identificar tres clusters que producen metabolitos secundarios conocidos, a saber, la destruxina B, la toxina HC y el dimetilcoprogeno (sideróforo) con funciones conocidas en la virulencia.
El cluster de genes responsable de la producción de dimetilcoprogeno (sideróforo) en A. brassicae consta de 22 genes, incluyendo los principales genes biosintéticos, oxidorreductasas y transportadores de sideróforos. Los sideróforos son compuestos quelantes del hierro, utilizados por los hongos para adquirir hierro férrico extracelular y se ha informado de que están implicados en la virulencia de los hongos. La identificación del clúster de genes responsable de la síntesis de sideróforos permitiría estudiar los sideróforos y su papel en la patogenicidad de A. brassicae. Además, también se identificó un clúster de tipo PKS formado por 12 genes, responsable de la producción de melanina (archivo adicional 4: tabla S4). El cluster de biosíntesis de melanina ha sido descrito previamente para A. alternata. Además, el factor de transcripción Amr1, que induce la producción de melanina, se ha caracterizado en A. brassicicola y se sabe que suprime la virulencia . Sin embargo, el papel de la melanina en la virulencia es ambiguo y específico de cada especie.
Los patógenos de las plantas pertenecientes al género de Alternaria parecen tener una capacidad dinámica para adquirir nuevo potencial de metabolitos secundarios para colonizar nuevos nichos ecológicos. La explicación más parsimoniosa para esta adquisición dinámica de potencial de metabolitos secundarios es la transferencia horizontal de genes dentro del género Alternaria y posiblemente con otros géneros. Existe una amplia evidencia en la literatura de que gran parte de los HSTs de Alternaria son llevados en los cromosomas prescindibles y el intercambio de estos cromosomas puede ampliar la especificidad del huésped . También identificamos un cluster NRPS, que posiblemente codifica la HC-toxina en uno de los DCs (scaffold 18) (Archivo adicional 4: Tabla S4). La toxina HC es un determinante de virulencia conocido del patógeno vegetal Cochliobolus carbonum, que infecta a genotipos de maíz que carecen de una copia funcional de HM1, una carbonil reductasa que desintoxica la toxina. Un informe reciente ha demostrado que A. jesenskae también puede producir la toxina HC, lo que lo convierte en el único hongo, aparte de C. carbonum, que produce la toxina. La presencia del grupo de genes de la toxina HC, un determinante de virulencia en C. carbonum, en un DC de A. brassicae apunta al hecho de que la transferencia horizontal de genes entre especies puede ser más común de lo esperado.
Aparte de la transferencia horizontal de genes, la rápida duplicación, divergencia y pérdida de los genes SM también puede contribuir a que el patógeno desarrolle nuevas capacidades metabólicas. Estos procesos de duplicación y divergencia pueden verse favorecidos por la proximidad de los grupos de metabolitos secundarios a los elementos de repetición que los hace propensos a las mutaciones RIP. Por lo tanto, comprobamos si los grupos de metabolitos secundarios también estaban asociados a regiones ricas en repeticiones. Se utilizó una prueba de permutación para comparar el solapamiento de las regiones ricas en repeticiones con un conjunto de genes aleatorio frente al solapamiento de los genes de los grupos de metabolitos secundarios. Los grupos de metabolitos secundarios se solaparon significativamente con las regiones ricas en repeticiones en comparación con el conjunto de genes aleatorios (valor P: 0,0017; puntuación Z: – 2,7963). Además, estos grupos se solaparon significativamente con elementos transponibles entre las regiones ricas en repeticiones (valor P: 0,0087; puntuación Z: 2,9871). Esto demuestra que ambos mecanismos descritos anteriormente para la adquisición de un nuevo potencial de metabolitos secundarios pueden ser posibles en el caso de A. brassicae. Los análisis a escala de población a nivel de especie y de género pueden arrojar luz sobre la prevalencia de estos mecanismos dentro del género Alternaria.
El análisis de sintenia revela la base genética de la exclusividad de la producción de Destruxina B por A. brassicae dentro del género Alternaria
La destruxina B representa una clase de depsipéptidos cíclicos que se sabe que es uno de los factores de patogenicidad clave de A. brassicae y se ha informado de que es una toxina específica del huésped de A. brassicae . No se ha informado de que ninguna otra especie de Alternaria produzca la destruxina B. Aquí informamos por primera vez de los grupos de genes biosintéticos responsables de la producción de Destruxina B en A. brassicae. El clúster consta de 10 genes, incluyendo la principal enzima biosintética codificada por un gen NRPS (DtxS1) y la enzima limitadora de la tasa, DtxS3 (aldo-ceto reductasa) (Archivo adicional 4: Tabla S4). Curiosamente, el análisis de sintenia de este clúster entre las seis especies de Alternaria mostró que estos dos genes no estaban presentes en ninguna de las otras Alternaria spp. aunque la sintenia general del clúster se mantuvo en todas estas especies (Fig. 5). La ausencia de los genes clave que codifican las enzimas DtxS1 y DtxS3 en el cluster de la Destruxina B en las otras especies de Alternaria explica la ausencia de la Destruxina B en esas especies.

Sintenia del cluster de la Destruxina B en las seis especies de Alternaria. DtxS1 y DtxS3 están marcados en rojo (etiquetados con sus respectivos IDs de genes) y están ausentes en todas las demás especies de Alternaria
La Destruxina B se ha reportado en el entomopatógeno Metarhizium robertsii , y en Ophiosphaerella herpotricha , la causa de la mancha muerta primaveral de la bermudagrass, aparte de A. brassicae. Wang et al., describieron el grupo biosintético de metabolitos secundarios responsable de la producción de Destruxina en M. robertsii . El grupo de Destruxina en las especies de Metarhizhium consiste en dos genes clave adicionales, a saber, DtxS2 y DtxS4. DtxS2 es responsable de la conversión de la Destruxina B en Destruxina A, C, D y E. No hemos podido encontrar ningún gen sintético en el clúster que se corresponda con DtxS2 o DtxS4 en A. brassicae. Se sabe que A. brassicae sólo produce Destruxina B y no las otras formas de Destruxina, lo que posiblemente explique la ausencia de DtxS2 en A. brassicae. DtxS4 es una descarboxilasa de aspartato que se requiere para la producción de β-alanina a partir de ácido aspártico y es esencial para la producción de la estructura troncal de la Destruxina. Además, los mutantes de deleción de DtxS4 fueron capaces de producir Destruxina cuando se les suplementó con β-alanina en el medio . Aunque DtxS4 no está presente en el grupo de la Destruxina, A. brassicae tiene una copia funcional de la aspartato descarboxilasa presente en otra parte del genoma y, por tanto, posiblemente puede asegurar el suministro de β-alanina para la síntesis de la Destruxina B. La historia evolutiva de la producción de Destruxina B dentro del género Metarhizhium se asemeja mucho a la de Alternaria. Los patógenos especializados, como M. acridum, carecen de las dos enzimas clave y, por tanto, no producen Destruxinas. Sin embargo, M. robertsii, un generalista con un rango de huéspedes más amplio, produce Destruxinas. Por lo tanto, se planteó la hipótesis de que las Destruxinas podrían ser responsables del establecimiento de la amplia gama de huéspedes de M. roberstii. Una hipótesis similar puede ser también cierta en el caso de A. brassicae, que tiene una amplia gama de huéspedes y puede infectar casi todas las Brassicaceae. Otros experimentos para determinar el rango de hospedaje de los mutantes nulos de Destruxina de A. brassicae pueden ayudar a aclarar el papel de esta importante fitotoxina en la ampliación del rango de hospedaje de A. brassicae.
Perfiles CAZyme distintos de A. brassicae y A. brassicicola dentro del género Alternaria
Las CAZimas (Enzimas Activas de Carbohidratos) son proteínas que participan en la degradación, reordenación o síntesis de enlaces glicosídicos. Los patógenos de las plantas secretan una amplia gama de CAZimas que descomponen los complejos polisacáridos de la pared celular de la planta. Constan de cinco clases distintas, a saber, glucosido hidrolasas (GH), glucosiltransferasas (GT), polisacárido liasas (PL), carbohidrato esterasas (CE) y módulos de unión a carbohidratos (CBM). Identificamos > 500 CAZimas en las seis Alternaria spp. incluyendo A. brassicae (Archivo adicional 5: Tabla S5). La distribución de las CAZymas de A. brassicae y A. brassicicola variaba de las de las otras Alternaria spp. formando así un grupo separado (Fig. 6). El número de enzimas de actividad auxiliar o de enzimas implicadas en la degradación de la pared celular vegetal varió considerablemente entre las diferentes especies comparadas. Casi el 46% de las CAZimas en A. brassicae fueron secretadas, de las cuales ~ 17% se predijeron como efectores.

Mapa de calor de los perfiles de las CAZimas en 16 especies del género Alternaria. La agrupación de los organismos se asemeja mucho a la de la filogenia de las especies, excepto A. brassicae y A. brassicicola, que se agrupan por separado con el grupo externo Stemphylium lycopersici. Las CAZimas se agruparon según la clasificación CAZy
La familia AA9 (antes GH61; monooxigenasas líticas de polisacáridos dependientes del cobre (LPMO)) está significativamente ampliada en comparación con las otras familias CAZy en el género Alternaria, con cada especie conteniendo > 20 copias del gen. El número de copias en Alternaria spp. es mucho mayor que el número de copias reportado para Botrytis y Fusarium spp. La familia AA9 está implicada en la degradación de los polisacáridos de la pared celular y se sabe que actúa sobre una serie de polisacáridos que incluyen el almidón, el xiloglucano, las celodextrinas y el glucomanano. Se ha planteado la hipótesis de que los LPMOs tienen un doble papel: escinden directamente los polisacáridos de la pared celular y actúan como generadores de ROS, contribuyendo así al estrés oxidativo que conduce a la necrosis en los tejidos vegetales. Sorprendentemente, 11 de las 26 proteínas AA9 presentes en A. brassicae son efectores secretados. La caracterización de estas CAZymes y su papel en la patogénesis podría ser objeto de nuevos estudios.
Perfiles de efectores superpuestos dentro del género Alternaria
Predecimos el repertorio de efectores de seis especies de Alternaria (Tabla 2), incluida A. brassicae, utilizando la línea de producción descrita en la sección de métodos. La mayoría de las pequeñas proteínas secretadas que se predijeron como efectores eran proteínas no caracterizadas/predecidas y, por tanto, pueden ser nuevos efectores específicos de la especie o del género. A. brassicae tenía la mayor proporción de proteínas desconocidas/predichas/hipotéticas en el conjunto de efectores candidatos. Encontramos que la mayoría de los efectores entre las seis especies eran comunes y se solapaban, lo que sugiere que los amplios mecanismos de patogénesis que implican efectores proteicos pueden conservarse dentro del género. Sin embargo, encontramos dos copias de una proteína que contiene un pliegue beta/gamma-cristalino que sólo está presente en A. brassicae y A. brassicicola y no en las otras cuatro especies de Alternaria. Una búsqueda posterior en la base de datos nr del NCBI reveló que esta proteína estaba completamente ausente en el género Alternaria y que las coincidencias más cercanas se encontraban en otros patógenos de plantas, a saber, las especies Macrophomina y Fusarium. Sin embargo, no se dispone de información sobre su función o papel en la patogenicidad en ninguna de las especies.
También pudimos establecer que algunos de los efectores en A. brassicae tienen el potencial de evolucionar adaptativamente, ya que también se asociaron significativamente con las regiones ricas en repeticiones del genoma (valor P: 0,0003; puntuación Z: – 2,8778). Por lo tanto, se requieren análisis a nivel de población para identificar los efectores bajo selección positiva, lo que podría arrojar luz sobre la evolución de la patogenicidad en A. brassicae. Los efectores identificados en este estudio revelan la amplia gama de proteínas que pueden estar implicadas en la patogénesis de A. brassicae. Se predijo que 39 de estos efectores eran CAZimas que desempeñaban diversas funciones en la degradación de la pared celular y los polisacáridos asociados. El genoma de A. brassicae contenía dos proteínas péptidas inductoras de necrosis y etileno, que han sido implicadas en la patogénesis de varios patógenos, incluyendo oomicetos y necrótrofos. Recientemente se ha demostrado que la proteína Hrip1 (Hypersensitive response inducing protein 1) de A. alternata es reconocida por MdNLR16 de una manera clásica de gen por gen, y la supresión de Hrip1 de A. alternata aumenta su virulencia . Un homólogo de Hrip1 también está presente en A. brassicae, pero no se predice que sea secretado fuera de la célula, aunque esto necesita ser verificado experimentalmente. La presencia de efectores que se reconocen de forma genética abre la posibilidad de identificar genes R complementarios en el hospedador que pueden utilizarse para desarrollar variedades o cultivares resistentes.