Genomic features of A. brassicae:n ja kahden muun A. alternata -isolaatin genomin erityispiirteet
Sekvensoimme kahden A. alternata -isolaatin (PN1 ja PN2) genomit, jotka tartuttivat B. juncea -lajia yhdessä A. brassicae:n kanssa. A. brassicae -yhdistelmä on kuvattu aiemmin . Kokoonpano koostui lyhyesti sanottuna yhdeksästä täydellisestä kromosomista ja yhdestä kromosomista, jonka toisesta päästä puuttui telomeerisia toistoja. Näiden kromosomien lisäksi oli kuusi kontigia, joista yksi oli kooltaan ~ 1 Mb, jotka yhdessä saattavat muodostaa tarpeettoman kromosomin (kuva 1). A. brassicae -kokoonpanon N50 oli 2,98 Mb (taulukko 1). Kaksi B. juncea -kasvia yhdessä infektoivaa isolaattia tunnistettiin A. alternataksi niiden ITS- ja GAPDH-sekvenssien perusteella. A. alternata -kokoonpanot Aat_PN1 ja Aat_PN2 koostuivat 14 kontigista, joiden yhteispituus oli 33,77 Mb, ja 15 kontigista, joiden yhteispituus oli 33,53 Mb (taulukko 1). Kummassakin kokoonpanossa kuusi kontigia sisälsi telomeerisia toistoja molemmissa päissä, joten ne edustavat todennäköisesti kokonaisia kromosomimolekyylejä. Neljä muuta kontigia molemmissa kokoonpanoissa sisälsi telomeerisia toistoja toisessa päässä, mutta ne olivat samankokoisia kuin täydet kromosomimolekyylit, kuten A. solani -bakteerissa on kuvattu. Näin ollen A. alternata -isolaattien genomikokoonpanot edustivat kummankin isolaatin kymmentä lähes täydellistä kromosomia.
Kokonaisen genomin kohdistukset sukulaislajin Alternaria spp. kanssa osoittivat, että genomit ovat keskenään syntenisiä ja että niiden välillä on vain vähäisiä uudelleenjärjestäytymisiä (kuva 2). Lisäksi kahden A. alternata -isolaatin sekvensointitiedoista saatiin myös mitokondriosekvenssit. A. alternata -kantojen mitokondriogenomit olivat noin 49 783 bp:n ja 50 765 bp:n kokoisia, ja ne olivat hyvin samankaltaisia aiemmin julkaistun A. alternata -lajin mitokondriogenomin kanssa .
Geenien ennustaminen toistojen peittämisen jälkeen johti 11593 geenin, 11495 geenin ja 11387 geenin identifiointiin A. brassicae -lajin, A. alternata -lajin PN1-lajin ja A. alternata -lajin PN2-lajin genomikokoelmissa. Tämä oli verrattavissa muissa Alternaria spp. lajeissa arvioituihin geenimääriin (taulukko 1). BUSCO-analyysi osoitti, että kolmessa genomissa ennustetut geenimallit kattoivat 98 prosenttia yhden kopion konservoiduista sienigeeneistä, mikä osoittaa, että kokoelmat ovat lähes täydellisiä. Ennustetut geenit annotoitiin kattavasti käyttämällä tietokantojen yhdistelmää, kuten menetelmiä käsittelevässä osassa on kuvattu (kuva 1). Näiden kolmen genomin lisäksi ennustimme geenejä de novo myös kolmen muun Alternaria-lajin genomikokoonpanoista, jotka sekvensoitiin käyttäen long-read-tekniikkaa: A. brassicicola (abra43), A. alternata (ATCC34957) ja A. solani (altNL03003) (taulukko 1). Näitä kuutta genomia ja niiden geeniennusteita käytettiin sekundaarisia metaboliitteja koodaavien geeniryhmien ja efektoreita koodaavien geenien vertaileviin analyyseihin.

Kuva 1

Yhteenvetona aineistosta, joka on muodostettu vuonna 1998, on käytetty seuraavia geenejä: 1. brassicae-genomista, (Ulommasta sisempään ympyränmuotoiset kappaleet) a pseudokromosomit/caffoldit, b proteiineja koodaavat geenit, c toistuvat elementit, d siirtyvät elementit (DNA ja LTR), e ennustetut sekundaarimetaboliittien klusterit, f erittyvät proteiinit, g ennustetut efektorit
Fylogenomianalyysi osoittaa erillisen kladin brassica-taudinaiheuttajille A. brassicae ja A. brassicicola Alternaria-suvun sisällä
Voidaksemme rekonstruoida tarkasti A. brassicae:n, kahden A. alternata -isolaatin (PN1 ja PN2) ja muiden Alternaria-lajien väliset eroavaisuudet ja sukulaisuussuhteet suoritimme fylogenomianalyysejä käyttäen 29:ää yksittäiskopioituihin ortologeihin perustuvaa ortologia, joilla oli korkein fylogeneettinen signaali Mirlo-ohjelman laskemana. Korkeamman fylogeneettisen signaalin omaavien geenien valinta johtaa fylogenioihin, jotka ovat paremmin yhteneväisiä lajipuun kanssa . Tuloksena saatu fylogenia osoitti, että suurikokoiset Alternaria- ja pienikokoiset Alternaria-lajit ryhmittyivät erikseen kahteen eri kladiin (kuva 3). Mielenkiintoista oli, että kaksi tärkeintä Brassicas-lajin patogeeniä eli A. brassicae ja A. brassicicola ryhmittyivät erillään kaikista muista Alternaria-lajeista, mikä mahdollisesti osoittaa erilaista kehityskulkua näiden kahden lajin yhteisten isäntäpreferenssien perusteella.
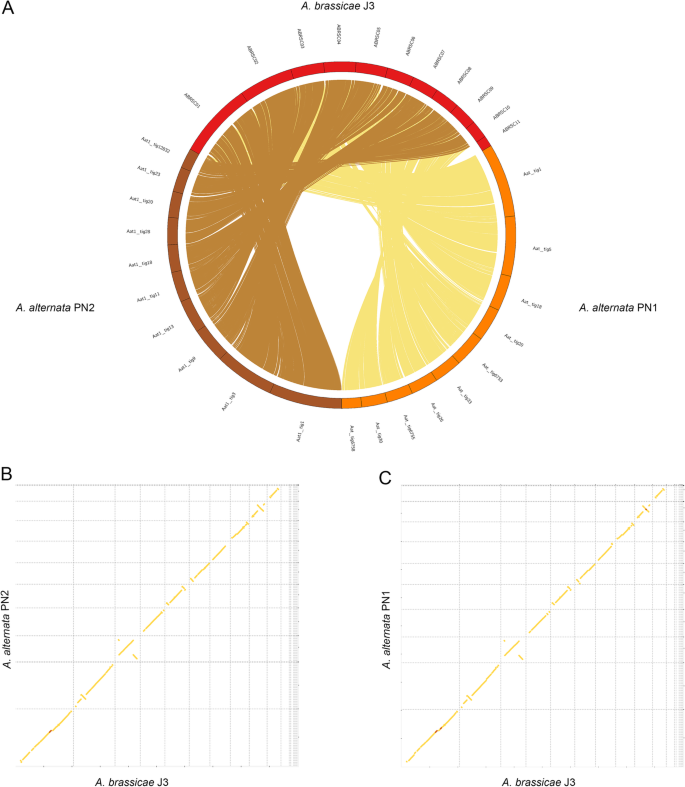
A. alternata PN1:n ja PN2:n ja A. brassicae:n koko genomin kohdistukset. a Circos-piirros, joka osoittaa A. alternata PN1:n ja PN2:n makrosynteesin A. alternata PN1:n ja PN2:n ja A. brassicae kaikkien contigien osalta lukuun ottamatta tarpeettomia contigeja (ABRSC11, scaffold13,17,18,19), b ja c A. brassicae:n ja A. alternata PN1:n ja PN2:n synteettiset pistekuvioinnit
Vertailevia analyysejä A. alternata PN1:n ja PN2:n välillä. alternata-isolaatteja, jotka on saatu eri isännistä
Vertailimme B. junceasta eristettyjen A. alternata PN1:n ja PN2:n genomeja A. alternata ATCC34957:n (eristetty durrasta) genomiin selvittääksemme, onko niiden genomisisällössä eroja, joiden ansiosta nämä voivat infektoida kahta hyvin erilaista lajia. A. alternata PN1:n ja PN2:n koko genomin kohdistaminen A. alternata ATCC34957:n genomiin osoitti, että syntenian taso on erittäin korkea ja että lajikohtaisia alueita ei ole. Näiden kolmen A. alternata -isolaatin välillä tunnistettiin 719, 152 ja 586 isolaattispesifistä geeniä (lisätiedosto 1: taulukko S1). Yli kaksi kolmasosaa kaikkien kolmen isolaatin isolaattispesifisistä geeneistä oli luonnehtimattomia proteiineja tai niillä ei ollut merkintöjä. Huomionarvoista on, että kaikki kolme isolaattia eivät sisältäneet tarpeettomia kromosomeja, jotka voivat antaa patogeenisuutta, kuten on raportoitu A. alternata -isolaateista, jotka infektoivat monia hedelmäkasveja, kuten sitrushedelmiä, päärynöitä ja omenoita. Kolmen isolaatin geenirepertuaari koostui myös samankaltaisesta määrästä ja tyypistä efektoreita, CAZymejä ja sekundaarimetaboliittien klustereita (taulukko 2). Lisäksi kaksi isolaattia PN1 ja PN2 eivät yksinään aiheuta infektio-oireita B. junceassa epifytoottisissa olosuhteissa (tietoja ei ole esitetty). Tuloksemme viittaavat siihen, että nämä A. alternata -isolaatit (PN1 ja PN2) voivat olla fakultatiivisia patogeeneja, jotka elävät saprofyyttistä elämäntapaa ja voivat siirtyä patogeeniseen elämäntapaan tietyissä ympäristöolosuhteissa.
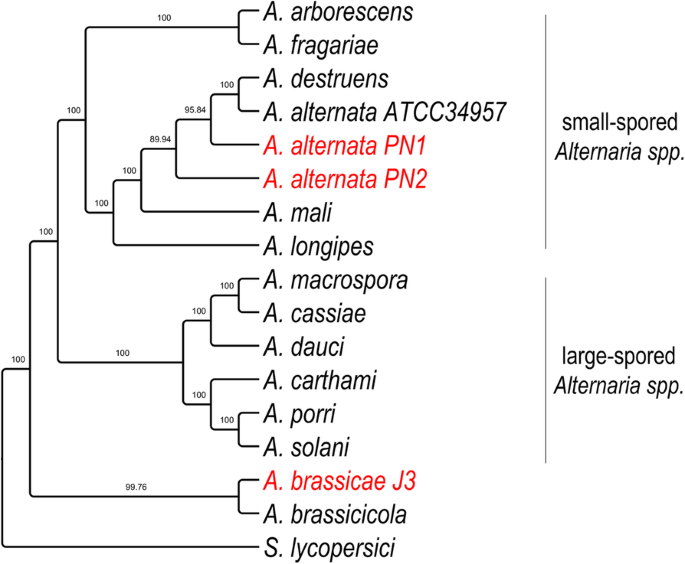
Alternaria-lajien fylogeneettistä puuta, jossa on S. lykopersici outgroup. Puu muodostettiin käyttäen 29 yhden kopion ortologia, joilla oli korkein fylogeneettinen signaali Mirlo-ohjelmalla laskettuna. Oksien tukiarvot 1000 bootstrap-toiston perusteella on esitetty
Runsas määrä toistorikkaita alueita ja transposable elementtejä A. brassicae
Filamenttikasvien patogeeneillä on yleensä erilainen genomin arkkitehtuuri, jossa toistojen määrä on suurempi. Toistopitoisuuden arviointi ja peittäminen RepeatModeler- ja RepeatMasker-ohjelmilla osoitti, että A. brassicaen genomi koostui ~ 9,33 prosentista toistoja verrattuna A. alternata -genomien 2,43 ja 2,64 prosenttiin toistoista. A. brassicae -lajin genomissa on suurin toistuvuuspitoisuus (~ 9,33 %) kaikista tähän mennessä sekvensoiduista Alternaria-lajeista. Analyysimme osoitti, että toistopitoisuus eroaa merkittävästi A. alternata -isolaattien ja muiden patogeenisten Alternaria-lajien välillä. Patogeenisillä Alternaria-lajeilla, erityisesti A. brassicae:lla ja A. brassicicola:lla, oli huomattavasti enemmän LTR/Gypsy- ja LTR/Copia-elementtejä (> 8X) verrattuna muihin A. alternata -isolaatteihin (patogeenisiin ja ei-patogeenisiin) (kuva 4). A. brassicae- ja A. brassicicola -lajien genomeissa oli myös DNA-transposonien yliedustus, joka oli ~ 5 % genomista, verrattuna < 1 %:iin muissa Alternaria-lajeissa (kuva 4).

Vertailu toistoelementtipitoisuudesta kuudessa Alternaria-lajeissa. Kuplien koko vastaa a) transponoituvien elementtien (TE) prosenttiosuutta genomissa, b TE:n kopiolukua genomissa
Tämä toistuvan DNA:n lisääntyminen ja sen jälkeen näiden alueiden päällekkäisten geenien evoluutio voi olla avain evolutiiviseen menestykseen, jossa nämä patogeenit ovat onnistuneet säilymään sukupolvien ajan isäntäkasvustojensa kanssa käydyissä yhteistoiminnallisissa konflikteissa. TE:iden läheisyys saattaa altistaa geenit toistojen aiheuttamille pistemutaatioille (RIP) ja siten nopeuttaa evoluutiota. Ektooppinen rekombinaatio samankaltaisten TE:iden välillä voi myös johtaa uusiin geeniyhdistelmiin ja siten lisätä proteiinien tai aineenvaihduntatuotteiden monimuotoisuutta.
Tarvittavan kromosomin esiintyminen isohaaraisessa A. brassicae -lajissa
Linjaspesifisiä (LS) kromosomeja tai tarvitsematonta kromosomia (DC, dispensable chromosomes) on raportoitu useista kasvipatogeenisistä lajeista, mukaan luettuna A. alternata. A. alternatan DC-kromosomien tiedetään antavan isolaatille virulenssin ja isäntäspesifisyyden. A. brassicae -lajin ja muiden Alternaria spp. -lajien koko genomin kohdistukset paljastivat, että noin 1 Mb:n kokoinen kontig ja muut pienemmät kontigit (66-366 kb) olivat spesifisiä A. brassicae -lajille eikä niillä ollut synteniaa minkään muiden Alternaria spp. -lajien alueen kanssa. Tämä sai meidät olettamaan, että nämä kontigit voivat yhdessä edustaa A. brassicae -lajin DC:tä. Vahvistaaksemme tämän etsimme kontigeista AaMSAS- ja ALT1-geenejä, jotka ovat tunnettuja Alternaria spp:n dispensoituvien kromosomien merkkigeenejä. Löysimme kaksi kopiota AaMSAS-geenistä osana kahta sekundaarimetaboliittien biosynteettistä klusteria 1 Mb:n kontigissa. Emme kuitenkaan löytäneet yhtään ALT1-geenin homologia. Lisäksi kontigien (ABRSC11, scaffold 13, 17, 18 ja 19) toistopitoisuutta verrattiin koko genomiin. Linjakohtaisten kontigien geenipitoisuus oli huomattavasti pienempi kuin ydinkromosomien (taulukko 3). Sitä vastoin DC-kontigit olivat TE-pitoisuudeltaan erittäin rikastuneita ydinkromosomeihin verrattuna (taulukko 3).
Vaikka DC ei ollutkaan rikastunut erittyviä proteiineja koodaavilla geeneillä, erittyvien efektoijien geenien osuus oli 30 % korkeampi ydinkromosomeihin verrattuna. Kaikki edellä mainitut todisteet viittaavat siihen, että A. brassicae saattaa todellakin sisältää DC:n. Alternaria-suvun DC:tä on tähän mennessä raportoitu vain pienikokoisten Alternaria-suvun lajeista, eikä suurikokoisten Alternaria-lajien tiedetä sisältävän DC:tä. Jää nähtäväksi, vaikuttaako DC A. brassicaen virulenssiin. Tulevissa tutkimuksissa olisi tarkoitus karakterisoida A. brassicaen dispensable-kromosomi ja korreloida sen esiintyminen eri isolaattien patogeenisyyteen.
Ortologia-analyysi paljastaa lajispesifisiä geenejä, joilla on oletettuja rooleja virulenssissa
Eroavaisuudet geenien pitoisuuksissa ja monimuotoisuudessa geenien sisällä vaikuttavat sopeutumiseen, kasvuun ja patogeenisyyteen. Luetteloidaksemme Alternaria-suvun ja Dothideomycetes-suvun geenisisällön erot suoritimme ortologia-analyysin yhdistetylle 3 60 216 proteiinin joukolle, joka oli peräisin 30 eri lajista (mukaan lukien 16 Alternaria-lajia), jotka kuuluvat Dothideomycetes-suvun lajeihin (Lisätiedosto 2: Taulukko S2), joista 3 45 321 proteiinia voitiin luokitella vähintään yhteen ortoryhmään. Tunnistimme 460 A. brassicae -spesifistä geeniä, jotka esiintyivät A. brassicae -lajissa mutta puuttuivat kaikista muista Alternaria-lajeista (lisätiedosto 3: taulukko S3). Näihin lajispesifisiin geeneihin kuului 35 erittyviä proteiineja koodaavaa geeniä, joista 11:n ennustettiin olevan efektoreita. Lisäksi 20 näistä lajispesifisistä geeneistä esiintyi DC:ssä. Suuri osa näistä proteiineista kuului luonnehtimattomien proteiinien luokkaan, joiden toimintaa ei tunneta. Testataksemme, ovatko nämä lajispesifiset geenit seurausta genomin toistorikkailla alueilla tapahtuvasta adaptiivisesta evoluutiosta, suoritimme permutaatiotestin, jolla vertasimme toistorikkaiden alueiden ja transposable elementtien päällekkäisyyttä satunnaisen geenijoukon kanssa näiden lajispesifisten geenien päällekkäisyyteen. Havaitsimme, että nämä lajispesifiset geenit olivat merkittävästi päällekkäisiä genomin toistorikkaiden alueiden (P-arvo: 9,99e-05; Z-tulos: – 4,825) ja transponoituvien elementtien (P-arvo: 0,0460; Z-tulos: 2,539) kanssa.
A. brassicaen sekundaarimetaboliittiprofiili ja sen yhteys transponoituviin elementteihin (TE:t)
Alternaria- ja Cochliobolus-suvut ovat tunnetusti merkittäviä isäntäkohtaisten sekundaarimetaboliitti-toksiinien tuottajia. Erityisesti Alternaria spp. tunnetaan kemiallisesti monipuolisten sekundaaristen aineenvaihduntatuotteiden tuotannosta, johon kuuluu isäntäkohtaisia toksiineja (HST) ja muita kuin HST-toksiineja. Näitä sekundaarisia aineenvaihduntatuotteita tuottavat yleensä ei-ribosomaaliset peptidisyntaasit (NRPS) ja polyketidisyntaasit (PKS). Tunnistimme viisi NRPS-tyyppistä SM-geeniryhmää, 12 PKS-tyyppistä geeniryhmää ja seitsemän terpeenin kaltaista geeniryhmää A. brassicae -kasvintuhoojasta (lisätiedosto 4: taulukko S4). Viidestä NRPS-klusterista pystyimme tunnistamaan kolme klusteria, jotka tuottavat tunnettuja sekundaarisia aineenvaihduntatuotteita, nimittäin Destruxin B:tä, HC-toksiinia ja dimetyylikoprogeenia (sideroforia), joilla on tunnetusti merkitystä virulenssissa.
Dimetyylikoprogeenin (sideroforin) tuotannosta vastuussa oleva geeniklusteri A. brassicae -bakteerissa koostuu 22 geenistä, mukaan lukien tärkeimpiä biosynteettisiä geenejä, hapettumis- eli hapettumis- tai hapettumisreduktaaseja ja sideroforin kuljettimia. Sideroforit ovat rautaa kelatoivia yhdisteitä, joita sienet käyttävät solunulkoisen rautaraudan hankkimiseen, ja niiden on raportoitu osallistuvan sienten virulenssiin. Siderofoorisynteesistä vastaavan geeniryhmän tunnistaminen mahdollistaisi siderofoorien ja niiden roolin tutkimisen A. brassicaen patogeenisuudessa. Lisäksi tunnistettiin 12 geenistä koostuva PKS-tyyppinen klusteri, joka vastaa melaniinin tuotannosta (lisätiedosto 4: taulukko S4). Melaniinin biosynteesiklusteri on kuvattu A. alternatan osalta aiemmin. Myös transkriptiotekijä Amr1, joka indusoi melaniinituotantoa, on luonnehdittu A. brassicicolalla, ja sen tiedetään tukahduttavan virulenssia . Melaniinin rooli virulenssissa on kuitenkin epäselvä ja lajikohtainen .
Alternaria-sukuun kuuluvilla kasvipatogeeneilla näyttää olevan dynaaminen kyky hankkia uusia sekundaarisia aineenvaihduntatuotteita kolonisoidakseen uusia ekologisia markkinarakoja. Yksinkertaisin selitys tälle sekundaarimetaboliittipotentiaalin dynaamiselle hankinnalle on horisontaalinen geeninsiirto Alternaria-suvun sisällä ja mahdollisesti muiden sukujen kanssa. Kirjallisuudessa on laajalti todisteita siitä, että suuri osa Alternarian HST-geeneistä kulkee tarpeettomilla kromosomeilla, ja näiden kromosomien vaihto voi laajentaa isäntäspesifisyyttä. Tunnistimme myös NRPS-klusterin, joka mahdollisesti koodaa HC-toksiinia yhdessä DC:ssä (scaffold 18) (lisätiedosto 4: taulukko S4). HC-toksiini on tunnettu virulenssideterminantti kasvipatogeeni Cochliobolus carbonumille, joka infektoi maissin genotyyppejä, joista puuttuu toimiva kopio HM1:stä, joka on karbonyylireduktaasi, joka myrkyttää toksiinin . Äskettäinen raportti osoitti, että myös A. jesenskae voi tuottaa HC-toksiinia, mikä tekee siitä ainoan muun sienen kuin C. carbonumin, joka tuottaa tätä toksiinia . HC-toksiinin geeniklusterin, joka on C. carbonumin virulenssimäärittäjä, esiintyminen A. brassicae -sienen DC:ssä viittaa siihen, että lajien välinen horisontaalinen geeninsiirto voi olla odotettua yleisempää.
Horisontiaalisen geeninsiirron lisäksi SM-geenien nopea monistuminen, erilaistuminen ja häviäminen voivat myös osaltaan vaikuttaa siihen, että taudinaiheuttaja kehittää uusia aineenvaihdunnallisia kykyjä. Näitä monistumis- ja erilaistumisprosesseja saattaa hyvinkin edesauttaa se, että sekundaarimetaboliittiklusterit ovat lähellä toistuvia elementtejä, mikä tekee niistä alttiita RIP-mutaatioille. Sen vuoksi testasimme, liittyvätkö sekundaarimetaboliittiklusterit myös toistorikkaisiin alueisiin. Permutaatiotestillä verrattiin toistorikkaiden alueiden päällekkäisyyttä satunnaisen geenijoukon kanssa sekundaarimetaboliittiklusterien geenien päällekkäisyyteen. Sekundaariset metaboliittiklusterit olivat merkittävästi päällekkäisiä toistorikkaiden alueiden kanssa verrattuna satunnaiseen geenijoukkoon (P-arvo: 0,0017; Z-score: – 2,7963). Lisäksi nämä klusterit limittyivät merkittävästi transponoituvien elementtien kanssa toistorikkailla alueilla (P-arvo: 0,0087; Z-score: 2,9871). Tämä osoittaa, että molemmat edellä kuvatut mekanismit uuden sekundaarimetaboliittipotentiaalin hankkimiseksi voivat olla mahdollisia A. brassicaen tapauksessa. Populaatiotason analyysit laji- ja sukutasolla voivat valottaa näiden mekanismien yleisyyttä Alternaria-suvun sisällä.
Syntenia-analyysi paljastaa geneettisen perustan Destruxin B:n tuotannon yksinoikeudelle A. brassicae-suvun sisällä Alternaria-suvussa
Destruksiini B edustaa syklisten depsipeptidien luokkaa, jonka tiedetään olevan yksi A. brassicaen keskeisistä patogeenisuustekijöistä, ja sen on raportoitu olevan A. brassicaen isäntäspesifinen toksiini . Minkään muun Alternaria-lajin ei ole raportoitu tuottavan Destruxin B:tä. Tässä raportoidaan ensimmäistä kertaa A. brassicae -lajin Destruxin B -tuotannosta vastaavat biosynteettiset geeniryhmät. Klusteri koostuu 10 geenistä, mukaan lukien NRPS-geenin (DtxS1) koodaama tärkein biosynteettinen entsyymi ja nopeutta rajoittava entsyymi DtxS3 (aldo-keto-reduktaasi) (Additional file 4: Table S4). Mielenkiintoista on, että tämän klusterin syntenia-analyysi kuuden Alternaria-lajin välillä osoitti, että näitä molempia geenejä ei esiintynyt missään muussa Alternaria spp:ssä, vaikka klusterin yleinen syntenia säilyi kaikissa näissä lajeissa (kuva 5). DtxS1- ja DtxS3-entsyymejä koodaavien avaingeenien puuttuminen Destruxin B -klusterista muissa Alternaria-lajeissa selittää Destruxin B:n puuttumisen näistä lajeista.

Destruxin B -klusterin syntenia kuudessa Alternaria-lajissa. DtxS1 ja DtxS3 on merkitty punaisella (merkitty vastaavilla geenitunnuksilla), ja ne puuttuvat kaikista muista Alternaria-lajeista
Destruksiini B:tä on raportoitu A. brassicae:n lisäksi entomopatogeenistä Metarhizium robertsii , ja bermudaruohon kevätkuolleen laikun aiheuttajasta Ophiosphaerella herpotricha . Wang et al. kuvasivat sekundaarimetaboliittien biosynteettisen klusterin, joka vastaa M. robertsii -bakteerin Destruxin-tuotannosta. Metarhizhium-lajin Destruxin-klusteri koostuu kahdesta muusta avaingeenistä eli DtxS2:sta ja DtxS4:stä. DtxS2 vastaa Destruxin B:n muuntamisesta Destruxin A:ksi, C:ksi, D:ksi ja E:ksi. Emme löytäneet klusterista yhtään syntenistä geeniä, joka vastaisi A. brassicaen DtxS2:ta tai DtxS4:ää. A. brassicaen tiedetään tuottavan vain Destruxin B:tä eikä muita Destruxin-muotoja, mikä saattaa mahdollisesti selittää DtxS2:n puuttumisen A. brassicaesta. DtxS4 on aspartaattidekarboksylaasi, jota tarvitaan β-alaniinin tuottamiseen asparagiinihaposta ja joka on välttämätön Destruxinin runkorakenteen tuottamiseksi. Lisäksi DtxS4:n deletoituneet mutantit pystyivät tuottamaan Destruxinia, kun niiden elatusaineeseen lisättiin β-alaniinia. Vaikka DtxS4:ää ei esiinny Destruxin-klusterissa, A. brassicae:lla on yksi toimiva kopio muualla genomissa esiintyvästä aspartaattidekarboksylaasista, ja näin ollen se voi mahdollisesti varmistaa β-alaniinin saannin Destruxin B:n synteesiä varten. Destruxin B:n tuotannon evoluutiohistoria Metarhizhium-suvussa muistutti läheisesti Alternarian tuotantoa. Erikoistuneilta patogeeneiltä, kuten M. acridumilta, puuttuu kaksi avainentsyymiä, eivätkä ne näin ollen tuota destruksiineja. M. robertsii, joka on generalisti ja jolla on laajempi isäntäalue, tuottaa kuitenkin Destruxineja. Siksi oletettiin, että Destruxiinit saattavat olla vastuussa M. roberstii -bakteerin laajemman isäntäalueen vakiintumisesta. Samanlainen hypoteesi voi pitää paikkansa myös A. brassicae -kasvintuhoojan kohdalla, jolla on laaja isäntäalue ja joka voi tartuttaa lähes kaikki Brassicaceae-kasvit. Lisäkokeet A. brassicaen Destruxin null -mutanttien isäntäalueen määrittämiseksi voivat auttaa selvittämään tämän tärkeän kasvimyrkyn roolia A. brassicaen isäntäalueen laajentamisessa.
A. brassicaen ja A. brassicicolan erilaiset CAZyymiprofiilit Alternaria-suvun sisällä
CAZyymit (Carbohydrate-Active enZymes, hiilihydraatti-aktiiviset enZyymit) ovat valkuaisaineiden proteiineja, jotka ovat osallisina glukosidisten sidoksien hajottamisessa, uudelleenjärjestelyssä tai synteesissä. Kasvipatogeenit erittävät monenlaisia CAZymejä, jotka hajottavat kasvin soluseinän monimutkaisia polysakkarideja. Ne koostuvat viidestä eri luokasta eli glykosidihydrolaaseista (GH), glykosyylitransferaaseista (GT), polysakkaridilyaaseista (PL), hiilihydraattiesteraaseista (CE) ja hiilihydraatteja sitovista moduuleista (CBM). Tunnistimme > 500 CAZyymiä kuudesta Alternaria spp:stä, mukaan lukien A. brassicae (lisätiedosto 5: taulukko S5). A. brassicaen ja A. brassicicolan CAZyymijakauma poikkesi muiden Alternaria spp:ien CAZyymijakaumasta, joten ne muodostivat erillisen klusterin (kuva 6). Apuaktiivisten entsyymien tai kasvin soluseinän hajotukseen osallistuvien entsyymien määrä vaihteli huomattavasti eri vertailtujen lajien välillä. Lähes 46 % A. brassicae -lajin CAZymeistä oli erittyviä, joista ~ 17 %:n ennustettiin olevan efektoivia aineita.
Kuvio 6
Kuvio 6
Lämpökartta 16:n lajin CAZymeiden profiileista Alternariat- suvun kanssa. Eliöiden klusteroituminen muistuttaa läheisesti lajifylogenian klusteroitumista lukuun ottamatta A. brassicae:tä ja A. brassicicola:ta, jotka klusteroituvat erikseen ulkoryhmän Stemphylium lycopersici kanssa. CAZy-geenit ryhmiteltiin CAZy-luokituksen
mukaan AA9-perhe (entinen GH61; kupari-riippuvaiset lyytiset polysakkaridimonooksygenaasit (LPMO)) on muihin CAZy-perheisiin verrattuna huomattavasti laajentunut Alternaria-suvussa, sillä kukin laji sisältää > 20 kopiota geeniä. Alternaria spp. -suvun kopiomäärät ovat paljon suuremmat kuin Botrytis ja Fusarium spp. -suvuista ilmoitetut kopiomäärät. AA9-perhe osallistuu soluseinän polysakkaridien hajottamiseen, ja sen tiedetään vaikuttavan useisiin polysakkarideihin, kuten tärkkelykseen, ksyloglukaaniin, sellodekstriineihin ja glukomannaaniin. LPMO:illa on oletettu olevan kaksoisrooli – ne pilkkovat suoraan soluseinän polysakkarideja ja toimivat ROS:n tuottajina ja siten edistävät oksidatiivista stressiä, joka johtaa kasvikudosten nekroosiin. Silmiinpistävää on, että A. brassicae -kasvintuhoojassa esiintyvistä 26:sta AA9-proteiinista 11:n ennustetaan olevan erittyviä vaikuttajia. Näiden CAZyymien karakterisointi ja niiden rooli patogeneesissä voisivat olla jatkotutkimusten kohteena.
Yllekkäiset efektoriprofiilit Alternaria-suvun sisällä
Ennustimme kuuden Alternaria-lajin (taulukko 2), mukaan lukien A. brassicae, efektorirepertuaarin käyttämällä menetelmiä käsittelevässä osassa kuvattua putkea. Suurin osa efektoreiksi ennustetuista pienistä erittyvistä proteiineista oli karakterisoimattomia/ennustettuja proteiineja, joten ne voivat olla uusia laji- tai sukukohtaisia efektoreita. A. brassicae -lajissa oli eniten tuntemattomia/ennustettuja/hypoteettisia proteiineja efektoriehdokkaiden joukossa. Huomasimme, että suurin osa efektoreista oli yhteisiä ja päällekkäisiä kuuden lajin välillä, mikä viittaa siihen, että patogeneesin laajat mekanismit, joihin liittyy proteiiniperäisiä efektoreja, voivat olla säilyneitä suvun sisällä. Löysimme kuitenkin kaksi kopiota beeta/gamma-kristallin-kertausta sisältävästä proteiinista, jota esiintyy vain A. brassicae- ja A. brassicicola -lajeissa, mutta ei neljässä muussa Alternaria-lajissa. Tarkempi haku NCBI:n nr-tietokannasta osoitti, että tämä proteiini puuttui kokonaan Alternaria-suvusta, ja lähimmät vastineet löytyivät muista kasvipatogeeneista eli Macrophomina- ja Fusarium-lajeista. Sen toiminnasta tai patogeenisyyteen liittyvästä roolista missään lajissa ei kuitenkaan ole tietoa.
Voimme myös todeta, että joillakin A. brassicae -lajin efektoreilla on potentiaalia kehittyä adaptiivisesti, koska ne olivat myös merkitsevästi yhteydessä genomin toistorikkaisiin alueisiin (P-arvo: 0,0003; Z-pistemäärä: – 2,8778). Tämän vuoksi tarvitaan populaatiotason analyysejä positiivisen valinnan kohteena olevien efektoreiden tunnistamiseksi, mikä voisi valottaa patogeenisuuden evoluutiota A. brassicae -lajissa. Tässä tutkimuksessa tunnistetut efektorit paljastavat, että A. brassicaen patogeneesiin voi osallistua monenlaisia proteiineja. Näistä efektoreista 39:n ennustettiin olevan CAZymejä, joilla on erilaisia tehtäviä soluseinän ja siihen liittyvien polysakkaridien hajottamisessa. A. brassicae -bakteerin genomi sisälsi kaksi nekroosia ja etyleeniä indusoivaa peptidiproteiinia, joiden on todettu osallistuvan useiden patogeenien, kuten oomykeettien ja nekrotrofien, patogeneesiin. A. alternatan Hrip1:n (Hypersensitive response inducing protein 1) on hiljattain osoitettu tunnistavan MdNLR16:n klassisella geenin ja geenin välisellä tavalla, ja Hrip1:n poistaminen A. alternatasta lisää sen virulenssia . Hrip1-homologia esiintyy myös A. brassicae -bakteerissa, mutta sen ei oleteta erittyvän solun ulkopuolelle, mutta tämä on vielä tarkistettava kokeellisesti. Geenin ja geenin välillä tunnistettavien efektoreiden esiintyminen avaa mahdollisuuden tunnistaa isännän täydentäviä R-geenejä, joita voidaan hyödyntää resistenttien lajikkeiden tai viljelykasvien kehittämisessä.