Qu’est-ce que la ChIP ?
L’immunoprécipitation de la chromatine, ou ChIP, est une technologie à base d’anticorps utilisée pour enrichir sélectivement des protéines spécifiques de liaison à l’ADN ainsi que leurs cibles d’ADN. La ChIP est utilisée pour étudier une interaction protéine-ADN particulière, plusieurs interactions protéine-ADN, ou des interactions à travers le génome entier ou un sous-ensemble de gènes.
La ChIP utilise des anticorps qui reconnaissent et lient sélectivement des protéines, y compris des histones, des modifications d’histones, des facteurs de transcription et des cofacteurs, pour fournir des informations sur les états de la chromatine et la transcription des gènes. Une combinaison d’analyse protéomique et de techniques de biologie moléculaire utilisées dans le ChIP permettent de comprendre l’expression et la régulation des gènes dans les cellules ou les tissus d’intérêt.
Quand le ChIP est-il utilisé ?
Typiquement, le ChIP est utilisé pour identifier l’abondance relative d’une protéine spécifique ou d’une modification protéique spécifique à une certaine région du génome. Le ChIP peut être utilisé pour répondre à une multitude de questions scientifiques impliquant l’interaction des protéines et de la chromatine. Par exemple, la ChIP peut être utilisée pour comparer la présence de certaines protéines à divers loci, cartographier les diverses protéines à travers une région génomique d’intérêt, ou quantifier la liaison des protéines à un gène inductible en réponse à un stimulus au fil du temps.
Comment fonctionne la ChIP ?
Le principe du ChIP est relativement simple et repose sur l’utilisation d’un anticorps pour isoler, ou précipiter, une certaine protéine, histone, facteur de transcription ou cofacteur et sa chromatine liée à partir d’un mélange de protéines qui a été extrait de cellules ou de tissus. D’où le nom de la technique : Immunoprécipitation de la chromatine. Dans la ChIP-PCR ou la ChIP-seq, les fragments d’ADN enrichis par l’immunité peuvent ensuite être identifiés et quantifiés à l’aide de réactifs PCR ou qPCR largement disponibles et de technologies de séquençage de nouvelle génération (NGS).
Qu’est-ce que la ChIP native (N-ChIP) par rapport à la ChIP réticulée (X-ChIP) ?
Il existe 2 types de techniques de ChIP qui peuvent être réalisées en fonction de la question expérimentale et du matériel de départ de l’expérience : 1) le ChIP natif (N-ChIP) et 2) le ChIP réticulé (X-ChIP). Les deux types de ChIP présentent des avantages et des inconvénients :
- Dans le N-ChIP, aucun agent fixateur n’est utilisé pour réticuler les protéines à la chromatine. Au lieu de cela, la chromatine native est isolée à partir de noyaux cellulaires qui sont digérés avec une nucléase. Comme les anticorps sont produits contre des antigènes non fixés, la N-ChIP offre l’avantage d’une meilleure reconnaissance et d’une meilleure liaison des anticorps à leurs antigènes cibles. La PCR peut ne pas être nécessaire pour l’analyse en aval en raison de la forte abondance des protéines histones. Bien que ces avantages fassent de la N-ChIP une méthode intéressante, elle ne peut être utilisée que pour la détection des histones. En outre, la perte de liaison des protéines au cours des étapes de digestion de la chromatine et d’immunoprécipitation peut biaiser les données ou empêcher les analyses appropriées.
- Dans la X-ChIP, des fixateurs chimiques tels que le formaldéhyde sont utilisés pour réticuler la protéine d’intérêt à l’ADN et la fragmentation de la chromatine est réalisée par sonication ou digestion par nucléase. L’avantage de la X-ChIP est qu’elle peut être utilisée avec des protéines histones et non-histones et qu’elle nécessite généralement moins de matériel cellulaire de départ que la N-ChIP. La X-ChIP minimise également les risques de perte de protéines de la chromatine pendant l’extraction, ce qui permet la détection d’interactions protéiques transitoires. Cependant, l’étape de précipitation est moins efficace et l’amplification de l’ADN par PCR est nécessaire pour les analyses en aval.
Quels sont les différents types de tests ChIP ?
Une fois l’immunoprécipitation de la chromatine elle-même terminée, plusieurs analyses en aval peuvent être menées sur la chromatine purifiée et les protéines associées, les histones, les facteurs de transcription et les cofacteurs. Les méthodes les plus courantes pour l’analyse d’un seul gène et l’analyse du génome entier sont la qPCR et la ChIP-seq, respectivement. La PCR et la ChIP-chip sont également des options pour l’analyse en aval.
5.1 Quels sont les avantages de la ChIP-PCR ?
La ChIP-PCR est réalisée pour analyser les modifications des histones et/ou la liaison des protéines à un sous-ensemble connu de loci cibles dans le génome. Dans la ChIP-PCR, les fragments d’ADN enrichis par l’immunité sont identifiés et quantifiés à l’aide de réactifs et de technologies PCR ou qPCR largement disponibles. La ChIP-qPCR permet d’effectuer des comparaisons rapides et quantitatives de régions spécifiques du génome dans plusieurs échantillons. Cette méthode est moins coûteuse et plus efficace en termes de temps que les méthodes de séquençage du génome entier.
5.2 Quels sont les avantages de la ChIP-chip ?
La technologie ChIP-chip fait référence à l’utilisation d’une puce à ADN pour analyser les fragments d’ADN enrichis par ChIP. L’utilisation de la technologie des puces à ADN en tuilage génomique permet une analyse du génome entier des protéines qui sont liées à l’ADN isolé et génère une carte génomique à haute résolution de la liaison aux protéines et des modifications des protéines. La puce ChIP a de multiples usages dans la recherche fondamentale ainsi que dans la recherche sur les maladies. Par exemple, elle peut être utilisée pour identifier les sites de liaison des facteurs de transcription, des amplificateurs et des répresseurs et pour comparer ces types de protéines liées dans des échantillons témoins et pathologiques. Cependant, depuis que le coût de la NGS a considérablement diminué et que des résultats similaires sont obtenus à l’aide de la ChIP-seq, davantage de personnes choisissent de réaliser la ChIP-seq au lieu de la ChIP-chip.
5.3 Quels sont les avantages de la ChIP-seq ?
Similaire à la ChIP-chip, la ChIP-seq fournit des informations sur la liaison des protéines à l’échelle du génome. Cependant, contrairement à la ChIP-chip, la ChIP-seq utilise la technologie NGS pour identifier les fragments d’ADN et les cartographier par rapport au génome entier.
La technologie d’amplification de l’ADN plus contemporaine permet de réaliser des analyses robustes en quelques jours avec une faible quantité d’ADN d’entrée. Lorsque le matériel de départ est rare, ces avancées technologiques dans les méthodes de préparation des librairies ont rendu possibles les expériences ChIP-seq.
En outre, une nouvelle technologie dans laquelle les échantillons d’ADN sont marqués de manière unique avec de courtes séquences, connues sous le nom de codes-barres, permet maintenant de regrouper des fragments individuels dans une seule voie de séquençage pour une analyse multiplexée. Cela a considérablement augmenté l’efficacité et diminué le coût des expériences de séquençage de l’ADN, ce qui soutient davantage les applications de ChIP-seq.
Ensemble, en raison des progrès de la technologie de séquençage de l’ADN, l’avantage de ChIP-seq est qu’un grand nombre d’échantillons d’ADN enrichis en ChIP peuvent être séquencés à bas prix dans une période de temps relativement courte avec une sensibilité et une précision plus élevées que la ChIP-chip.
Quelles sont les différentes étapes du test ChIP ?
Les essais ChIP suivent un protocole général :
- Réticulation des protéines à l’ADN pour X-ChIP uniquement
- Lyses cellules
- Fragmentation de la chromatine par digestion (pour X-ChIP et N-ChIP) ou par cisaillement basé sur la sonication-.(pour X-ChIP seulement)
- Immunoprécipitation en utilisant des anticorps spécifiques
- Nettoyage de l’ADN pour les analyses en aval
- Analyse de l’ADN par PCR, qPCR, microarray, ou NGS
Important, les contrôles positifs et négatifs à chaque étape sont intégraux pour déterminer si une expérience ChIP a été réussie.
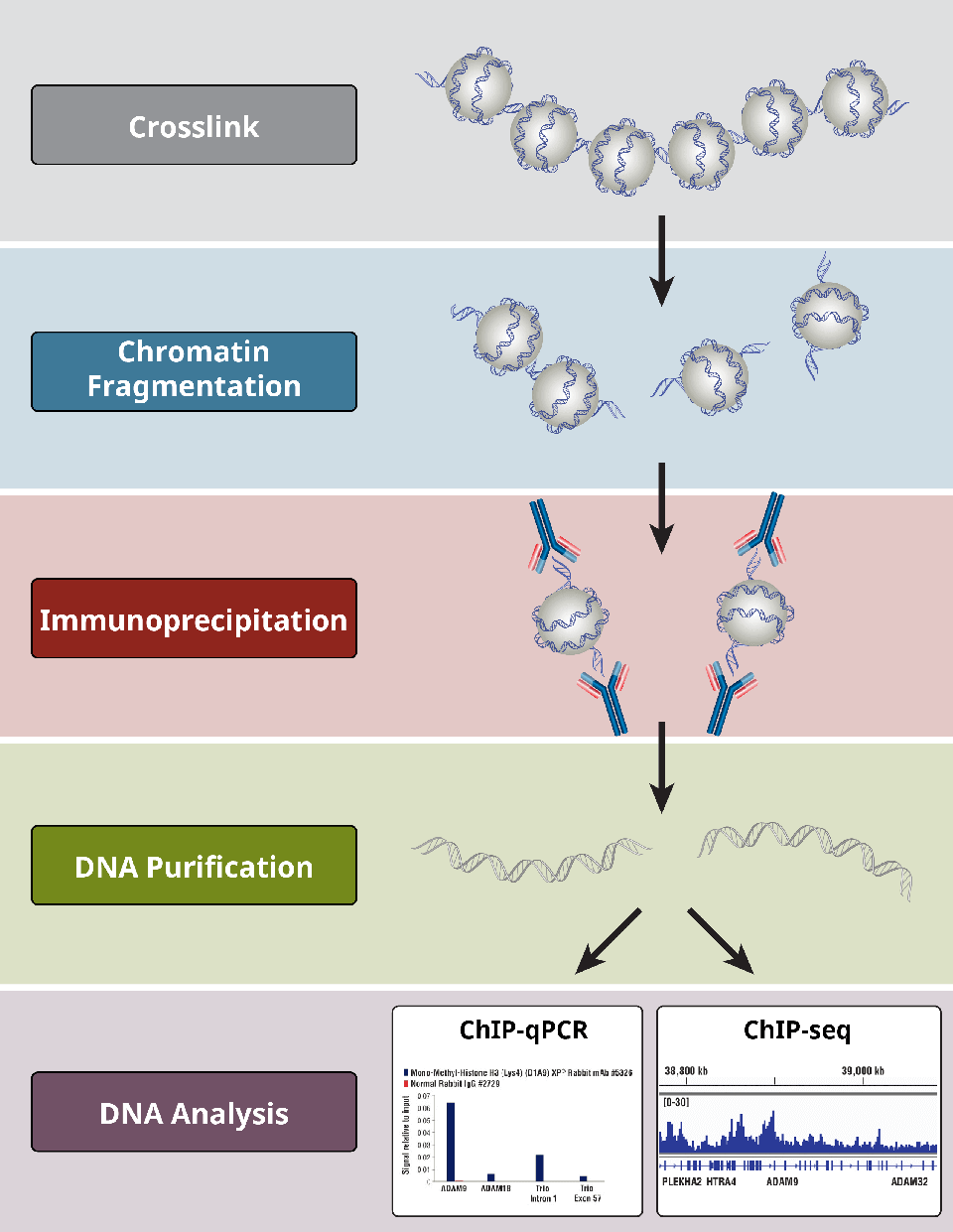
Vue d’ensemble des étapes les plus critiques d’un protocole ChIP.
Vue d’ensemble des étapes les plus critiques d’un protocole ChIP.
6.1 Comment réticuler les cellules et les tissus pour le ChIP ?
Les réactifs de réticulation sont utilisés pour » fixer » les protéines à l’ADN qu’elles lient. Les réactifs à base de formaldéhyde sont généralement utilisés pour réaliser cette fixation. Les cellules et les tissus sont généralement fixés de manière similaire, mais les tissus nécessitent un temps de fixation plus long et une livraison de fixation plus rapide afin de perméabiliser rapidement le tissu cible avant qu’il ne commence à dégénérer.
La sur-fixation de la chromatine peut réduire l’efficacité de la fragmentation par sonication, en plus d’inhiber la liaison des anticorps à leurs cibles protéiques. Par conséquent, le temps de fixation doit être déterminé empiriquement afin de permettre une liaison anticorps-antigène maximale tout en obtenant une réticulation idéale des protéines à leur ADN cible.
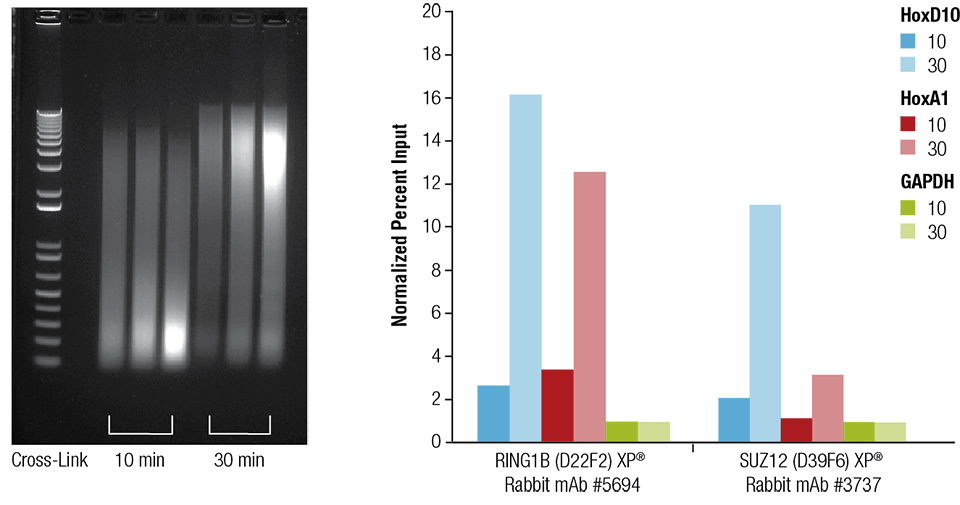
Le cœur (H), le cerveau (B) et le foie (L) de souris ont été réticulés pendant 10 ou 30 minutes comme indiqué (panneau de gauche). La chromatine a été préparée et soniquée pendant 4 minutes. La ChIP a été réalisée en utilisant la chromatine préparée à partir de tissu cardiaque avec les anticorps indiqués en utilisant le kit SimpleChIP® Plus Sonication Chromatin IP Kit #56383 et l’ADN enrichi a été quantifié par PCR en temps réel en utilisant des amorces pour les gènes indiqués (panneau de droite). La quantité d’ADN immunoprécipité dans chaque échantillon est représentée comme un signal normalisé par rapport au loci négatif GAPDH, qui est égal à 1.
Le cœur (H), le cerveau (B) et le foie (L) de la souris ont été réticulés pendant 10 ou 30 minutes comme indiqué (panneau de gauche). La chromatine a été préparée et soniquée pendant 4 minutes. La ChIP a été réalisée en utilisant la chromatine préparée à partir de tissu cardiaque avec les anticorps indiqués en utilisant le kit SimpleChIP® Plus Sonication Chromatin IP Kit #56383 et l’ADN enrichi a été quantifié par PCR en temps réel en utilisant des amorces pour les gènes indiqués (panneau de droite). La quantité d’ADN immunoprécipité dans chaque échantillon est représentée sous forme de signal normalisé par rapport au loci négatif GAPDH, qui est égal à 1.
6.2 Comment fragmenter la chromatine?
La fragmentation de la chromatine est essentielle au succès d’une expérience de ChIP. La fragmentation de la chromatine est nécessaire pour solubiliser la chromatine et permet sa coprécipitation. En outre, la résolution du test ChIP dépend de la fragmentation de la chromatine, puisque la taille du fragment d’ADN détermine la résolution du test ChIP.
La digestion enzymatique utilise la nucléase micrococale (MNase), qui clive l’ADN double brin entre les nucléosomes pour générer des fragments de chromatine. Alors qu’une digestion complète par la MNase génère des fragments d’ADN de 150 paires de bases (mono-nucléosomes), une digestion incomplète génère des fragments d’ADN entre 150 et 750 paires de bases (mono-, di- et tri-nucléosomes). La sonication utilise une force mécanique pour fragmenter la chromatine. La sonication fragmente la chromatine entre et à l’intérieur des nucléosomes, générant une gamme de fragments de chromatine entre 150 et 1000 paires de bases.
Pour la X-ChIP, on utilise soit la digestion enzymatique soit la sonication pour cisailler la chromatine. Les conditions de sonication dans le protocole ChIP par sonication doivent être déterminées empiriquement, car elles diffèrent selon le type de cellule et les conditions expérimentales. Les conditions de digestion sont plus cohérentes entre les différents types de cellules et de tissus, mais la taille du fragment de chromatine doit tout de même être analysée avant l’IP.
Pour la N-ChIP, des nucléases sont utilisées pour fragmenter la chromatine afin de maintenir la liaison des protéines dans les échantillons non fixés. La fragmentation par nucléase doit également être déterminée de manière empirique afin de minimiser la surdigestion de la chromatine.
6.3 Pourquoi utiliser la digestion enzymatique pour la ChIP ?
La digestion par nucléase doit être utilisée pour la N-ChIP car les protéines ne sont pas réticulées à l’ADN et les conditions difficiles associées à la fragmentation par sonication entraîneraient la dissociation des protéines de la chromatine de l’ADN. La N-ChIP est idéale pour analyser les interactions protéine histone-ADN, car la liaison histone-ADN est très forte et stable. Cependant, la N-ChIP ne fonctionne pas bien pour l’analyse de la liaison chromatinienne des facteurs de transcription et des cofacteurs.
La digestion enzymatique ou la sonication peuvent être utilisées pour fragmenter la chromatine dans la X-ChIP. Les avantages de la digestion enzymatique comprennent la cohérence de la fragmentation et des conditions de fragmentation douces (chaleur et détergent plus faibles) qui préservent mieux l’intégrité de la chromatine et des épitopes d’anticorps, ce qui entraîne un enrichissement immunitaire accru de la chromatine liée au facteur de transcription et au cofacteur.
6.4 Pourquoi utiliser la sonication pour fragmenter la chromatine pour la ChIP ?
Contrairement à la fragmentation de la chromatine obtenue par digestion enzymatique, la sonication repose sur des forces mécaniques pour fragmenter la chromatine en plus petits morceaux. La taille idéale des fragments de chromatine pour l’enrichissement immunitaire se situe entre 200 et 1000 paires de bases. La sonication est la méthode traditionnelle utilisée pour fragmenter la chromatine et peut être réalisée à l’aide d’un sonicateur à sonde traditionnel ou de sonicateurs à bain d’eau plus avancés qui fournissent une sonication plus ciblée. La sonication génère des fragments de chromatine véritablement aléatoires ; cependant, elle nécessite une optimisation poussée sur différentes lignées cellulaires et différents tissus et est difficile à reproduire d’une expérience à l’autre. La nécessité de tampons hautement détergents et la chaleur générée pendant la sonication peuvent endommager l’intégrité à la fois de la chromatine et des épitopes d’anticorps sur les protéines chromatiniennes.
6.5 Optimisation de la sonication de la chromatine pour la ChIP
La fragmentation de la chromatine basée sur la sonication utilise traditionnellement des tampons hautement détergents et génère de la chaleur, les deux pouvant endommager l’intégrité de la chromatine et des épitopes d’anticorps. Par conséquent, la quantité de sonication utilisée pour fragmenter la chromatine doit être déterminée expérimentalement pour différentes lignées cellulaires et différents tissus. Il faut identifier et utiliser la quantité minimale de sonication nécessaire pour générer des fragments d’ADN de 150 à 1000 paires de bases afin de minimiser les dommages à la chromatine.
Avant de se lancer dans un test ChIP complet avec une analyse en aval par qPCR, puce à ADN ou NGS, il faut utiliser l’électrophorèse sur gel pour analyser les échantillons de chromatine soniqués pendant différents temps. La taille des fragments dépend du temps de sonication – la taille des fragments diminue lorsque le temps de sonication augmente. Cependant, les données suggèrent qu’un temps de sonication plus long ne permet pas d’obtenir de meilleurs résultats. Par conséquent, l’exécution de l’ADN immunoprécipité purifié sur un gel et la détermination de la taille idéale du fragment est une méthode simple pour déterminer la quantité minimale de sonication requise pour la taille d’ADN souhaitée et pour éviter les dommages inutiles à la chromatine.

La chromatine digérée par une enzyme a été exécutée sur un gel d’agarose. Le couloir 1 montre une chromatine sous-digérée. Le couloir 2 montre la chromatine correctement digérée, et le couloir 3 montre la chromatine qui est surdigérée.
La chromatine digérée par une enzyme a été passée sur un gel d’agarose. Le couloir 1 montre la chromatine qui est sous-digérée. Le couloir 2 montre la chromatine correctement digérée, et le couloir 3 montre la chromatine qui est surdigérée.
6.6 Comment choisir un anticorps pour le ChIP ?
Choisir un anticorps approprié pour une expérience de ChIP fait partie intégrante de son succès. Les anticorps utilisés dans une expérience ChIP doivent être spécifiques de la protéine d’intérêt et avoir une haute affinité pour l’antigène. Le meilleur choix d’anticorps pour une expérience ChIP ou ChIP-seq est un anticorps validé par ChIP ou ChIP-seq. Si aucun anticorps validé par ChIP n’est disponible pour le gène d’intérêt, le meilleur choix suivant est un anticorps qui a été validé par IP. Il est important de noter que tous les anticorps validés en IP ne fonctionnent pas en ChIP et que tous les anticorps validés en ChIP ne fonctionnent pas en ChIP-seq. En outre, plus un anticorps est validé dans d’autres applications, telles que western, IP, IF, flow et IHC, plus on peut avoir confiance dans la performance et la spécificité de l’anticorps. La validation d’un anticorps doit être suivie de la détermination empirique de la concentration optimale de l’anticorps, ainsi que des conditions de lavage IP.
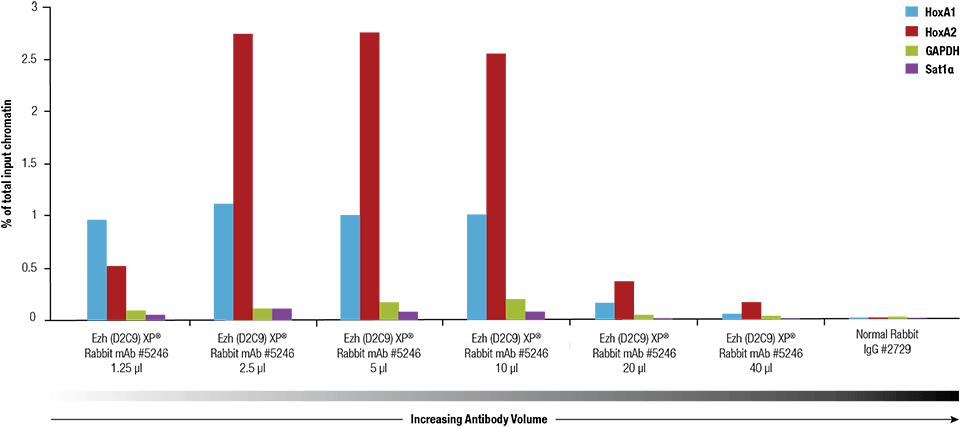
Ezh2 (D2C9) XP® Rabbit mAb #5246 a été titré à l’aide du SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 sur de la chromatine réticulée préparée à partir de 4 x 106 cellules NCCIT.
Ezh2 (D2C9) XP® Rabbit mAb #5246 a été titré en utilisant le SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 sur de la chromatine réticulée préparée à partir de 4 x 106 cellules NCCIT.
6.7 Comment réaliser l’immunoprécipitation
Les anticorps sont utilisés pour capturer une protéine d’intérêt et son ADN lié. La concentration d’anticorps doit être déterminée de manière empirique, avec un point de départ général de 0,5-2,0 μg d’anticorps utilisés pour 10 μg d’ADN chromatinien (correspond à environ 4 x 106 cellules). La rigueur des tampons et les temps de lavage doivent également être déterminés empiriquement car ils dépendent de l’affinité de l’anticorps pour son antigène cible. Généralement, les incubations anticorps:chromatine se font pendant 2 heures à une nuit.
Les complexes anticorps-antigène (+ADN) sont capturés par affinité sur une résine liant les anticorps. Dans les expériences de ChIP, cette résine est généralement composée de billes magnétiques, de sépharose ou d’agarose de qualité ChIP conjuguées à la protéine A et/ou à la protéine B. Les anticorps se lient aux billes de protéine A et/ou de protéine G avec diverses affinités selon l’espèce dans laquelle ils ont été développés et le sous-type IgG de leur chaîne lourde. Les billes sont généralement incubées avec anticorps:chromatine pendant 2 à 4 heures.
Des étapes de lavage sont nécessaires pour éliminer la chromatine non liée aux anticorps, suivies de l’inversion des liaisons croisées (pour X-ChIP) et de la purification de l’ADN. En outre, une IP de contrôle IgG doit être réalisée pour déterminer le bruit de fond (signal:bruit). Des anticorps de contrôle positif (c’est-à-dire l’histone H3 totale) et/ou des amorces qPCR de contrôle positif (pour des loci de liaison de protéines cibles positifs et négatifs connus) doivent également être inclus pour déterminer la liaison non spécifique. Pour des résultats optimaux, le CQ de la PI de chromatine par qPCR doit être effectué avant l’analyse NGS en aval.
6.8 Comment éluer la chromatine des billes de protéine A/G
La chromatine est éluée des billes de protéine A/G à l’aide de détergent et de chaleur. Un « vortex » à faible vitesse ou un mélange est nécessaire pour maintenir les billes en suspension et pour augmenter l’élution de la chromatine.
6.9 Comment inverser les liaisons croisées de la chromatine
Les liaisons croisées sont inversées par une chaleur élevée et un sel élevé (ces deux éléments sont vitaux). On ajoute également de la protéinase K pour digérer les protéines chromatiniennes associées et des anticorps ajoutés, ce qui permet une purification plus efficace de l’ADN en aval.
6.10 Comment purifier l’ADN
Après l’élimination des liaisons transversales de la chromatine, l’ADN est soit purifié par des méthodes classiques de phénol-chloroforme suivies d’une précipitation à l’éthanol, soit par des kits de purification de l’ADN sur colonne.
Comment analyser l’ADN enrichi
Une fois l’ADN purifié, plusieurs analyses en aval peuvent être réalisées, notamment la ChIP-PCR, la ChIP-qPCR, la ChIP-chip et la ChIP-seq.
7.1 Analyse ChIP-PCR et ChIP-qPCR
Les analyses ChIP-PCR et ChIP-qPCR sont les meilleures pour l’analyse de gènes uniques et peuvent être utilisées pour amplifier et quantifier des fragments spécifiques d’ADN de manière rapide et rentable.
7.2 Analyse ChIP-chip
L’analyse ChIP-chip utilisant des puces à ADN en tuile pour créer une carte haute résolution, à l’échelle du génome, de la liaison et de la modification des protéines.
7.3 Analyse ChIP-seq
L’analyse ChIP-seq utilise la technologie NGS standard pour aligner l’ADN purifié avec des génomes entiers préalablement annotés pour identifier des profils de liaison protéique à l’échelle du génome.