Caractéristiques génomiques d’A. brassicae et de deux autres isolats co-infectants de A. alternata
Nous avons séquencé les génomes de deux isolats de A. alternata (PN1 et PN2) qui co-infectaient B. juncea avec A. brassicae. L’assemblage de A. brassicae a été décrit précédemment. En bref, l’assemblage était constitué de neuf chromosomes complets et d’un chromosome dont les répétitions télomériques manquaient à l’une des extrémités. En dehors de ces chromosomes, il y avait six contigs dont l’un d’entre eux avait une taille de ~ 1 Mb, qui peuvent ensemble constituer un chromosome inutile (Fig. 1). Le N50 de l’assemblage de A. brassicae était de 2,98 Mb (Tableau 1). Les deux isolats co-infectant B. juncea ont été identifiés comme étant A. alternata sur la base de leurs séquences ITS et GAPDH. Les assemblages Aat_PN1 et Aat_PN2 d’A. alternata étaient composés de 14 contigs totalisant 33,77 Mb, et de 15 contigs totalisant 33,53 Mb, respectivement (Tableau 1). Six contigs dans chacun des deux assemblages contenaient des répétitions télomériques aux deux extrémités et sont donc très probablement des molécules chromosomiques complètes. Quatre autres contigs dans les deux assemblages contenaient des répétitions télomériques sur une extrémité mais étaient de taille similaire aux molécules de chromosomes complets comme décrit dans A. solani . Par conséquent, les assemblages de génome pour les isolats d’A. alternata représentaient dix chromosomes presque complets de chacun des deux isolats.
Les alignements de génome entier avec les Alternaria spp. apparentées ont montré une synténie globale entre les génomes avec des réarrangements mineurs (figure 2). De plus, des séquences mitochondriales ont également été obtenues à partir des données de séquençage pour les deux isolats d’A. alternata. Les génomes mitochondriaux des souches d’A. alternata avaient une taille d’environ 49 783 pb et 50 765 pb respectivement et présentaient une grande similitude avec le génome mitochondrial d’A. alternata publié précédemment.
La prédiction des gènes après le masquage des répétitions a permis d’identifier 11593, 11495 et 11387 gènes dans les assemblages de génomes d’A. brassicae, A. alternata PN1 et PN2, respectivement. Ces chiffres étaient comparables aux nombres de gènes estimés dans d’autres Alternaria spp. (Tableau 1). L’analyse BUSCO a montré que les modèles de gènes prédits dans les trois génomes couvraient 98 % des gènes fongiques conservés en copie unique, ce qui indique que les assemblages sont presque complets. Les gènes prédits ont été annotés de manière exhaustive en utilisant une combinaison de bases de données comme décrit dans la section Méthodes (Fig. 1). En plus de ces trois génomes, nous avons également prédit des gènes de novo dans les assemblages de génomes de trois autres espèces d’Alternaria qui ont été séquencées en utilisant des technologies de longue durée, à savoir A. brassicicola (abra43), A. alternata (ATCC34957) et A. solani (altNL03003) (Tableau 1). Ces six génomes et leurs prédictions génétiques ont été utilisés pour les analyses comparatives des groupes de gènes codant pour les métabolites secondaires et des gènes codant pour les effecteurs.

Sommaire du génome de A. brassicae, (Des pistes circulaires extérieures aux pistes circulaires intérieures) a pseudochromosomes/scaffolds, b Gènes codant pour des protéines, c Eléments répétés, d Eléments transposables (ADN et LTR), e Groupes de métabolites secondaires prédits, f Protéines sécrétées, g Effecteurs prédits
L’analyse phylogénomique attribue un clade distinct pour l’A. brassica infectant et l’A. brassica. brassicae et A. brassicicola au sein du genre Alternaria
Afin de reconstituer avec précision la divergence et la relation entre A. brassicae, les deux isolats d’A. alternata (PN1 et PN2) et les autres espèces d’Alternaria, nous avons effectué des analyses phylogénomiques en utilisant 29 orthologues à copie unique qui présentaient le signal phylogénétique le plus élevé tel que calculé par le programme Mirlo. La sélection des gènes ayant le signal phylogénétique le plus élevé conduit à des phylogénies qui sont plus conformes à l’arbre des espèces. La phylogénie résultante a montré que les espèces Alternaria à grandes spores et Alternaria à petites spores se regroupent séparément en deux clades différents (Fig. 3). Il est intéressant de noter que les deux principaux pathogènes des Brassicas, à savoir A. brassicae et A. brassicicola, se sont regroupés séparément de toutes les autres espèces d’Alternaria, indiquant peut-être une trajectoire d’évolution différente basée sur les préférences d’hôte communes de ces deux espèces.
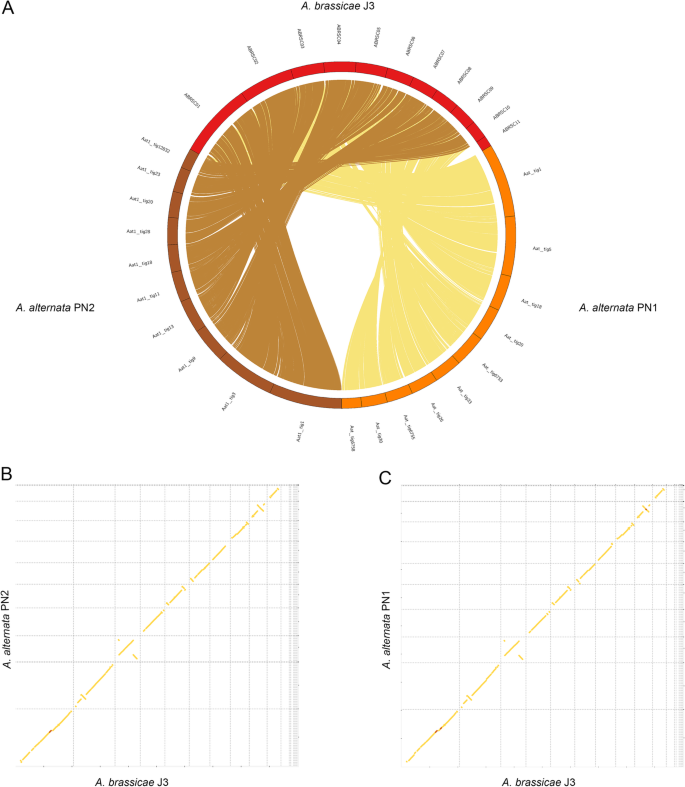
Alignements du génome entier de A. alternata PN1 et PN2 avec A. brassicae. a Tracé circulaire montrant la macrosynténie de A. alternata PN1 et PN2 avec A. brassicae à travers tous les contigs sauf les contigs dispensables (ABRSC11, scaffold13,17,18,19), b et c Dotplots synténiques d’A. brassicae avec A. alternata PN1 et PN2
Analyses comparatives d’A. alternata obtenus à partir de différents hôtes
Nous avons comparé les génomes d’A. alternata PN1 et PN2 (isolés de B. juncea) à celui d’A. alternata ATCC34957 (isolé du sorgho) pour identifier toute différence dans leur contenu génomique qui pourrait permettre à ceux-ci d’infecter deux espèces très différentes. Les alignements du génome entier d’A. alternata PN1 et PN2 avec celui d’A. alternata ATCC34957 ont révélé des niveaux très élevés de synténie et l’absence de toute région spécifique à l’espèce. Nous avons identifié 719, 152 et 586 gènes spécifiques aux isolats entre les trois isolats d’A. alternata, respectivement (fichier supplémentaire 1 : tableau S1). Plus de deux tiers des gènes spécifiques aux isolats dans les trois isolats étaient des protéines non caractérisées ou n’avaient pas d’annotations. Notamment, les trois isolats ne contenaient pas de chromosomes dispensables pouvant conférer une pathogénicité, comme cela a été signalé pour les isolats d’A. alternata infectant de nombreuses cultures fruitières telles que les agrumes, les poires et les pommes. Les répertoires génétiques des trois isolats comprenaient également un nombre et un type similaires d’effecteurs, de CAZymes et de groupes de métabolites secondaires (Tableau 2). De plus, les deux isolats PN1 et PN2 ne provoquent pas de symptômes d’infection par eux-mêmes chez B. juncea dans des conditions épiphytotiques (données non présentées). Nos résultats suggèrent que ces isolats d’A. alternata (PN1 et PN2) peuvent être des pathogènes facultatifs qui mènent un mode de vie saprophyte et peuvent passer à un mode de vie pathogène dans certaines conditions environnementales.
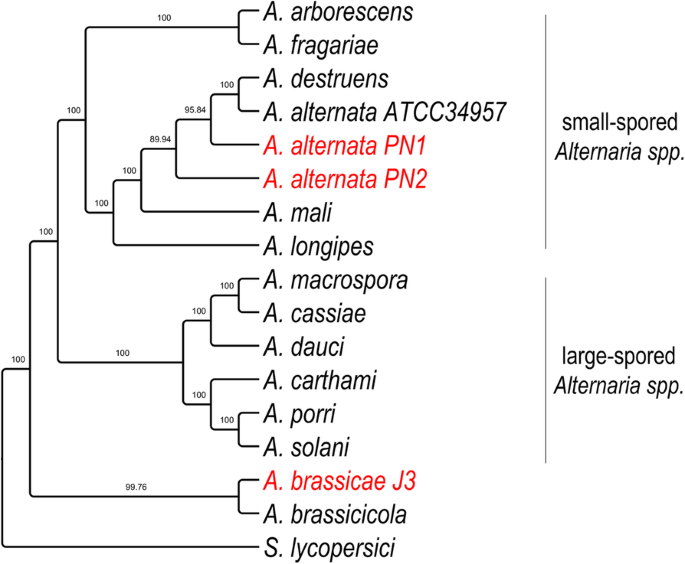
Arbre phylogénétique des espèces d’Alternaria avec S. lycopersici comme outgroup. L’arbre a été construit en utilisant 29 orthologues à copie unique, qui avaient le signal phylogénétique le plus élevé tel que calculé dans Mirlo. Les valeurs de soutien des branches à partir de 1000 répliques bootstrap sont indiquées
Une abondance de régions riches en répétitions et d’éléments transposables chez A. brassicae
Les agents pathogènes des plantes filamenteuses ont tendance à avoir une architecture génomique distincte avec un contenu de répétition plus élevé. L’estimation du contenu en répétitions et le masquage à l’aide de RepeatModeler et RepeatMasker ont révélé que le génome d’A. brassicae était constitué de ~ 9,33 % de répétitions, contre 2,43 et 2,64 % de répétitions dans les génomes d’A. alternata. Le génome d’A. brassicae présente le contenu le plus élevé en répétitions (~ 9,33%) parmi toutes les espèces d’Alternaria séquencées à ce jour. Notre analyse a montré que le contenu en répétitions diffère significativement entre les isolats d’A. alternata et les autres espèces d’Alternaria pathogènes. Les espèces pathogènes d’Alternaria, en particulier A. brassicae et A. brassicicola, ont un répertoire considérablement plus important d’éléments LTR/Gypsy et LTR/Copia (> 8X) par rapport aux autres isolats d’A. alternata (pathogènes et non pathogènes) (Fig. 4). Les génomes d’A. brassicae et d’A. brassicicola présentaient également une surreprésentation des transposons d’ADN, qui s’élevaient à ~ 5 % du génome, contre < 1 % chez les autres espèces d’Alternaria (Fig. 4).

Comparaison du contenu en répétitions chez six espèces d’Alternaria. La taille des bulles correspond (a) au pourcentage d’éléments transposables (TE) dans le génome, b au nombre de copies du TE dans le génome
Cette prolifération d’ADN répétitif et l’évolution ultérieure de gènes chevauchant ces régions peuvent être la clé du succès évolutif dans lequel ces pathogènes ont réussi à persister sur des générations de conflit coévolutif avec leurs hôtes. La proximité des TE expose potentiellement les gènes aux mutations ponctuelles induites par la répétition (RIP) et donc à une évolution accélérée. La recombinaison ectopique entre des TE similaires peut également donner lieu à de nouvelles combinaisons de gènes et ainsi augmenter la diversité des protéines ou des métabolites.
Présence d’un chromosome dispensable chez A. brassicae à grandes spores
Des chromosomes spécifiques de lignée (LS) ou des chromosomes dispensables (DC) ont été signalés chez plusieurs espèces phytopathogènes dont A. alternata. Les DC chez A. alternata sont connus pour conférer la virulence et la spécificité d’hôte à l’isolat. Les alignements du génome entier d’A. brassicae avec d’autres Alternaria spp. ont révélé qu’un contig d’environ 1 Mb avec d’autres contig plus petits (66-366 kb) était spécifique à A. brassicae et ne présentait aucune synténie avec une région des autres Alternaria spp. Cependant, une synténie partielle a été observée lorsque le contig a été aligné avec les séquences d’autres chromosomes dispensables signalés chez Alternaria spp. Ceci nous a conduit à émettre l’hypothèse que ces contigs ensemble pourraient représenter un DC d’A. brassicae. Pour confirmer cette hypothèse, nous avons recherché dans les contigs la présence des gènes AaMSAS et ALT1, qui sont des gènes marqueurs connus pour les chromosomes dispensables chez Alternaria spp. Nous avons trouvé deux copies du gène AaMSAS dans le cadre de deux groupes de biosynthèse de métabolites secondaires sur le contigu de 1 Mb. Cependant, nous n’avons pas trouvé d’homologue du gène ALT1. De plus, le contenu en répétitions des contigs (ABRSC11, scaffold 13, 17, 18 et 19) a été comparé au génome entier. Le contenu génétique des contigs spécifiques à la lignée était significativement plus faible que celui des chromosomes centraux (Tableau 3). Inversement, les contigs du DC étaient fortement enrichis en contenu TE par rapport aux chromosomes centraux (Tableau 3).
Bien que, le DC n’était pas enrichi en gènes codant pour des protéines sécrétées, la proportion de gènes effecteurs sécrétés était 30% plus élevée par rapport aux chromosomes centraux. Toutes les preuves ci-dessus indiquent qu’A. brassicae peut en effet abriter une DC. Les CD chez Alternaria spp. n’ont été signalées jusqu’à présent que chez les Alternaria spp. à petites spores et aucune espèce d’Alternaria à grandes spores n’est connue pour abriter des CD. Il reste à voir si les CD contribuent à la virulence d’A. brassicae. Les études futures impliqueraient la caractérisation du chromosome dispensable chez A. brassicae et la corrélation de sa présence avec la pathogénicité de différents isolats.
L’analyse orthologique révèle des gènes spécifiques aux espèces avec des rôles putatifs dans la virulence
Les différences dans le contenu des gènes et la diversité au sein des gènes contribuent à l’adaptation, la croissance et la pathogénicité. Afin de cataloguer les différences dans le contenu génétique au sein du genre Alternaria et des Dothidéomycètes, nous avons effectué une analyse d’orthologie sur l’ensemble combiné de 3 60 216 protéines provenant de 30 espèces différentes (dont 16 espèces d’Alternaria) appartenant aux Dothidéomycètes (fichier additionnel 2 : tableau S2) dont 3 45 321 protéines ont pu être assignées à au moins un des orthogroupes. Nous avons identifié 460 gènes spécifiques d’A. brassicae qui étaient présents chez A. brassicae mais absents chez toutes les autres espèces d’Alternaria (fichier supplémentaire 3 : tableau S3). Ces gènes spécifiques à l’espèce comprenaient 35 gènes codant pour des protéines sécrétées, dont 11 ont été prédits comme effecteurs. De plus, 20 de ces gènes spécifiques à une espèce étaient présents sur le DC. Un grand nombre de ces protéines appartenaient à la catégorie des protéines non caractérisées et sans fonction connue. Afin de vérifier si ces gènes spécifiques à une espèce sont le résultat d’une évolution adaptative dans les régions riches en répétitions du génome, nous avons effectué un test de permutation pour comparer le chevauchement des régions riches en répétitions et des éléments transposables avec un ensemble de gènes aléatoires au chevauchement de ces gènes spécifiques à une espèce. Nous avons constaté que ces gènes spécifiques aux espèces se chevauchaient de manière significative avec les régions riches en répétitions (valeur P : 9,99e-05 ; Z-score : – 4,825) et les éléments transposables (valeur P : 0,0460 ; Z-score : 2,539) dans le génome.
Profil des métabolites secondaires d’A. brassicae et son association avec les éléments transposables (TEs)
Les genres Alternaria et Cochliobolus sont connus pour être les principaux producteurs de toxines de métabolites secondaires spécifiques à l’hôte. Les Alternaria spp. sont particulièrement connues pour la production de métabolites secondaires chimiquement diversifiés, qui comprennent les toxines spécifiques de l’hôte (HST) et les non-HST. Ces métabolites secondaires sont généralement générés par des synthases peptidiques non ribosomales (NRPS) et des synthases polycétides (PKS). Nous avons identifié cinq clusters de gènes SM de type NRPS, 12 clusters de gènes de type PKS et sept clusters de gènes de type terpène chez A. brassicae (Additional file 4 : Table S4). Parmi les cinq groupes NRPS, nous avons pu identifier trois groupes qui produisent des métabolites secondaires connus, à savoir la destruxine B, la toxine HC et le diméthylcoprogène (sidérophore), dont le rôle dans la virulence est connu.
Le groupe de gènes responsable de la production de diméthylcoprogène (sidérophore) chez A. brassicae se compose de 22 gènes, dont les principaux gènes de biosynthèse, les oxydoréductases et les transporteurs de sidérophore. Les sidérophores sont des composés chélateurs du fer, utilisés par les champignons pour acquérir du fer ferrique extracellulaire et ont été signalés comme étant impliqués dans la virulence fongique. L’identification du groupe de gènes responsable de la synthèse des sidérophores permettrait d’étudier les sidérophores et leur rôle dans la pathogénicité chez A. brassicae. De plus, un cluster de type PKS composé de 12 gènes, responsable de la production de mélanine a également été identifié (Additional file 4 : Table S4). Le cluster de biosynthèse de la mélanine a été décrit précédemment pour A. alternata. De même, le facteur de transcription Amr1, qui induit la production de mélanine, a été caractérisé chez A. brassicicola et est connu pour supprimer la virulence . Cependant, le rôle de la mélanine dans la virulence est ambigu et spécifique à chaque espèce.
Les phytopathogènes appartenant au genre d’Alternaria semblent avoir une capacité dynamique à acquérir un nouveau potentiel de métabolites secondaires pour coloniser de nouvelles niches écologiques. L’explication la plus parcimonieuse de cette acquisition dynamique du potentiel en métabolites secondaires est le transfert horizontal de gènes au sein du genre Alternaria et éventuellement avec d’autres genres. Il existe de nombreuses preuves dans la littérature qu’une grande partie des HST d’Alternaria est portée par les chromosomes dispensables et que l’échange de ces chromosomes peut élargir la spécificité de l’hôte. Nous avons également identifié un cluster NRPS, codant probablement pour la HC-toxine dans l’un des DCs (scaffold 18) (Additional file 4 : Table S4). La HC-toxine est un déterminant de virulence connu du phytopathogène Cochliobolus carbonum, qui infecte les génotypes de maïs dépourvus d’une copie fonctionnelle de HM1, une carbonyl réductase qui détoxifie la toxine. Un rapport récent a montré que A. jesenskae pouvait également produire de l’HC-toxine, ce qui en fait le seul champignon autre que C. carbonum à produire cette toxine. La présence du cluster de gènes de la HC-toxine, un déterminant de virulence chez C. carbonum, dans un DC d’A. brassicae indique que le transfert horizontal de gènes entre espèces peut être plus commun que prévu.
A part le transfert horizontal de gènes, la duplication, la divergence et la perte rapides des gènes SM peuvent également contribuer à ce que l’agent pathogène évolue vers de nouvelles capacités métaboliques. Ces processus de duplication et de divergence peuvent être facilités par la proximité des grappes de métabolites secondaires avec les éléments répétés, ce qui les rend vulnérables aux mutations RIP. Nous avons donc testé si les groupes de métabolites secondaires étaient également associés à des régions riches en répétitions. Un test de permutation a été utilisé pour comparer le chevauchement des régions riches en répétitions avec un ensemble de gènes aléatoires et le chevauchement des gènes des groupes de métabolites secondaires. Les groupes de métabolites secondaires chevauchent de manière significative les régions riches en répétitions par rapport à l’ensemble de gènes aléatoires (valeur P : 0,0017 ; score Z : – 2,7963). De même, ces groupes se chevauchent de manière significative avec les éléments transposables parmi les régions riches en répétitions (valeur P : 0,0087 ; score Z : 2,9871). Cela montre que les deux mécanismes décrits ci-dessus pour l’acquisition d’un nouveau potentiel de métabolites secondaires peuvent être possibles dans le cas de A. brassicae. Des analyses à l’échelle des populations au niveau des espèces et du genre peuvent éclairer la prévalence de ces mécanismes au sein du genre Alternaria.
L’analyse de la synténie révèle la base génétique de l’exclusivité de la production de Destruxine B par A. brassicae au sein du genre Alternaria
La Destruxine B représente une classe de depsipeptides cycliques qui est connue pour être l’un des facteurs clés de la pathogénicité d’A. brassicae et a été signalée comme étant une toxine spécifique de l’hôte d’A. brassicae . La destruxine B n’a pas été signalée comme étant produite par aucune des autres espèces d’Alternaria. Nous rapportons ici pour la première fois les clusters de gènes biosynthétiques responsables de la production de la Destruxine B chez A. brassicae. Le groupe se compose de 10 gènes, y compris la principale enzyme biosynthétique codée par un gène NRPS (DtxS1) et l’enzyme limitant la vitesse, DtxS3 (aldo-keto reductase) (Additional file 4 : Table S4). Il est intéressant de noter que l’analyse de la synténie de ce groupe parmi les six espèces d’Alternaria a montré que ces deux gènes n’étaient présents dans aucune des autres espèces d’Alternaria, bien que la synténie globale du groupe ait été maintenue dans toutes ces espèces (Fig. 5). L’absence des gènes clés codant pour les enzymes DtxS1 et DtxS3 dans le cluster Destruxin B chez les autres espèces d’Alternaria explique l’absence de Destruxin B chez ces espèces.

Synténie du cluster Destruxin B chez les six espèces d’Alternaria. DtxS1 et DtxS3 sont marqués en rouge (étiquetés avec les ID de gènes respectifs) et sont absents de toutes les autres espèces d’Alternaria
La Destruxine B a été signalée par l’entomopathogène Metarhizium robertsii , et Ophiosphaerella herpotricha , la cause de la tache morte printanière du bermudagrass, en dehors de A. brassicae. Wang et al. ont décrit le groupe de métabolites secondaires biosynthétiques responsables de la production de la Destruxine chez M. robertsii . Le groupe Destruxin dans les espèces Metarhizhium se compose de deux gènes clés supplémentaires, à savoir DtxS2 et DtxS4. DtxS2 est responsable de la conversion de la Destruxine B en Destruxine A, C, D, et E. Nous n’avons pu trouver aucun gène syntétique dans le cluster qui correspond à DtxS2 ou DtxS4 chez A. brassicae. A. brassicae est connu pour ne produire que la Destruxine B et non les autres formes de Destruxine, ce qui pourrait éventuellement expliquer l’absence de DtxS2 chez A. brassicae. DtxS4 est une aspartate décarboxylase qui est nécessaire pour la production de β-alanine à partir de l’acide aspartique et est essentielle pour la production de la structure du squelette de la Destruxine. De plus, les mutants de délétion de DtxS4 ont été capables de produire de la Destruxine lorsqu’ils ont été supplémentés en β-alanine dans le milieu. Même si DtxS4 n’est pas présent dans le cluster de la Destruxine, A. brassicae possède une copie fonctionnelle de l’aspartate décarboxylase présente ailleurs dans le génome et peut donc éventuellement assurer l’approvisionnement en β-alanine pour la synthèse de la Destruxine B. L’histoire évolutive de la production de la Destruxine B au sein du genre Metarhizhium ressemble beaucoup à celle d’Alternaria. Les pathogènes spécialisés tels que M. acridum sont dépourvus des deux enzymes clés et ne produisent donc pas de Destruxines. Cependant, M. robertsii, un généraliste avec une gamme d’hôtes plus large, produit des Destruxines. On a donc émis l’hypothèse que les Destruxines pourraient être responsables de l’établissement de la gamme d’hôtes étendue de M. robertsii. Une hypothèse similaire peut également être vraie dans le cas d’A. brassicae, qui a une large gamme d’hôtes et peut infecter presque toutes les Brassicaceae. D’autres expériences visant à déterminer la gamme d’hôtes des mutants nuls de Destruxine d’A. brassicae peuvent aider à clarifier le rôle de cette importante phytotoxine dans l’extension de la gamme d’hôtes d’A. brassicae.
Profils CAZyme distincts d’A. brassicae et A. brassicicola au sein du genre Alternaria
Les CAZymes (Carbohydrate-Active enZymes) sont des protéines impliquées dans la dégradation, le réarrangement ou la synthèse des liaisons glycosidiques. Les agents pathogènes des plantes sécrètent une gamme variée de CAZymes qui dégradent les polysaccharides complexes de la paroi cellulaire des plantes. Ils se composent de cinq classes distinctes, à savoir les glycoside hydrolases (GH), les glycosyltransférases (GT), les polysaccharide lyases (PL), les estérases de glucides (CE) et les modules de liaison aux glucides (CBM). Nous avons identifié > 500 CAZymes dans les six Alternaria spp. y compris A. brassicae (fichier additionnel 5 : tableau S5). La distribution des CAZymes d’A. brassicae et d’A. brassicicola différait de celle des autres Alternaria spp. formant ainsi un groupe distinct (Fig. 6). Le nombre d’enzymes à activité auxiliaire ou d’enzymes impliquées dans la dégradation des parois cellulaires végétales varie considérablement entre les différentes espèces comparées. Près de 46% des CAZymes chez A. brassicae étaient sécrétés, parmi lesquels ~ 17% étaient prédits comme effecteurs.

Carte thermique des profils de CAZymes chez 16 espèces du genre Alternaria. Le regroupement des organismes ressemble étroitement à celui de la phylogénie des espèces, à l’exception de A. brassicae et A. brassicicola, qui se regroupent séparément avec l’outgroupe Stemphylium lycopersici. Les CAZymes ont été regroupés selon la classification CAZy
La famille AA9 (anciennement GH61 ; polysaccharide monooxygénases lytiques dépendantes du cuivre (LPMO)) est significativement élargie par rapport aux autres familles CAZy dans le genre Alternaria, chaque espèce contenant > 20 copies du gène. Le nombre de copies dans les espèces d’Alternaria est beaucoup plus élevé que le nombre de copies rapporté pour Botrytis et Fusarium. La famille AA9 est impliquée dans la dégradation des polysaccharides de la paroi cellulaire et est connue pour agir sur une gamme de polysaccharides incluant l’amidon, le xyloglucane, les cellodextrines et le glucomannane. On a émis l’hypothèse que les LPMO ont un double rôle : ils clivent directement les polysaccharides de la paroi cellulaire et agissent comme un générateur de ROS, contribuant ainsi au stress oxydatif qui entraîne la nécrose des tissus végétaux. Il est frappant de constater que 11 des 26 protéines AA9 présentes chez A. brassicae sont censées être des effecteurs sécrétés. La caractérisation de ces CAZymes et leur rôle dans la pathogenèse pourraient faire l’objet d’études supplémentaires.
Profils d’effecteurs superposés au sein du genre Alternaria
Nous avons prédit le répertoire d’effecteurs de six espèces d’Alternaria (tableau 2), y compris A. brassicae, en utilisant le pipeline décrit dans la section méthodes. La plupart des petites protéines sécrétées prédites comme effecteurs étaient des protéines non caractérisées/prédites et peuvent donc être de nouveaux effecteurs spécifiques à une espèce ou à un genre. A. brassicae avait la plus grande proportion de protéines inconnues/prédites/hypothétiques dans l’ensemble des effecteurs candidats. Nous avons constaté que la plupart des effecteurs entre les six espèces étaient communs et se chevauchaient, ce qui suggère que les mécanismes généraux de la pathogenèse impliquant des effecteurs protéiques peuvent être conservés au sein du genre. Cependant, nous avons trouvé que deux copies d’une protéine contenant un pli bêta/gamma-cristallin étaient présentes uniquement chez A. brassicae et A. brassicicola et pas chez les quatre autres espèces d’Alternaria. Une recherche plus poussée dans la base de données nr du NCBI a révélé que cette protéine était complètement absente du genre Alternaria et que les correspondances les plus proches étaient trouvées chez d’autres agents pathogènes des plantes, à savoir les espèces Macrophomina et Fusarium. Cependant, aucune information n’est disponible quant à sa fonction ou son rôle dans la pathogénicité de l’une ou l’autre des espèces.
Nous avons également pu établir que certains des effecteurs chez A. brassicae ont le potentiel d’évoluer de manière adaptative puisqu’ils étaient également associés de manière significative aux régions riches en répétitions du génome (valeur P : 0,0003 ; Z-score : – 2,8778). Des analyses au niveau des populations sont donc nécessaires pour identifier les effecteurs soumis à une sélection positive, ce qui pourrait faire la lumière sur l’évolution du pouvoir pathogène chez A. brassicae. Les effecteurs identifiés dans cette étude révèlent le large éventail de protéines qui peuvent être impliquées dans la pathogenèse d’A. brassicae. 39 de ces effecteurs ont été prédits comme étant des CAZymes ayant divers rôles dans la dégradation de la paroi cellulaire et des polysaccharides associés. Le génome de A. brassicae contenait deux protéines peptidiques induisant la nécrose et l’éthylène, qui ont été impliquées dans la pathogenèse de divers pathogènes, y compris les oomycètes et les nécrotrophes. Il a été récemment démontré que la protéine Hrip1 (Hypersensitive response inducing protein 1) de A. alternata est reconnue par MdNLR16 de manière classique, gène pour gène, et que la délétion de Hrip1 chez A. alternata augmente sa virulence. Un homologue de Hrip1 est également présent chez A. brassicae, mais il n’est pas prévu qu’il soit sécrété en dehors de la cellule, bien que cela doive être vérifié expérimentalement. La présence d’effecteurs qui sont reconnus d’une manière gène pour gène ouvre la possibilité d’identifier des gènes R complémentaires dans l’hôte qui peuvent être utilisés pour développer des variétés ou des cultivars résistants.