Mi a ChIP?
A kromatin immunprecipitáció vagy ChIP egy antitest-alapú technológia, amelyet specifikus DNS-kötő fehérjék és DNS-célpontjaik szelektív dúsítására használnak. A ChIP egy adott fehérje-DNS kölcsönhatás, több fehérje-DNS kölcsönhatás vagy a teljes genomot vagy a gének egy részhalmazát érintő kölcsönhatások vizsgálatára szolgál.
A ChIP olyan antitesteket használ, amelyek szelektíven felismerik és megkötik a fehérjéket, beleértve a hisztonokat, hisztonmódosulásokat, transzkripciós faktorokat és kofaktorokat, hogy információt szolgáltassanak a kromatin állapotáról és a génátírásról. A ChIP-ben alkalmazott proteomikai elemzés és molekuláris biológiai technikák kombinációja lehetővé teszi a génexpresszió és a szabályozás megértését az érdeklődésre számot tartó sejtekben vagy szövetekben.
Mikor használják a ChIP-et?
A ChIP-et tipikusan egy adott fehérje vagy egy adott fehérjemódosítás relatív gyakoriságának azonosítására használják a genom egy bizonyos régiójában. A ChIP számos, a fehérjék és a kromatin kölcsönhatását érintő tudományos kérdés megválaszolására használható. A ChIP például felhasználható bizonyos fehérjék jelenlétének összehasonlítására különböző lokuszokon, a különböző fehérjék feltérképezésére egy érdekes genomi régióban, vagy egy indukálható génhez való fehérjekötődés számszerűsítésére egy ingerre adott időbeli válaszként.
Hogyan működik a ChIP?
A ChIP elve viszonylag egyszerű, és egy antitest használatán alapul, amely egy bizonyos fehérje, hiszton, transzkripciós faktor vagy kofaktor és az ahhoz kötött kromatin izolálására vagy kicsapására szolgál egy sejtekből vagy szövetekből kivont fehérjekeverékből. Innen ered a technika neve: Kromatin immunprecipitáció. A ChIP-PCR vagy ChIP-seq során az immun-dúsított DNS fragmentumok ezután széles körben elérhető PCR vagy qPCR reagensek és újgenerációs szekvenálási (NGS) technológiák segítségével azonosíthatók és számszerűsíthetők.
Mi a natív ChIP (N-ChIP) vs. keresztkötésű ChIP (X-ChIP)?
A kísérleti kérdéstől és a kísérlet kiindulási anyagától függően 2féle ChIP technikát lehet alkalmazni: 1) natív ChIP (N-ChIP) és 2) keresztkötött ChIP (X-ChIP). Mindkét ChIP-típusnak vannak előnyei és hátrányai:
- Az N-ChIP-ben nem használunk rögzítőszert a fehérjék kromatinhoz való keresztkötéséhez. Ehelyett natív kromatint izolálunk sejtmagokból, amelyeket nukleázzal emésztünk. Mivel az antitesteket nem fixált antigének ellen termelik, az N-ChIP előnye, hogy az antitestek jobban felismerik a célantigéneket és jobban kötődnek hozzájuk. A hisztonfehérjék nagy abundanciája miatt nem feltétlenül van szükség PCR-re a downstream analízishez. Bár ezek az előnyök vonzóvá teszik az N-ChIP-et, az csak hisztonok kimutatására használható. Ezenkívül a kromatin emésztési és immunprecipitációs lépések során a fehérjék kötődésének elvesztése torzíthatja az adatokat vagy akadályozhatja a megfelelő elemzéseket.
- Az X-ChIP-ben kémiai fixálószereket, például formaldehidet használnak az érdeklődő fehérje DNS-hez való keresztkötéséhez, és a kromatin fragmentálását szonikációval vagy nukleáz emésztéssel érik el. Az X-ChIP előnye, hogy hiszton és nem hiszton fehérjékkel is alkalmazható, és általában kevesebb sejtes kiindulási anyagot igényel, mint az N-ChIP. Az X-ChIP minimalizálja a kromatinfehérje-vesztés esélyét is az extrakció során, lehetővé téve az átmeneti fehérje kölcsönhatások kimutatását. A kicsapási lépés azonban kevésbé hatékony, és a downstream elemzésekhez PCR-rel történő DNS-amplifikációra van szükség.
Melyek a ChIP-vizsgálatok különböző típusai?
Miután maga a kromatin immunprecipitáció befejeződött, számos downstream elemzés végezhető a tisztított kromatinon és a kapcsolódó fehérjéken, hisztonokon, transzkripciós faktorokon és kofaktorokon. A leggyakoribb módszerek az egyes gének elemzésére és a teljes genom elemzésére a qPCR, illetve a ChIP-seq. A PCR és a ChIP-chip is lehetőség a downstream elemzésre.
5.1. Milyen előnyei vannak a ChIP-PCR-nek?
A ChIP-PCR-t a hisztonmódosulások és/vagy a fehérjék kötődésének elemzésére végzik a genomban lévő céllociok ismert részhalmazához. A ChIP-PCR során az immun által dúsított DNS-fragmentumokat széles körben elérhető PCR- vagy qPCR-reagensek és technológiák segítségével azonosítják és számszerűsítik. A ChIP-qPCR segítségével a genomon belüli specifikus régiók gyors és kvantitatív összehasonlítása több mintában is elérhető. Ez olcsóbb és időhatékonyabb, mint a teljes genom szekvenálási módszerek.
5.2. Mik a ChIP-chip előnyei?
A ChIP-chip technológia a DNS-mikroarray chip felhasználására utal a ChIP-immunokkal dúsított DNS-fragmentumok elemzésére. A genomi tiling microarray technológia alkalmazása lehetővé teszi az izolált DNS-hez kötött fehérjék egész genomra kiterjedő elemzését, és nagy felbontású genomi térképet hoz létre a fehérjék kötődéséről és a fehérjemódosításokról. A ChIP-chip többszörösen felhasználható az alapkutatásban és a betegségalapú kutatásban egyaránt. Például felhasználható a transzkripciós faktorok, enhancerek és represszorok kötőhelyeinek azonosítására, valamint az ilyen típusú kötött fehérjék összehasonlítására kontroll és kóros mintákban. Mivel azonban az NGS költségei jelentősen csökkentek, és a ChIP-seq segítségével hasonló eredmények érhetők el, egyre többen választják a ChIP-seq elvégzését a ChIP-chip helyett.
5.3. Mik a ChIP-seq előnyei?
A ChIP-chiphez hasonlóan a ChIP-seq is információt szolgáltat a fehérjék genomszintű kötődéséről. A ChIP-chip-pel ellentétben azonban a ChIP-seq az NGS-technológiát használja a DNS-fragmentumok azonosítására és a teljes genomra való leképezésére.
A korszerűbb DNS-amplifikációs technológia lehetővé teszi, hogy a robusztus elemzést alacsony mennyiségű bemeneti DNS-sel néhány nap alatt elvégezzük. Ha kevés a kiindulási anyag, a könyvtárkészítési módszerek e technológiai fejlesztései lehetővé tették a ChIP-seq-kísérleteket.
Az új technológia, amelyben a DNS-mintákat rövid szekvenciákkal, úgynevezett vonalkódokkal egyedileg megjelölik, ma már lehetővé teszi, hogy az egyes fragmentumokat egyetlen szekvenálási sávban egyesítsék a multiplex analízishez. Ez jelentősen növelte a DNS-szekvenálási kísérletek hatékonyságát és csökkentette a költségeket, ami tovább támogatja a ChIP-seq alkalmazásokat.
A DNS-szekvenálási technológia fejlődésének köszönhetően a ChIP-seq előnye, hogy nagyszámú ChIP-dúsított DNS-mintát lehet olcsón, viszonylag rövid idő alatt szekvenálni, a ChIP-chiphez képest nagyobb érzékenységgel és pontossággal.
Melyek a ChIP-vizsgálat különböző lépései?
A ChIP-próbák egy általános protokollt követnek:
- A fehérjék DNS-hez való keresztkötése csak az X-ChIP esetében
- Cellulízis
- Chromatin fragmentálása emésztéssel (X-ChIP és N-ChIP esetében) vagy szonikációval-alapú nyírással (csak X-ChIP esetében)
- Immunoprecipitáció specifikus antitestekkel
- DNS-tisztítás a downstream elemzésekhez
- DNS-analízis PCR segítségével, qPCR, microarray vagy NGS
Fontos, hogy a pozitív és negatív kontrollok minden egyes lépésnél szerves részét képezik annak meghatározásának, hogy a ChIP-kísérlet sikeres volt-e.
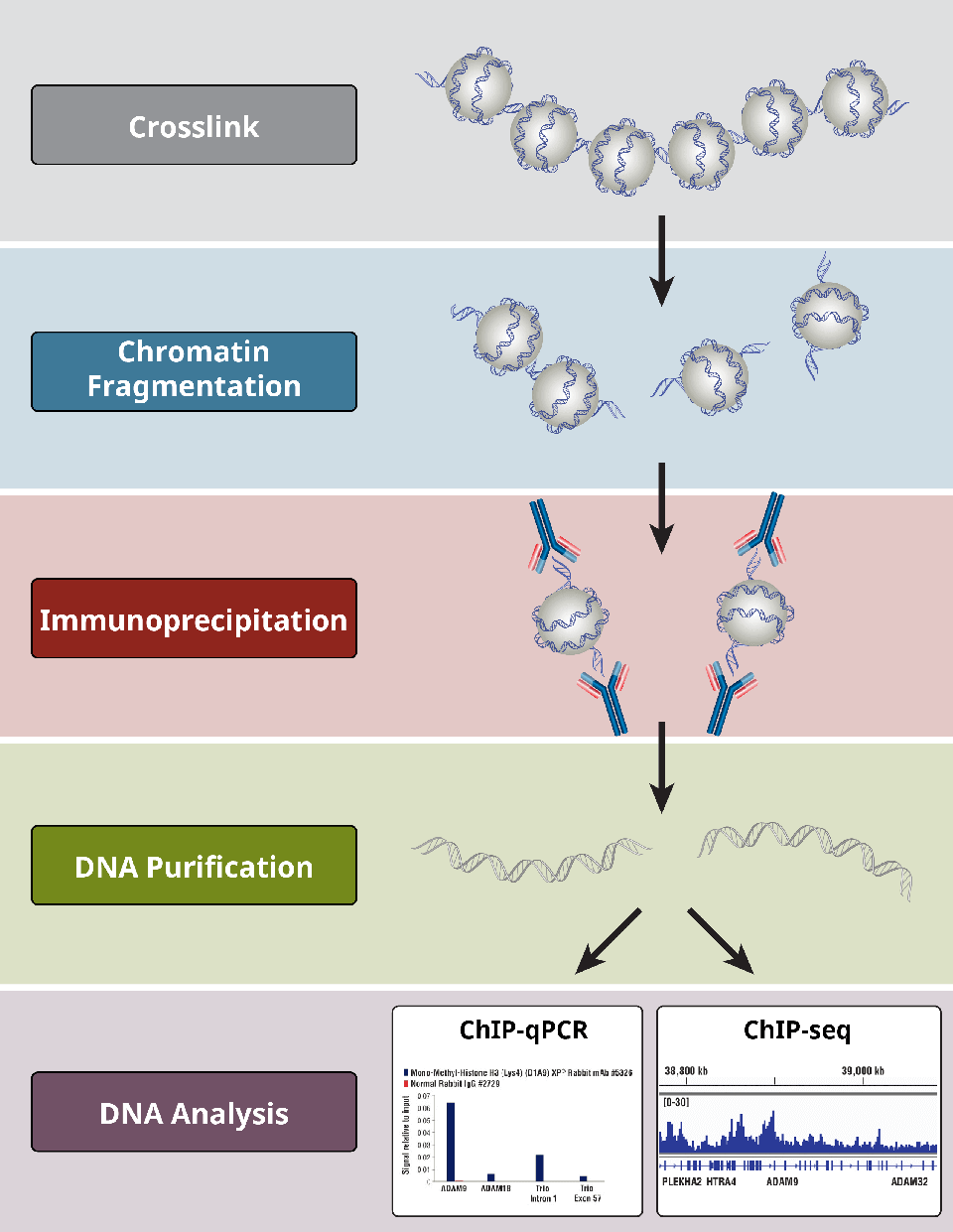
Áttekintés a ChIP protokoll legkritikusabb lépéseiről.
Áttekintés a ChIP protokoll legkritikusabb lépéseiről.
6.1 Hogyan történik a sejtek és szövetek keresztkötése ChIP-hez?
A keresztkötő reagenseket a fehérjék “rögzítésére” használják az általuk megkötött DNS-hez. Ennek a rögzítésnek az eléréséhez jellemzően formaldehid alapú reagenseket használnak. A sejteket és a szöveteket általában hasonló módon fixálják, de a szövetek hosszabb fixálási időt és gyorsabb fixálási szállítást igényelnek annak érdekében, hogy gyorsan átjárják a célszövetet, mielőtt az degenerálódni kezdene.
A kromatin túlzott fixálása csökkentheti a szonikációval történő fragmentálás hatékonyságát, amellett, hogy gátolja az antitestek kötődését a fehérje célpontjaikhoz. Ezért a fixálási időt empirikusan kell meghatározni annak érdekében, hogy az antitest-antigén kötődés maximális legyen, ugyanakkor a fehérjék ideális keresztkötése a cél-DNS-ükhöz megvalósuljon.
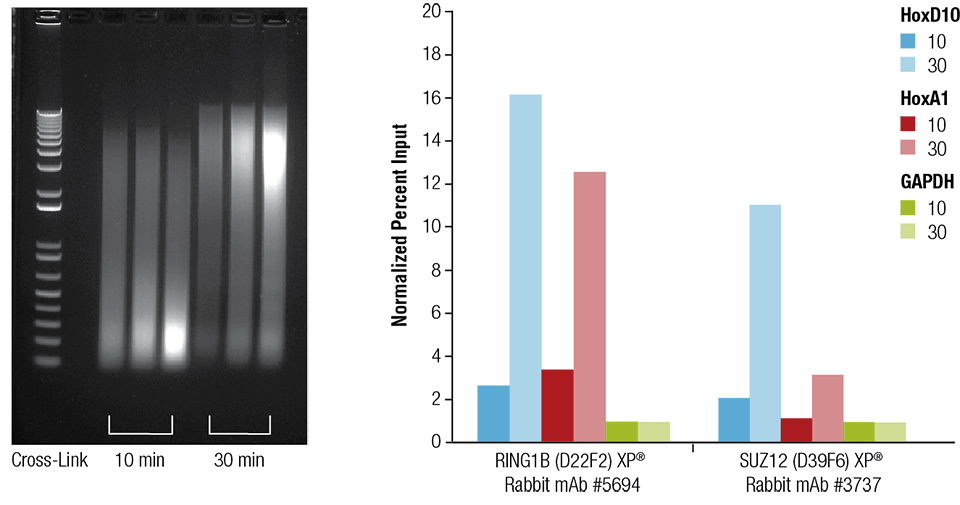
Egerek szívét (H), agyát (B) és máját (L) 10 vagy 30 percig keresztkötöttük a jelzett módon (bal oldali panel). A kromatint előkészítettük és 4 percig szonikáztuk. A szívszövetből előkészített kromatinnal ChIP-et végeztünk a jelzett antitestekkel a SimpleChIP® Plus Sonication Chromatin IP Kit #56383 segítségével, majd a feldúsított DNS-t valós idejű PCR segítségével számszerűsítettük a jelzett génekhez tartozó primerek segítségével (jobb oldali panel). Az immunprecipitált DNS mennyiségét az egyes mintákban a negatív lokuszhoz, a GAPDH-hoz képest normalizált jelként ábrázoltuk, amely egyenlő 1.
Egerek szívét (H), agyát (B) és máját (L) a jelzetteknek megfelelően 10 vagy 30 percig keresztkötéssel kezeltük (bal panel). A kromatint előkészítettük és 4 percig szonikáztuk. A szívszövetből előkészített kromatinnal ChIP-et végeztünk a jelzett antitestekkel a SimpleChIP® Plus Sonication Chromatin IP Kit #56383 segítségével, majd a feldúsított DNS-t valós idejű PCR segítségével számszerűsítettük a jelzett génekhez tartozó primerek segítségével (jobb oldali panel). Az immunprecipitált DNS mennyiségét az egyes mintákban a negatív lokusz GAPDH-hoz normalizált jelként ábrázoljuk, amely egyenlő 1.
6.2 Hogyan fragmentáljuk a kromatint?
A kromatin fragmentálása elengedhetetlen a ChIP-kísérlet sikeréhez. A kromatin fragmentálása szükséges a kromatin szolubilizálásához és lehetővé teszi annak koprecipitációját. Ezenkívül a ChIP-vizsgálat felbontása a kromatin fragmentációjától függ, mivel a DNS-fragmentum mérete határozza meg a ChIP-vizsgálat felbontását.
Az enzimatikus emésztés mikrokocka nukleázt (MNáz) használ, amely a nukleoszómák közötti kettősszálú DNS-t hasítja, hogy kromatinfragmentumokat hozzon létre. Míg a teljes MNáz-emésztés 150 bázispárnyi DNS-fragmentumokat (mono-nukleoszómákat), a nem teljes emésztés 150 és 750 bázispár közötti DNS-fragmentumokat (mono-, di- és tri-nukleoszómákat) hoz létre. A szonikáció mechanikai erőt alkalmaz a kromatin fragmentálására. A szonikáció a nukleoszómák között és a nukleoszómákon belül is fragmentálja a kromatint, 150 és 1000 bázispár közötti kromatinfragmentumokat generálva.
Az X-ChIP esetében vagy enzimatikus emésztést, vagy szonikációt alkalmazunk a kromatin nyírására. A szonikációs ChIP protokoll szonikációs körülményeit empirikusan kell meghatározni, mivel azok sejttípusonként és kísérleti feltételenként eltérőek. Az emésztési feltételek egységesebbek a különböző sejttípusok és szövetek esetében, de a kromatin fragmentum méretét még az IP előtt elemezni kell.
Az N-ChIP esetében nukleázokat használnak a kromatin fragmentálására, hogy a fehérjekötődést a nem fixált mintákban is fenntartsák. A nukleázok fragmentációját szintén empirikusan kell meghatározni a kromatin túlzott emésztésének minimalizálása érdekében.
6.3 Miért használjunk enzimatikus emésztést a ChIP-hez?
Nukleázos emésztést kell használni az N-ChIP-hez, mivel a fehérjék nem kötődnek keresztkötéssel a DNS-hez, és a szonikáción alapuló fragmentációhoz kapcsolódó kemény körülmények a kromatinfehérjék DNS-től való disszociációját eredményeznék. Az N-ChIP ideális a hisztonfehérje-DNS kölcsönhatások elemzésére, mivel a hiszton-DNS kötődés nagyon erős és stabil. Az N-ChIP azonban nem alkalmas a transzkripciós faktorok és kofaktorok kromatinkötésének elemzésére.
A kromatin fragmentálására az X-ChIP-ben vagy enzimatikus emésztés, vagy szonikáció használható. Az enzimatikus emésztés előnyei közé tartozik a fragmentáció konzisztenciája és az enyhe fragmentációs körülmények (alacsonyabb hő és detergens), amelyek jobban megőrzik a kromatin és az antitest epitópok integritását, ami a transzkripciós faktorokhoz és kofaktorokhoz kötött kromatin nagyobb mértékű immundúsítását eredményezi.
6.4 Miért használjunk szonikációt a kromatin ChIP-hez történő fragmentálására?
Az enzimatikus emésztéssel elért kromatin fragmentációval ellentétben a szonikáció mechanikai erőkre támaszkodik a kromatin kisebb darabokra történő fragmentálásához. Az immundúsításhoz ideális kromatinfragmentumok mérete 200 és 1000 bázispár között van. A szonikáció a hagyományos módszer a kromatin fragmentálására, és elvégezhető hagyományos szondaszonikátorral vagy fejlettebb vízfürdős szonikátorokkal, amelyek fókuszáltabb szonikációt biztosítanak. A szonikáció valóban randomizált kromatinfragmentumokat hoz létre; azonban széles körű optimalizálást igényel a különböző sejtvonalak és szövetek esetében, és nehezen reprodukálható kísérletről kísérletre. A magas detergens pufferek követelménye és a szonikáció során keletkező hő károsíthatja mind a kromatin, mind a kromatinfehérjék antitest epitópjainak integritását.
6.5 A kromatin szonikáció optimalizálása ChIP-hez
A szonikáción alapuló kromatinfragmentálás hagyományosan magas detergens puffereket használ és hőt termel, mindkettő károsíthatja a kromatin és az antitest epitópok integritását. Ezért a kromatin fragmentálásához használt szonikáció mennyiségét kísérletileg kell meghatározni a különböző sejtvonalak és szövetek esetében. Meg kell határozni és alkalmazni kell a 150-1000 bázispár DNS-fragmentumok létrehozásához szükséges minimális szonikációs mennyiséget a kromatin károsodásának minimalizálása érdekében.
A teljes ChIP-vizsgálat megkezdése előtt, amelyhez qPCR, DNS-chip vagy NGS általi downstream analízis kapcsolódik, gélelektroforézist kell alkalmazni a különböző ideig szonikált kromatinminták elemzésére. A fragmentum mérete függ a szonikációs időtől – a fragmentum mérete a szonikációs idő növekedésével csökken. Az adatok azonban arra utalnak, hogy a hosszabb szonikációs idő nem vezet jobb eredményekhez. Ezért a tisztított immunprecipitált DNS gélen történő futtatása és az ideális fragmensméret meghatározása egyszerű módszer a kívánt DNS-mérethez szükséges minimális szonikációs idő meghatározására és a kromatin felesleges károsodásának elkerülésére.

Az enzimmel emésztett kromatint agarózgélen futtattuk. Az 1. sáv az alulemésztett kromatint mutatja. A 2. sáv a megfelelően emésztett kromatint, a 3. sáv pedig a túlemésztett kromatint mutatja.
Az enzimekkel emésztett kromatint agarózgélen futtattuk. Az 1. sáv az alulemésztett kromatint mutatja. A 2. sáv a megfelelően emésztett kromatint, a 3. sáv pedig a túlemésztett kromatint mutatja.
6.6 Hogyan válasszunk antitestet a ChIP-hez?
A megfelelő antitest kiválasztása a ChIP-kísérlet sikeréhez elengedhetetlen. A ChIP-kísérletben használt antitesteknek specifikusnak kell lenniük az érdeklődésre számot tartó fehérjére, és nagy affinitással kell rendelkezniük az antigénhez. A ChIP- vagy ChIP-seq-kísérlethez a legjobb antitestválasztás a ChIP- vagy ChIP-seq-validált antitest. Ha a kívánt génhez nem áll rendelkezésre ChIP-validált antitest, a következő legjobb választás egy IP-ben validált antitest. Fontos megjegyezni, hogy nem minden IP-validált antitest működik ChIP-ben, és nem minden ChIP-validált antitest működik ChIP-seq-ben. Ezenkívül minél több antitestet validáltak más alkalmazásokban, mint például western, IP, IF, flow és IHC, annál nagyobb a bizalom az antitest teljesítménye és specificitása iránt. Az antitest validálását az antitest optimális koncentrációjának empirikus meghatározásának kell követnie, az IP mosási körülményekkel együtt.
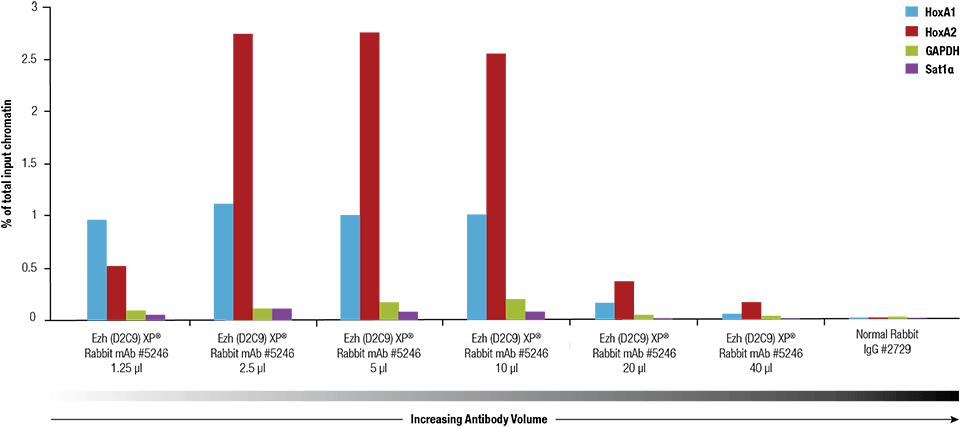
Ezh2 (D2C9) XP® Rabbit mAb #5246-ot a SimpleChIP® Plus Enzimatikus kromatin IP Kit #9005 segítségével titráltuk 4 x 106 NCCIT sejtből készített keresztkötésű kromatinon.
Ezh2 (D2C9) XP® Rabbit mAb #5246-ot a SimpleChIP® Plus Enzimatikus kromatin IP Kit #9005 segítségével titráltuk 4 x 106 NCCIT sejtből készített keresztkötésű kromatinon.
6.7 Az immunprecipitáció elvégzésének módja
Az antitesteket az érdeklődésre számot tartó fehérje és a hozzá kötött DNS rögzítésére használjuk. Az antitest-koncentrációt empirikusan kell meghatározni, általános kiindulási pontként 0,5-2,0 μg antitestet kell használni 10 μg kromatin-DNS-re (ez körülbelül 4 x 106 sejtnek felel meg). A pufferek szigorát és a mosási időt szintén empirikusan kell meghatározni, mivel ezek az antitestnek a célantigénhez való affinitásától függnek. Általában az antitest:kromatin inkubációkat 2 órától egy éjszakán át tartanak.
Az antitest-antigén (+DNS) komplexeket affinitással rögzítik egy antitestkötő gyantán. A ChIP-kísérletekben ez a gyanta jellemzően ChIP-minőségű mágneses, szefaróz vagy agaróz gyöngyökből áll, amelyek fehérje A és/vagy fehérje B konjugáltak. Az antitestek különböző affinitással kötődnek a fehérje A és/vagy fehérje G gyöngyökhöz attól függően, hogy milyen fajban fejlesztették ki őket és milyen IgG altípusú a nehézláncuk. A gyöngyöket általában 2-4 órán keresztül inkubálják az antitest:kromatinnal.
A nem antitesthez kötött kromatin eltávolításához mosási lépésekre van szükség, majd a keresztkötések visszafordítása (X-ChIP esetén) és a DNS tisztítása következik. Ezenkívül IgG kontroll IP-t kell végezni a háttér (jel:zaj) meghatározásához. Pozitív kontroll antitesteket (pl. teljes hiszton H3) és/vagy pozitív kontroll qPCR primereket (ismert pozitív és negatív célfehérje kötődési lókuszokhoz) is be kell vonni a nem specifikus kötődés meghatározásához. Az optimális eredmények érdekében a kromatin IP qPCR-rel történő QC-jét a downstream NGS-analízis előtt kell elvégezni.
6.8 Hogyan eluáljuk a kromatint a protein A/G gyöngyökből
A kromatint detergens és hő segítségével eluáljuk a protein A/G gyöngyökből. Alacsony sebességű “vortexelésre” vagy keverésre van szükség a gyöngyök szuszpenzióban tartásához és a kromatin elúciójának fokozásához.
6.9 Hogyan fordítsuk vissza a kromatin keresztkötéseit
A keresztkötéseket magas hővel és magas sóval fordítjuk vissza (mindkettő létfontosságú összetevő). Proteináz K-t is adunk a kapcsolódó kromatinfehérjék és a hozzáadott antitestek emésztéséhez, ami hatékonyabb downstream DNS-tisztítást tesz lehetővé.
6.10 Hogyan tisztítsuk a DNS-t
A kromatin keresztkötések eltávolítása után a DNS-t vagy klasszikus fenol-kloroformos, majd etanolos kicsapási módszerekkel vagy oszlopalapú DNS-tisztító készletekkel tisztítjuk.
Hogyan elemezzük a feldúsított DNS-t
A DNS tisztítása után számos downstream analízis végezhető, beleértve a ChIP-PCR, ChIP-qPCR, ChIP-chip és ChIP-seq elemzést.
7.1 ChIP-PCR és ChIP-qPCR analízis
A ChIP-PCR és ChIP-qPCR analízisek a legalkalmasabbak egyetlen gén elemzésére, és a DNS specifikus fragmentumainak gyors és költséghatékony módon történő felerősítésére és mennyiségi meghatározására használhatók.
7.2 ChIP-chip-analízis
A ChIP-chip-analízis csempézett DNS-mikroarray-chipeket használ a fehérjekötődések és fehérjemódosítások genomszintű, nagy felbontású térképének létrehozására.
7.3 ChIP-seq-elemzés
A ChIP-seq-elemzés a tisztított DNS és a korábban annotált teljes genomok összehangolására standard NGS-technológiát használ a genomszintű fehérjekötődési profilok azonosítására.