Genomic features of A. brassicae en twee andere co-infecterende A. alternata isolaten
We hebben de genomen gesequeneerd van twee isolaten van A. alternata (PN1 en PN2) die B. juncea co-infecteerden met A. brassicae. De A. brassicae assemblage is eerder beschreven. Kort samengevat bestond de assemblage uit negen volledige chromosomen en één chromosoom waarbij telomerische herhalingen aan een van de uiteinden ontbraken. Afgezien van deze chromosomen, waren er zes contigs waarvan een van hen was ~ 1 Mb groot, die samen een overbodig chromosoom vormen (Fig. 1). De N50 van de A. brassicae assemblage was 2,98 Mb (Tabel 1). De twee isolaten die B. juncea co-infecteerden werden geïdentificeerd als A. alternata op basis van hun ITS- en GAPDH-sequenties. De A. alternata assemblages Aat_PN1 en Aat_PN2 bestonden uit 14 contigs met een totaal van 33.77 Mb, en 15 contigs met een totaal van 33.53 Mb, respectievelijk (Tabel 1). Zes contigs in elk van de twee assemblages telomerische herhalingen aan beide uiteinden bevatten en daarom zijn het meest waarschijnlijk om volledige chromosomale moleculen vertegenwoordigen. Vier andere contigs in beide assemblies telomerische herhalingen aan een uiteinde, maar waren van vergelijkbare grootte van volledige chromosoom moleculen zoals beschreven in A. solani . Daarom vertegenwoordigden de genoomassemblages voor A. alternata isolaten tien bijna-complete chromosomen van elk van de twee isolaten.
Geheel-genoom alignments met verwante Alternaria spp. toonden een algehele syntenie tussen de genomen met kleine herschikkingen (Fig. 2). Daarnaast werden ook mitochondriale sequenties verkregen uit de sequencing data voor de twee isolaten van A. alternata. De mitochondriale genomen van de A. alternata stammen waren respectievelijk ongeveer 49.783 bp en 50.765 bp groot en vertoonden een grote overeenkomst met het eerder gepubliceerde mitochondriale genoom van A. alternata.
Genvoorspelling na herhaalde maskering resulteerde in de identificatie van 11593, 11495, en 11387 genen in respectievelijk de A. brassicae, A. alternata PN1, en PN2 genoom-assemblages. Dit was vergelijkbaar met de geschatte genaantallen in andere Alternaria spp. (Tabel 1). BUSCO analyse toonde aan dat de genmodellen voorspeld in de drie genomen 98% van de single copy geconserveerde fungale genen dekten, wat aangeeft dat de assemblages bijna volledig waren. De voorspelde genen werden uitvoerig geannoteerd met behulp van een combinatie van databases zoals beschreven in de Methodes sectie (Fig. 1). Naast de drie genomen, voorspelden we ook genen de novo in de genoom-assemblages van drie andere Alternaria soorten die waren gesequenced met behulp van long-read technologieën, namelijk A. brassicicola (abra43), A. alternata (ATCC34957), en A. solani (altNL03003) (Tabel 1). Deze zes genomen en hun genvoorspellingen werden gebruikt voor de vergelijkende analyses van secundaire metaboliet-coderende genclusters en effector-coderende genen.

Samenvatting van het genoom van A. brassicae-genoom, (van buiten naar binnen cirkelvormige sporen) a pseudochromosomen/scaffolds, b Eiwitcoderende genen, c Herhalingselementen, d Overdraagbare elementen (DNA en LTR), e Voorspelde clusters van secundaire metabolieten, f Afgescheiden eiwitten, g Voorspelde effectoren
Phylogenomische analyse wijst een aparte clade toe voor de Brassica-infecterende A. brassicae en A. brassicicola binnen het Alternaria genus
Om de divergentie en verwantschap tussen A. brassicae, de twee A. alternata isolaten (PN1 en PN2), en de andere Alternaria soorten nauwkeurig te reconstrueren, hebben we fylogenomische analyses uitgevoerd met behulp van 29 single copy orthologs die het hoogste fylogenetische signaal hadden zoals berekend door het programma Mirlo. Selectie van genen met hogere fylogenetische signalen leidt tot fylogenieën die meer congruent zijn met de soortenboom. De resulterende fylogenie toonde aan dat de groot-sporige Alternaria en de klein-sporige Alternaria soorten afzonderlijk geclusterd waren in twee verschillende clades (Fig. 3). Interessant is dat de twee belangrijkste pathogenen van de Brassica’s, namelijk A. brassicae en A. brassicicola, apart van alle andere Alternaria soorten geclusterd zijn, wat mogelijk duidt op een verschillend evolutietraject gebaseerd op de gemeenschappelijke gastheervoorkeuren van deze twee soorten.
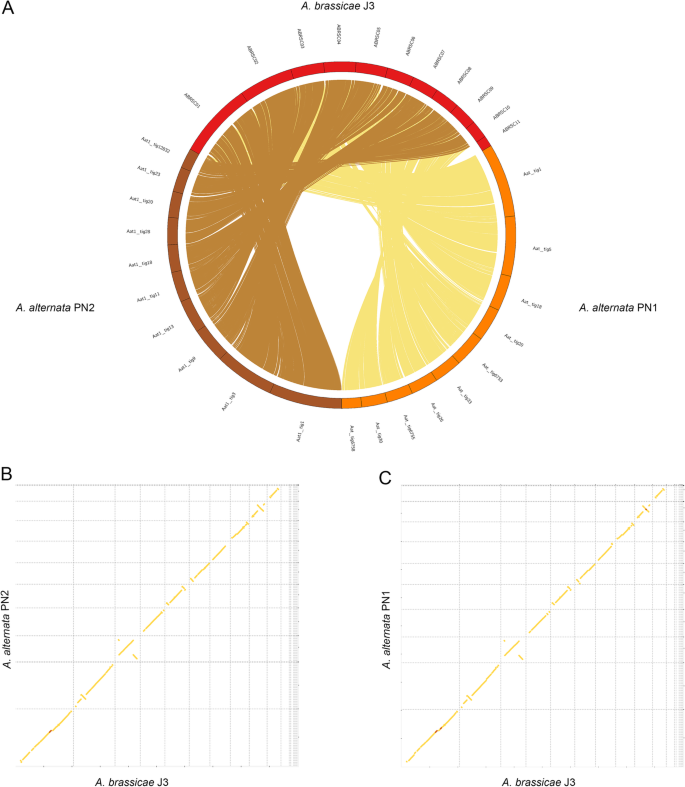
Whole-genome alignments van A. alternata PN1 en PN2 met A. brassicae. a Circos plot toont macrosyntenie van A. alternata PN1 en PN2 met A. brassicae over alle contigs behalve de dispensable contigs (ABRSC11, scaffold13,17,18,19), b en c Syntenic dotplots van A. brassicae met A. alternata PN1 en PN2
Vergelijkende analyses van A. alternata-isolaten verkregen uit verschillende gastheren
We hebben de genomen van A. alternata PN1 en PN2 (geïsoleerd uit B. juncea) vergeleken met die van A. alternata ATCC34957 (geïsoleerd uit sorghum) om eventuele verschillen in hun genoominhoud te identificeren, waardoor deze twee zeer verschillende soorten zouden kunnen infecteren. De volledige genoomuitlijningen van A. alternata PN1 en PN2 met die van A. alternata ATCC34957 onthulden zeer hoge niveaus van syntenie en de afwezigheid van enige soortspecifieke regio’s. We identificeerden 719, 152, en 586 isolaat-specifieke genen tussen de drie isolaten van A. alternata, respectievelijk (Additional file 1: Tabel S1). Meer dan tweederde van de isolaatspecifieke genen in de drie isolaten waren niet gekarakteriseerde eiwitten of hadden geen annotaties. Opmerkelijk is dat alle drie de isolaten geen onvervangbare chromosomen bevatten die pathogeniteit kunnen verschaffen, zoals gerapporteerd is voor A. alternata isolaten die veel fruitgewassen infecteren, zoals citrus, peer en appel. Het genenrepertoire van de drie isolaten bestond ook uit een vergelijkbaar aantal en type effectoren, CAZymes, en secundaire metabolietclusters (Tabel 2). Bovendien veroorzaken de twee isolaten PN1 en PN2 op zichzelf geen infectiesymptomen in B. juncea onder epifytotische omstandigheden (gegevens niet aangetoond). Onze resultaten suggereren dat deze isolaten van A. alternata (PN1 en PN2) facultatieve pathogenen kunnen zijn die een saprofytische levensstijl leiden en onder bepaalde omgevingsomstandigheden kunnen overgaan tot een pathogene levensstijl.
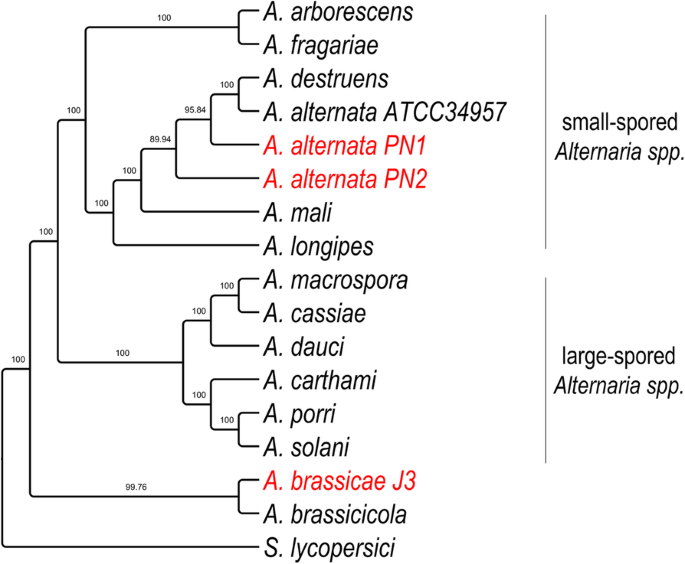
Phylogenetische boom van Alternaria-soorten met S. lycopersici als outgroup. De boom is geconstrueerd met behulp van 29 single copy orthologs, die het hoogste fylogenetische signaal hadden zoals berekend in Mirlo. Vertakkingsondersteuningswaarden van 1000 bootstrap-replicaten worden getoond
Een overvloed aan herhalingsrijke regio’s en transponeerbare elementen in A. brassicae
Filamenteuze plantpathogenen hebben de neiging om een aparte genoomarchitectuur te hebben met een hoger herhalingsgehalte. Schatting van het herhalingsgehalte en maskering met behulp van RepeatModeler en RepeatMasker toonden aan dat het genoom van A. brassicae bestond uit ~ 9,33% herhalingen, vergeleken met 2,43 en 2,64% herhalingen in de genomen van A. alternata. Het genoom van A. brassicae bevat het hoogste aantal herhalingen (~ 9,33%) van alle Alternaria soorten die tot nu toe zijn gesequenteerd. Onze analyse toonde aan dat het repeat gehalte significant verschilt tussen de A. alternata isolaten en de andere pathogene Alternaria soorten. De pathogene Alternaria soorten, met name A. brassicae en A. brassicicola, hadden een aanzienlijk groter repertoire van LTR/Gypsy en LTR/Copia elementen (> 8X) in vergelijking met de andere A. alternata isolaten (pathogeen en niet-pathogeen) (Fig. 4). De A. brassicae en A. brassicicola genomen hadden ook een oververtegenwoordiging van DNA transposons, die ~ 5% van het genoom bedroeg, in vergelijking met < 1% in de andere Alternaria soorten (Fig. 4).

Vergelijking van herhalingsgehalte in zes Alternaria soorten. De grootte van de bubbels komt overeen met het (a) percentage transponeerbare elementen (TEs) in het genoom, b kopiegetal van de TE in het genoom
De toename van repetitief DNA en de daaropvolgende evolutie van genen die deze regio’s overlappen, kunnen de sleutel zijn tot het evolutionaire succes waarin deze ziekteverwekkers erin geslaagd zijn te overleven gedurende generaties van co-evolutionair conflict met hun gastheren. De nabijheid van TE’s stelt de genen mogelijk bloot aan door herhaling geïnduceerde puntmutaties (RIP) en daardoor aan een versnelde evolutie. Ectopische recombinatie tussen gelijksoortige TE’s kan ook resulteren in nieuwe combinaties van genen en daardoor de diversiteit van eiwitten of metabolieten vergroten.
Aanwezigheid van een dispensabel chromosoom in de groot-sporige A. brassicae
Lineage-specifieke (LS) chromosomen of dispensabele chromosomen (DC) zijn gemeld bij verscheidene fytopathogene soorten, waaronder A. alternata. DC’s in A. alternata staan erom bekend virulentie en gastheerspecificiteit aan het isolaat te verlenen. De whole-genome alignments van A. brassicae met andere Alternaria spp. toonden aan dat een contig van ca. 1 Mb samen met andere kleinere contigs (66-366 kb) specifiek was voor A. brassicae en geen syntenie vertoonde met enige regio in de andere Alternaria spp. Echter, gedeeltelijke syntenie werd waargenomen wanneer de contig werd uitgelijnd met de sequenties van andere dispensable chromosomen gerapporteerd in Alternaria spp. . Dit bracht ons tot de hypothese dat deze contigs samen een DC van A. brassicae zouden kunnen vertegenwoordigen. Om dit te bevestigen hebben we de contigs doorzocht op de aanwezigheid van AaMSAS en ALT1 genen, welke bekende markergenen zijn voor dispensable chromosomen in Alternaria spp. Wij vonden twee kopieën van het AaMSAS-gen als onderdeel van twee secundaire metaboliet biosynthese clusters op de 1 Mb contig. We vonden echter geen homoloog van het ALT1-gen. Bovendien werd het herhalingsgehalte van de contigs (ABRSC11, scaffold 13, 17, 18, en 19) vergeleken met het volledige genoom. De gen inhoud van de lineage-specifieke contigs was aanzienlijk lager dan die van de kern chromosomen (tabel 3). Omgekeerd waren de DC contigs sterk verrijkt in TE-gehalte in vergelijking met de kernchromosomen (tabel 3).
Hoewel de DC niet verrijkt was met genen die coderen voor afgescheiden eiwitten, was het aandeel van afgescheiden effectorgenen 30% hoger in vergelijking met de kernchromosomen. Al het bovenstaande wijst erop dat A. brassicae inderdaad een DC kan herbergen. DC’s in Alternaria spp. zijn tot nu toe alleen gerapporteerd van de klein-sporige Alternaria spp. en van geen enkele groot-sporige Alternaria soort is bekend dat ze DC’s bevatten. Het valt nog te bezien of de DC bijdragen aan de virulentie van A. brassicae. Toekomstige studies zouden de karakterisering van het dispensable chromosoom in A. brassicae omvatten en de correlatie van zijn aanwezigheid met de pathogeniciteit van verschillende isolaten.
Orthologie-analyse onthult soortspecifieke genen met vermeende rollen in virulentie
Verschillen in geninhoud en diversiteit binnen genen dragen bij tot aanpassing, groei en pathogeniciteit. Om de verschillen in geninhoud binnen het Alternaria genus en de Dothideomyceten te catalogiseren, voerden we een orthologie-analyse uit op de gecombineerde set van 3.60.216 eiwitten van 30 verschillende soorten (waaronder 16 Alternaria soorten) die behoren tot de Dothideomyceten (Additional file 2: Tabel S2), waarvan 3.45.321 eiwitten konden worden toegewezen aan ten minste één van de orthogroepen. We identificeerden 460 A. brassicae-specifieke genen die aanwezig waren in A. brassicae maar afwezig in alle andere Alternaria soorten (Additional file 3: Tabel S3). Deze soortspecifieke genen omvatten 35 gesecreteerde eiwitcoderende genen waarvan 11 als effectoren werden voorspeld. Bovendien waren 20 van deze soortspecifieke genen aanwezig op de DC. Een groot aantal van deze eiwitten behoorde tot de categorie van ongekarakteriseerde eiwitten waarvan de functie niet bekend is. Om te testen of deze soortspecifieke genen het resultaat zijn van adaptieve evolutie die plaatsvindt in de herhaal-rijke gebieden van het genoom, voerden we een permutatietest uit om de overlap van herhaal-rijke gebieden en transposable elementen met een willekeurige genenset te vergelijken met de overlap van deze soortspecifieke genen. Wij vonden dat deze soortspecifieke genen significant overlapten met herhalingsrijke regio’s (P-waarde: 9,99e-05; Z-score: – 4,825) en transposabele elementen (P-waarde: 0,0460; Z-score: 2,539) in het genoom.
Secondary metabolite profile of A. brassicae and its association with transposable elements (TEs)
De geslachten Alternaria en Cochliobolus staan bekend als de belangrijkste producenten van gastheerspecifieke secundaire metaboliettoxinen. Vooral Alternaria spp. staan bekend om de productie van chemisch diverse secundaire metabolieten, die gastheerspecifieke toxinen (HST’s) en niet-HST’s omvatten. Deze secundaire metabolieten worden gewoonlijk gegenereerd door non-ribosomale peptide synthasen (NRPS) en polyketide synthasen (PKS). We identificeerden vijf NRPS type SM genclusters, 12 PKS type genclusters en zeven terpeen-achtige genclusters in A. brassicae (Additional file 4: Tabel S4). Van de vijf NRPS-clusters konden we drie clusters identificeren die bekende secundaire metabolieten produceren, namelijk destruxine B, HC-toxine en dimethylcoprogeen (siderofoor) met bekende rollen in virulentie.
De gencluster die verantwoordelijk is voor de productie van dimethylcoprogeen (siderofoor) in A. brassicae bestaat uit 22 genen, waaronder de belangrijkste biosynthesegenen, oxidoreductasen en siderofore-transporteurs. Sideroforen zijn ijzerchelaterende verbindingen die door schimmels worden gebruikt om extracellulair ijzer te verwerven en zijn naar verluidt betrokken bij de virulentie van schimmels. De identificatie van de gencluster verantwoordelijk voor de synthese van sideroforen zou de studie van sideroforen en hun rol in pathogeniciteit bij A. brassicae mogelijk maken. Daarnaast werd ook een cluster van het type PKS, bestaande uit 12 genen, verantwoordelijk voor de melanineproductie geïdentificeerd (Additional file 4: Tabel S4). De melanine biosynthese cluster is eerder beschreven voor A. alternata. Ook de transcriptiefactor Amr1, die de melanineproductie induceert, is gekarakteriseerd in A. brassicicola en staat erom bekend virulentie te onderdrukken. De rol van melanine in virulentie is echter onduidelijk en soortspecifiek.
De plantenpathogenen behorend tot het geslacht Alternaria lijken een dynamische capaciteit te hebben om nieuw secundair metabolietpotentieel te verwerven om nieuwe ecologische niches te koloniseren. De meest voor de hand liggende verklaring voor deze dynamische verwerving van secundair metabolietpotentieel is horizontale genoverdracht binnen het genus Alternaria en mogelijk met andere geslachten. Er is uitgebreid bewijsmateriaal in de literatuur dat veel van de HSTs van Alternaria op de afneembare chromosomen gedragen worden en uitwisseling van deze chromosomen kan de gastheerspecificiteit verbreden. We identificeerden ook een NRPS cluster, mogelijk coderend voor HC-toxine in een van de DCs (scaffold 18) (Additional file 4: Table S4). HC-toxine is een bekende virulentie determinant van de plant pathogeen Cochliobolus carbonum, die maïs genotypes infecteert die een functionele kopie van HM1 missen, een carbonyl reductase dat het toxine ontgift. Een recent rapport toonde aan dat A. jesenskae ook HC-toxine kan produceren, waardoor het de enige andere schimmel is dan C. carbonum die het toxine produceert. De aanwezigheid van een HC-toxine gencluster, een virulentie determinant bij C. carbonum, in een DC van A. brassicae wijst erop dat interspecies horizontale genoverdracht vaker kan voorkomen dan verwacht.
Naast horizontale genoverdracht kunnen snelle duplicatie, divergentie en verlies van de SM genen er ook toe bijdragen dat de ziekteverwekker nieuwe metabolische capaciteiten ontwikkelt. Deze processen van duplicatie en divergentie kunnen worden bevorderd door de nabijheid van de secundaire metabolietclusters bij de repeat-elementen, waardoor ze vatbaar zijn voor RIP-mutaties. Daarom hebben we getest of de secundaire metabolietclusters ook geassocieerd waren met repeat-rijke regio’s. Een permutatietest werd gebruikt om de overlap van repeat-rijke regio’s met een willekeurige genenset te vergelijken met de overlap van secundaire metabolietclustergenen. De secundaire metabolietclusters overlapten de herhaalrijke regio’s significant in vergelijking met de willekeurige genenset (P-waarde: 0,0017; Z-score: – 2,7963). Ook overlapten deze clusters significant met transposable elementen onder de herhaalbare-rijke regio’s (P-waarde: 0.0087; Z-score: 2.9871). Dit toont aan dat beide hierboven beschreven mechanismen voor de verwerving van nieuw secundair metabolietpotentieel mogelijk zijn in het geval van A. brassicae. Populatie-analyses op soort- en genusniveau kunnen licht werpen op de prevalentie van deze mechanismen binnen het Alternaria genus.
Synteny-analyse onthult de genetische basis van de exclusiviteit van Destruxine B productie door A. brassicae binnen het Alternaria genus
Destruxine B vertegenwoordigt een klasse van cyclische depsipeptiden waarvan bekend is dat het een van de belangrijkste pathogeniciteitsfactoren van A. brassicae is en waarvan gerapporteerd is dat het een gastheerspecifiek toxine van A. brassicae is. Destruxine B is niet gerapporteerd voor productie door andere Alternaria soorten. Hier rapporteren we voor de eerste keer de biosynthetische genclusters die verantwoordelijk zijn voor de productie van Destruxine B in A. brassicae. De cluster bestaat uit 10 genen, waaronder het belangrijkste biosynthetische enzym dat wordt gecodeerd door een NRPS-gen (DtxS1) en het snelheidsbeperkende enzym, DtxS3 (aldo-keto reductase) (Additional file 4: Tabel S4). Interessant is dat synteny-analyse van deze cluster onder de zes Alternaria soorten aantoonde dat deze beide genen in geen van de andere Alternaria spp. aanwezig waren, hoewel de algemene synteny van de cluster in al deze soorten gehandhaafd bleef (Fig. 5). De afwezigheid van de sleutelgenen die coderen voor de enzymen DtxS1 en DtxS3 in de Destruxine B-cluster in de andere Alternaria-soorten verklaart de afwezigheid van Destruxine B in deze soorten.

Syntenie van de Destruxine B-cluster in de zes Alternaria-soorten. DtxS1 en DtxS3 zijn rood gemarkeerd (gelabeld met de respectieve gen-ID’s) en zijn afwezig in alle andere Alternaria-soorten
Destruxine B is gemeld van het entomopathogeen Metarhizium robertsii , en Ophiosphaerella herpotricha , de veroorzaker van voorjaarsdooie vlek van bermudagras, afgezien van A. brassicae. Wang et al. beschreven de secundaire metaboliet biosynthese cluster die verantwoordelijk is voor Destruxin productie in M. robertsii . De Destruxine-cluster in de Metarhizhium-soorten bestaat uit twee bijkomende sleutelgenen, namelijk DtxS2 en DtxS4. DtxS2 is verantwoordelijk voor de omzetting van Destruxine B in Destruxine A, C, D en E. We konden in de cluster geen syntenisch gen vinden dat overeenkomt met DtxS2 of DtxS4 in A. brassicae. Van A. brassicae is bekend dat het alleen Destruxine B produceert en niet de andere vormen van Destruxine, wat mogelijk de afwezigheid van DtxS2 in A. brassicae verklaart. DtxS4 is een aspartaat-decarboxylase dat nodig is voor de productie van β-alanine uit asparaginezuur en dat essentieel is voor de productie van de ruggengraatstructuur van Destruxine. Bovendien waren deletiemutanten van DtxS4 in staat om Destruxine te produceren wanneer zij werden aangevuld met β-alanine in het medium. Hoewel DtxS4 niet aanwezig is in de Destruxine cluster, heeft A. brassicae één functionele kopie van aspartaat decarboxylase elders in het genoom en kan daardoor mogelijk zorgen voor de aanvoer van β-alanine voor de synthese van Destruxine B. De evolutionaire geschiedenis van de Destruxine B productie binnen het genus Metarhizhium lijkt sterk op die van Alternaria. De gespecialiseerde pathogenen zoals M. acridum missen de twee sleutelenzymen en produceren dus geen Destruxinen. M. robertsii, een generalist met een breder gastheerbereik produceert echter wel Destruxinen. Daarom werd de hypothese geformuleerd dat Destruxinen verantwoordelijk zouden kunnen zijn voor het grotere gastheerbereik van M. roberstii. Een soortgelijke hypothese zou ook kunnen gelden voor A. brassicae, die een breed gastheerbereik heeft en bijna alle Brassicaceae kan infecteren. Verdere experimenten om het gastheerbereik van Destruxin null mutanten van A. brassicae te bepalen, kunnen helpen om de rol van dit belangrijke fytotoxine in de uitbreiding van het gastheerbereik van A. brassicae te verduidelijken.
Distinctieve CAZyme profielen van A. brassicae en A. brassicicola binnen het Alternaria genus
CAZymen (Carbohydrate-Active enZymes) zijn proteïnen die betrokken zijn bij de afbraak, herschikking of synthese van glycosidebindingen. Plantpathogenen scheiden een hele reeks CAZymes af die de complexe polysacchariden in de plantencelwand afbreken. Zij bestaan uit vijf verschillende klassen, namelijk glycosidehydrolasen (GH), glycosyltransferasen (GT), polysaccharidelyasen (PL), koolhydraatesterasen (CE) en koolhydraatbindende modules (CBM). We identificeerden > 500 CAZymen in de zes Alternaria spp. inclusief A. brassicae (Additional file 5: Tabel S5). De CAZyme verdeling van A. brassicae en A. brassicicola verschilde van die van de andere Alternaria spp. en vormde zo een aparte cluster (Fig. 6). Het aantal enzymen met hulpactiviteit of de enzymen die betrokken zijn bij de afbraak van de plantencelwand, varieerde aanzienlijk tussen de verschillende vergeleken soorten. Bijna 46% van de CAZymen in A. brassicae werden gesecreteerd, waarvan ~ 17% voorspeld werd effectoren te zijn.

Heatmap van de CAZyme-profielen in 16 soorten met Alternaria-geslacht. De clustering van de organismen komt sterk overeen met die van de soortenfylogenie, behalve A. brassicae en A. brassicicola, die apart clusteren met de outgroup Stemphylium lycopersici. De CAZymen werden gegroepeerd volgens de CAZy classificatie
De AA9 familie (voorheen GH61; koper-afhankelijke lytische polysaccharide monooxygenases (LPMO)) is significant vergroot in vergelijking met de andere CAZy families in het Alternaria genus met elke soort die > 20 kopieën van het gen bevat. De kopie-aantallen in de Alternaria spp. zijn veel hoger dan de kopie-aantallen gerapporteerd voor Botrytis en Fusarium spp. . De AA9-familie is betrokken bij de afbraak van polysachariden in de celwand en staat bekend om zijn werking op een reeks polysachariden, waaronder zetmeel, xyloglucan, cellodextrinen en glucomannaan. Er wordt verondersteld dat LPMO’s een tweeledige rol hebben – rechtstreeks de polysacchariden van de celwand afbreken, en optreden als ROS-generator en aldus bijdragen tot de oxidatieve stress die leidt tot necrose in de plantenweefsels. Opvallend is dat van 11 van de 26 AA9 eiwitten aanwezig in A. brassicae voorspeld wordt dat het gesecreteerde effectoren zijn. De karakterisering van deze CAZymen en hun rol in de pathogenese zou het onderwerp kunnen zijn van verdere studies.
Overlappende effector profielen binnen het Alternaria genus
Wij voorspelden het effector repertoire van zes Alternaria soorten (Tabel 2) waaronder A. brassicae met behulp van de pijplijn beschreven in de methodes sectie. De meeste kleine gesecreteerde eiwitten die als effector werden voorspeld, waren niet gekarakteriseerde/geprediceerde eiwitten en kunnen dus nieuwe soort- of genus-specifieke effectoren zijn. A. brassicae had het grootste aandeel onbekende/voorspelde/hypothetische eiwitten in de reeks kandidaat-effectoren. Wij vonden dat de meeste effectoren tussen de zes soorten gemeenschappelijk waren en elkaar overlapten, wat suggereert dat de brede mechanismen van pathogenese waarbij eiwiteffectoren betrokken zijn, binnen het genus geconserveerd kunnen zijn. We vonden echter dat twee kopieën van een beta/gamma-kristalline vouw-bevattend eiwit alleen aanwezig zijn in A. brassicae en A. brassicicola en niet in de andere vier Alternaria soorten. Een verdere zoekactie in de nr database van NCBI bracht aan het licht dat dit eiwit volledig afwezig was in het Alternaria genus en dat de dichtstbijzijnde overeenkomsten werden gevonden in andere plantpathogenen, namelijk Macrophomina en Fusarium soorten. Er is echter geen informatie beschikbaar over zijn functie of rol in pathogeniciteit in een van de soorten.
We konden ook vaststellen dat sommige van de effectoren in A. brassicae het potentieel hebben om adaptief te evolueren, omdat ze ook significant geassocieerd waren met de herhaal-rijke regio’s van het genoom (P-waarde: 0,0003; Z-score: – 2,8778). Analyses op populatieniveau zijn daarom nodig om de effectoren te identificeren die onder positieve selectie staan, wat licht zou kunnen werpen op de evolutie van pathogeniciteit in A. brassicae. De in deze studie geïdentificeerde effectoren onthullen de brede waaier van eiwitten die betrokken kunnen zijn bij de pathogenese van A. brassicae. Van 39 van deze effectoren werd voorspeld dat het CAZymes zijn die een verschillende rol spelen bij de afbraak van de celwand en de geassocieerde polysacchariden. Het genoom van A. brassicae bevat twee necrose- en ethyleen-inducerende peptide-eiwitten, die betrokken zijn bij de pathogenese van diverse ziekteverwekkers, waaronder oomyceten en necrotrofen. Van Hrip1 (Hypersensitive response inducing protein 1) van A. alternata is onlangs aangetoond dat het door MdNLR16 wordt herkend op een klassieke gen-voor-gen manier, en deletie van Hrip1 van A. alternata verhoogt de virulentie . Een homoloog van Hrip1 is ook aanwezig in A. brassicae, maar er wordt niet voorspeld dat het buiten de cel wordt gesecreteerd, hoewel dit experimenteel moet worden geverifieerd. De aanwezigheid van effectoren die op een gen-voor-gen manier worden herkend, opent de mogelijkheid om complementaire R-genen in de gastheer te identificeren die kunnen worden gebruikt om resistente variëteiten of cultivars te ontwikkelen.