Genomowe cechy A. brassicae i dwóch innych współinfekujących izolatów A. alternata
Sekwencjonowaliśmy genomy dwóch izolatów A. alternata (PN1 i PN2), które współinfekowały B. juncea z A. brassicae. Asocjacja A. brassicae została wcześniej opisana. W skrócie, zespół ten składał się z dziewięciu kompletnych chromosomów i jednego chromosomu z brakiem powtórzeń telomerowych na jednym z końców. Poza tymi chromosomami znajdowało się sześć kontigów, z których jeden miał rozmiar ~ 1 Mb, co razem może stanowić chromosom zbędny (Ryc. 1). N50 zespołu A. brassicae wynosiło 2,98 Mb (Tabela 1). Dwa izolaty współzakażające B. juncea zidentyfikowano jako A. alternata na podstawie ich sekwencji ITS i GAPDH. Asocjacje A. alternata Aat_PN1 i Aat_PN2 składały się z 14 kontigów o łącznej długości 33,77 Mb i 15 kontigów o łącznej długości 33,53 Mb, odpowiednio (Tabela 1). Sześć kontigów w każdym z tych dwóch złożeń zawierało powtórzenia telomeryczne na obu końcach i dlatego najprawdopodobniej reprezentują pełne cząsteczki chromosomalne. Cztery inne kontigi w obu asemblacjach zawierały powtórzenia telomeryczne na jednym końcu, ale były podobnej wielkości jak pełne cząsteczki chromosomów, co opisano u A. solani. W związku z tym, genomy izolatów A. alternata reprezentowały dziesięć prawie kompletnych chromosomów każdego z dwóch izolatów.
Porównania całych genomów z pokrewnymi Alternaria spp. wykazały ogólną syntezę pomiędzy genomami z niewielkimi rearanżacjami (Rys. 2). Dodatkowo uzyskano również sekwencje mitochondrialne z danych sekwencjonowania dla dwóch izolatów A. alternata. Genomy mitochondrialne szczepów A. alternata miały wielkość odpowiednio 49 783 bp i 50 765 bp i wykazywały wysokie podobieństwo do wcześniej opublikowanego genomu mitochondrialnego A. alternata .
Przewidywanie genów w wyniku maskowania powtórzeń doprowadziło do identyfikacji 11593, 11495 i 11387 genów odpowiednio w genomach A. brassicae, A. alternata PN1 i PN2. Była to liczba porównywalna z liczbą genów oszacowaną u innych gatunków Alternaria spp. (Tabela 1). Analiza BUSCO wykazała, że modele genów przewidywane w trzech genomach obejmowały 98% jednokopijnych, konserwatywnych genów grzybów, co wskazuje na bliską kompletności asemblację. Przewidywane geny zostały kompleksowo zanotowane przy użyciu kombinacji baz danych, jak opisano w rozdziale Metody (Rys. 1). Oprócz tych trzech genomów, przewidzieliśmy również geny de novo w zespołach genomów trzech innych gatunków Alternaria, które zostały zsekwencjonowane przy użyciu technologii long-read, a mianowicie A. brassicicola (abra43), A. alternata (ATCC34957) i A. solani (altNL03003) (Tabela 1). Te sześć genomów i ich predykcje genów zostały wykorzystane do analiz porównawczych klastrów genów kodujących metabolity wtórne i genów kodujących efektory.

Summary of A. brassicae genome, (Od zewnętrznych do wewnętrznych ścieżek kolistych) a pseudochromosomy/scaffolds, b geny kodujące białka, c elementy powtarzalne, d elementy transpozycyjne (DNA i LTR), e przewidywane skupiska metabolitów wtórnych, f białka wydzielane, g przewidywane efektory
Analiza filogenomiczna przypisuje osobny klad dla zakażających Brassica A. brassicae i A. brassicicola w obrębie rodzaju Alternaria
W celu dokładnej rekonstrukcji dywergencji i relacji pomiędzy A. brassicae, dwoma izolatami A. alternata (PN1 i PN2) oraz innymi gatunkami Alternaria, przeprowadziliśmy analizy filogenomiczne z wykorzystaniem 29 ortologów o pojedynczej kopii, które miały najwyższy sygnał filogenetyczny obliczony przez program Mirlo. Wybór genów o wyższym sygnale filogenetycznym prowadzi do filogenez, które są bardziej zgodne z drzewem gatunkowym. Otrzymana filogeneza wykazała, że gatunki Alternaria o dużych i małych zarodnikach grupują się oddzielnie w dwa różne klady (Rys. 3). Co ciekawe, dwa główne patogeny kapustnych viz. A. brassicae i A. brassicicola grupowały się oddzielnie od wszystkich innych gatunków Alternaria, prawdopodobnie wskazując na inną trajektorię ewolucyjną opartą na wspólnych preferencjach żywicielskich tych dwóch gatunków.
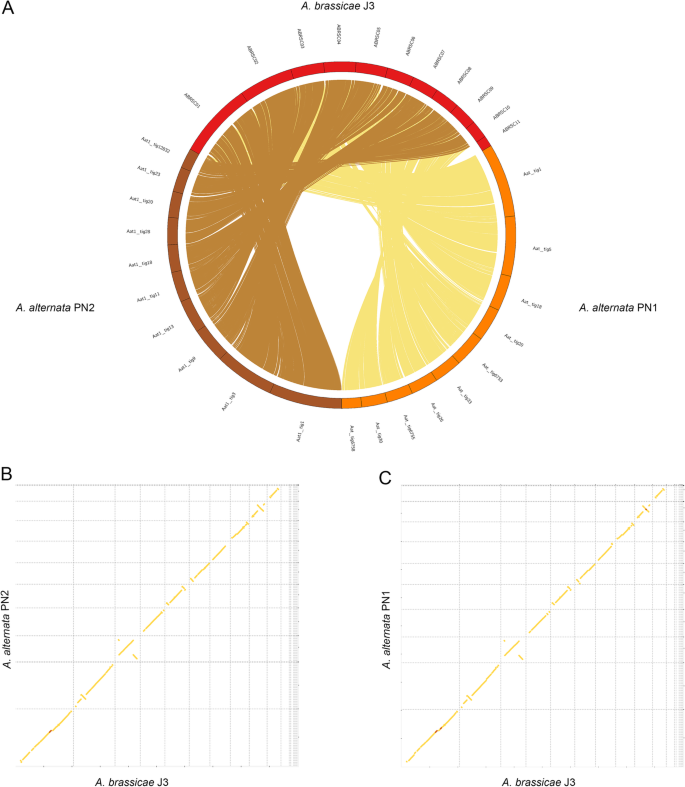
Whole-genome alignments of A. alternata PN1 and PN2 with A. brassicae. a Circos plot showing macrosynteny of A. alternata PN1 and PN2 with A. brassicae we wszystkich kontigach z wyjątkiem kontigów dyspensowalnych (ABRSC11, scaffold13,17,18,19), b i c Syntenic dotplots of A. brassicae with A. alternata PN1 and PN2
Comparative analyses of A. alternata uzyskanych z różnych gospodarzy
Porównaliśmy genomy A. alternata PN1 i PN2 (wyizolowanych z B. juncea) z genomem A. alternata ATCC34957 (wyizolowanego z sorgo) w celu zidentyfikowania wszelkich różnic w ich zawartości genomowej, które mogłyby umożliwić im infekowanie dwóch bardzo różnych gatunków. Alignacje całych genomów A. alternata PN1 i PN2 do A. alternata ATCC34957 ujawniły bardzo wysoki poziom syntenii i brak jakichkolwiek regionów specyficznych dla gatunku. Zidentyfikowaliśmy odpowiednio 719, 152 i 586 genów specyficznych dla poszczególnych izolatów A. alternata (Dodatkowy plik 1: Tabela S1). Ponad dwie trzecie genów specyficznych dla izolatów we wszystkich trzech izolatach było białkami niescharakteryzowanymi lub nie posiadającymi adnotacji. Na uwagę zasługuje fakt, że wszystkie trzy izolaty nie zawierały żadnych zbędnych chromosomów, które mogłyby nadawać patogeniczność, jak to miało miejsce w przypadku izolatów A. alternata infekujących wiele upraw owocowych, takich jak cytrusy, gruszki i jabłka. Repertuar genów trzech izolatów zawierał również podobną liczbę i rodzaj efektorów, CAZymów i klastrów metabolitów wtórnych (tab. 2). Dodatkowo, dwa izolaty PN1 i PN2 nie wywołują samodzielnie objawów infekcji u B. juncea w warunkach epifitycznych (dane nie pokazane). Nasze wyniki sugerują, że te izolaty A. alternata (PN1 i PN2) mogą być fakultatywnymi patogenami, które prowadzą saprofityczny tryb życia i mogą przejść na patogeniczny tryb życia w pewnych warunkach środowiskowych.
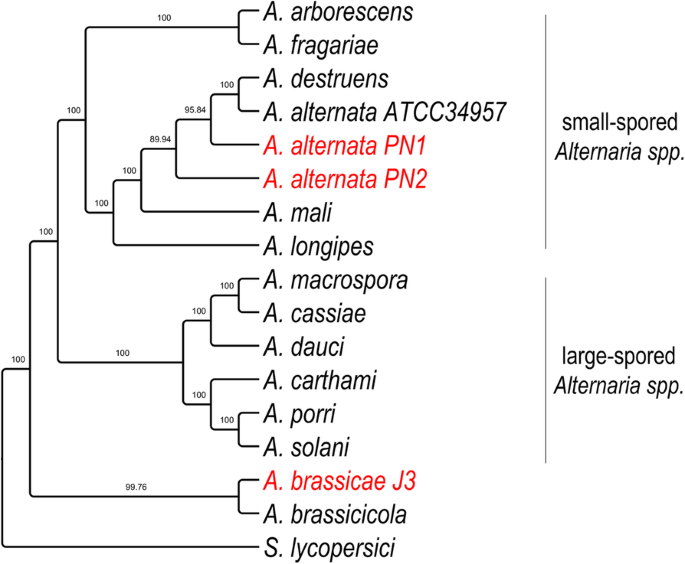
Drzewo filogenetyczne gatunków Alternaria z S. lycopersici jako outgrupą. Drzewo zostało skonstruowane przy użyciu 29 jednokopijnych ortologów, które miały najwyższy sygnał filogenetyczny obliczony w Mirlo. Wartości wsparcia gałęzi z 1000 powtórzeń bootstrapowych są pokazane
Obfitość regionów bogatych w powtórzenia i elementy transpozycyjne u A. brassicae
Patogeny roślin włóknistych mają tendencję do posiadania odrębnej architektury genomu z większą zawartością powtórzeń. Oszacowanie zawartości powtórzeń i maskowanie przy użyciu RepeatModeler i RepeatMasker ujawniło, że genom A. brassicae składa się z ~ 9,33% powtórzeń w porównaniu do 2,43 i 2,64% powtórzeń w genomach A. alternata. Genom A. brassicae zawiera najwięcej powtórzeń (~ 9,33%) spośród wszystkich gatunków Alternaria zsekwencjonowanych do tej pory. Nasza analiza wykazała, że zawartość powtórzeń różni się znacząco pomiędzy izolatami A. alternata a innymi patogennymi gatunkami Alternaria. Patogenne gatunki Alternaria, zwłaszcza A. brassicae i A. brassicicola, charakteryzowały się znacznie większym repertuarem elementów LTR/Gypsy i LTR/Copia (> 8X) w porównaniu do pozostałych izolatów A. alternata (patogennych i niepatogennych) (ryc. 4). Genomy A. brassicae i A. brassicicola charakteryzowały się również nadreprezentacją transpozonów DNA, które stanowiły ~ 5% genomu, w porównaniu do < 1% u pozostałych gatunków Alternaria (Ryc. 4).

Porównanie zawartości powtórzeń u sześciu gatunków Alternaria. Rozmiar bąbelków odpowiada (a) procentowi elementów transpozycyjnych (TE) w genomie, (b) liczbie kopii TE w genomie
Ta proliferacja powtarzalnego DNA i późniejsza ewolucja genów nakładających się na te regiony może być kluczem do sukcesu ewolucyjnego, w którym patogeny te zdołały przetrwać przez pokolenia współewolucyjnego konfliktu ze swoimi gospodarzami. Bliskość TE potencjalnie naraża geny na mutacje punktowe indukowane powtórzeniami (RIP), a tym samym na przyspieszoną ewolucję. Ektopowa rekombinacja między podobnymi TE może również skutkować nowymi kombinacjami genów, a tym samym zwiększać różnorodność białek lub metabolitów.
Występowanie chromosomu dyspensowalnego u wielkoskorupowego A. brassicae
Chromosomy specyficzne dla danej linii (LS) lub chromosomy dyspensowalne (DC) zostały zgłoszone przez kilka gatunków fitopatogenicznych, w tym A. alternata. DC u A. alternata są znane z nadawania izolatowi wirulencji i specyficzności w stosunku do gospodarza. Wyrównanie całego genomu A. brassicae z innymi Alternaria spp. wykazało, że kontig o wielkości ok. 1 Mb wraz z innymi mniejszymi kontigami (66-366 kb) był specyficzny dla A. brassicae i nie wykazywał syntezy z żadnym regionem u innych Alternaria spp. Jednakże, częściowa synteza została zaobserwowana, gdy kontig ten został wyrównany do sekwencji innych zbędnych chromosomów występujących u Alternaria spp. To skłoniło nas do wysunięcia hipotezy, że te kontigi razem mogą reprezentować DC A. brassicae. Aby to potwierdzić, przeszukaliśmy kontigi pod kątem obecności genów AaMSAS i ALT1, które są znanymi genami markerowymi dla chromosomów dyspensowalnych u Alternaria spp. Znaleźliśmy dwie kopie genu AaMSAS jako część dwóch klastrów biosyntezy metabolitów wtórnych na kontigu 1 Mb. Nie znaleźliśmy natomiast żadnego homologa genu ALT1. Dodatkowo, zawartość powtórzeń w kontigach (ABRSC11, rusztowania 13, 17, 18 i 19) została porównana z całym genomem. Zawartość genów w kontigach specyficznych dla poszczególnych linii była znacząco niższa niż w chromosomach rdzeniowych (Tabela 3). Natomiast kontigi DC były silnie wzbogacone w zawartość TE w porównaniu z chromosomami rdzeniowymi (Tabela 3).
Chociaż DC nie był wzbogacony w geny kodujące białka wydzielane, to udział wydzielanych genów efektorowych był o 30% wyższy w porównaniu z chromosomami rdzeniowymi. Wszystkie powyższe dowody wskazują na to, że A. brassicae może rzeczywiście posiadać DC. DC u Alternaria spp. zostały dotychczas opisane jedynie w przypadku Alternaria spp. o małych porach, natomiast nie stwierdzono występowania DC u Alternaria spp. o dużych porach. Nie wiadomo jeszcze, czy DC przyczyniają się do wirulencji A. brassicae. Przyszłe badania obejmą charakterystykę chromosomu dyspensowalnego u A. brassicae i korelację jego obecności z patogenicznością różnych izolatów.
Analiza ortologiczna ujawnia specyficzne gatunkowo geny z przypuszczalną rolą w wirulencji
Różnice w zawartości genów i różnorodności w obrębie genów przyczyniają się do adaptacji, wzrostu i patogeniczności. W celu skatalogowania różnic w zawartości genów w obrębie rodzaju Alternaria i Dothideomycetes, przeprowadziliśmy analizę ortologiczną na połączonym zestawie 3,60,216 białek z 30 różnych gatunków (w tym 16 gatunków Alternaria) należących do Dothideomycetes (plik dodatkowy 2: Tabela S2), z których 3,45,321 białek można było przypisać do co najmniej jednej z ortogrup. Zidentyfikowaliśmy 460 genów specyficznych dla A. brassicae, które były obecne u A. brassicae, ale nieobecne u wszystkich innych gatunków Alternaria (Dodatkowy plik 3: Tabela S3). Wśród tych gatunkowo specyficznych genów znalazło się 35 genów kodujących białka wydzielane, z których 11 uznano za efektorowe. Dodatkowo, 20 z tych gatunkowo specyficznych genów było obecnych na DC. Duża liczba tych białek należała do kategorii białek niescharakteryzowanych, których funkcja nie jest znana. W celu sprawdzenia, czy te specyficzne gatunkowo geny są wynikiem ewolucji adaptacyjnej zachodzącej w bogatych w powtórzenia regionach genomu, przeprowadziliśmy test permutacji, aby porównać nakładanie się regionów bogatych w powtórzenia i elementów transpozycyjnych z losowym zestawem genów z nakładaniem się tych specyficznych gatunkowo genów. Stwierdziliśmy, że te specyficzne gatunkowo geny nakładały się znacząco z regionami bogatymi w powtórzenia (P-value: 9.99e-05; Z-score: – 4.825) i elementami transpozycyjnymi (P-value: 0.0460; Z-score: 2.539) w genomie.
Secondary metabolite profile of A. brassicae and its association with transposable elements (TEs)
Rodzaje Alternaria i Cochliobolus znane są jako główni producenci specyficznych dla gospodarza toksyn z metabolitów wtórnych. Alternaria spp. są szczególnie znane z produkcji chemicznie zróżnicowanych metabolitów wtórnych, które obejmują toksyny specyficzne dla gospodarza (HST) i nie-HST. Te wtórne metabolity są zwykle generowane przez nierybosomalne syntazy peptydowe (NRPS) i syntazy poliketydowe (PKS). W A. brassicae zidentyfikowaliśmy pięć klastrów genów SM typu NRPS, 12 klastrów genów typu PKS i siedem klastrów genów terpenopodobnych (plik dodatkowy 4: Tabela S4). Spośród pięciu klastrów NRPS udało się zidentyfikować trzy klastry, które produkują znane metabolity wtórne, takie jak destruktyna B, toksyna HC i dimetylkoprogen (siderofor) o znanej roli w wirulencji.
Klaster genów odpowiedzialny za produkcję dimetylkoprogenu (sideroforu) u A. brassicae składa się z 22 genów, w tym głównych genów biosyntezy, oksydoreduktaz i transporterów sideroforu. Siderofory są związkami chelatującymi żelazo, wykorzystywanymi przez grzyby do pozyskiwania zewnątrzkomórkowego żelaza żelazowego i, jak się okazuje, są zaangażowane w wirulencję grzybów. Identyfikacja klastra genów odpowiedzialnych za syntezę sideroforów umożliwiłaby badania nad sideroforami i ich rolą w patogenności A. brassicae. Dodatkowo zidentyfikowano również klaster typu PKS składający się z 12 genów, odpowiedzialny za produkcję melaniny (plik dodatkowy 4: Tabela S4). Klaster biosyntezy melaniny został już wcześniej opisany dla A. alternata. Również czynnik transkrypcyjny Amr1, który indukuje produkcję melaniny, został scharakteryzowany u A. brassicicola i jest znany z tego, że hamuje wirulencję. Jednakże, rola melaniny w wirulencji jest niejednoznaczna i specyficzna dla danego gatunku.
Patogeny roślinne należące do rodzaju Alternaria wydają się mieć dynamiczną zdolność do nabywania potencjału nowych metabolitów wtórnych w celu kolonizacji nowych nisz ekologicznych. Najbardziej prawdopodobnym wyjaśnieniem dla tego dynamicznego nabywania potencjału metabolitów wtórnych jest horyzontalny transfer genów w obrębie rodzaju Alternaria i ewentualnie z innymi rodzajami. W literaturze można znaleźć wiele dowodów na to, że znaczna część HST Alternaria przenoszona jest na chromosomach dyspensowalnych, a wymiana tych chromosomów może poszerzać specyficzność gospodarza. Zidentyfikowaliśmy również klaster NRPS, prawdopodobnie kodujący HC-toksynę w jednym z DC (scaffold 18) (plik dodatkowy 4: Tabela S4). HC-toksyna jest znanym determinantem wirulencji patogenu roślinnego Cochliobolus carbonum, który infekuje genotypy kukurydzy pozbawione funkcjonalnej kopii HM1, reduktazy karbonylowej, która detoksykuje toksynę. Ostatnie doniesienie wykazało, że A. jesenskae również może produkować HC-toksynę, co czyni go jedynym grzybem innym niż C. carbonum, który produkuje tę toksynę. Obecność klastra genów HC-toksyny, determinanty wirulencji u C. carbonum, w DC A. brassicae wskazuje na fakt, że międzygatunkowy horyzontalny transfer genów może być bardziej powszechny niż się spodziewano.
Oprócz horyzontalnego transferu genów, szybka duplikacja, dywergencja i utrata genów SM może również przyczynić się do rozwoju nowych zdolności metabolicznych patogenu. Te procesy duplikacji i dywergencji mogą być wspomagane przez bliskość klastrów metabolitów wtórnych do elementów powtarzających, co czyni je podatnymi na mutacje RIP. Dlatego sprawdziliśmy, czy klastry metabolitów wtórnych są również związane z regionami bogatymi w powtórzenia. Test permutacji został użyty do porównania nakładania się regionów bogatych w powtórzenia z losowym zestawem genów z nakładaniem się genów z klastrów metabolitów wtórnych. Klastry metabolitów wtórnych znacząco nakładały się na regiony bogate w powtórzenia w porównaniu z losowym zestawem genów (P-value: 0.0017; Z-score: – 2.7963). Ponadto, klastry te nakładały się istotnie na elementy transpozycyjne w regionach bogatych w powtórzenia (P-value: 0.0087; Z-score: 2.9871). Wskazuje to, że oba opisane powyżej mechanizmy nabywania potencjału nowych metabolitów wtórnych mogą być możliwe w przypadku A. brassicae. Analizy w skali populacyjnej na poziomie gatunku i rodzaju mogą rzucić światło na powszechność tych mechanizmów w obrębie rodzaju Alternaria.
Analiza synteniczna ujawnia genetyczne podstawy wyłączności produkcji Destruksyny B przez A. brassicae w obrębie rodzaju Alternaria
Destruksyna B reprezentuje klasę cyklicznych depsipeptydów, o której wiadomo, że jest jednym z kluczowych czynników patogenności A. brassicae i została uznana za specyficzną dla gospodarza toksynę A. brassicae. Destruksyna B nie była dotychczas produkowana przez żaden z pozostałych gatunków Alternaria. Po raz pierwszy opisujemy klastry genów biosyntezy odpowiedzialne za produkcję Destruksyny B u A. brassicae. Klaster składa się z 10 genów, w tym głównego enzymu biosyntetycznego kodowanego przez gen NRPS (DtxS1) oraz enzymu ograniczającego tempo produkcji, DtxS3 (reduktaza aldo-keto) (plik dodatkowy 4: Tabela S4). Co ciekawe, analiza syntenii tego klastra wśród sześciu gatunków Alternaria wykazała, że oba te geny nie były obecne u żadnego z pozostałych gatunków Alternaria spp. chociaż ogólna syntenia klastra była zachowana u wszystkich tych gatunków (Rys. 5). Brak kluczowych genów kodujących enzymy DtxS1 i DtxS3 w klastrze Destruksyny B u pozostałych gatunków Alternaria tłumaczy brak Destruksyny B u tych gatunków.

Synteza klastra Destruksyny B u sześciu gatunków Alternaria. DtxS1 i DtxS3 są zaznaczone na czerwono (oznaczone odpowiednimi identyfikatorami genów) i są nieobecne u wszystkich pozostałych gatunków Alternaria
Destruksyna B została opisana z entomopatogenu Metarhizium robertsii , i Ophiosphaerella herpotricha , przyczyny wiosennej martwej plamistości trawy bermudagrass, oprócz A. brassicae. Wang i wsp., opisali klaster biosyntezy metabolitów wtórnych odpowiedzialny za produkcję Destruxin u M. robertsii . Klaster Destruxin u gatunków Metarhizhium składa się z dwóch dodatkowych kluczowych genów DtxS2 i DtxS4. DtxS2 jest odpowiedzialny za przekształcanie Destruksyny B w Destruksynę A, C, D i E. Nie udało nam się znaleźć żadnego syntenicznego genu w klastrze, który odpowiadałby DtxS2 lub DtxS4 u A. brassicae. Wiadomo, że A. brassicae produkuje tylko Destruksynę B, a nie inne formy Destruksyny, co może tłumaczyć brak DtxS2 u A. brassicae. DtxS4 jest dekarboksylazą asparaginianową, która jest wymagana do produkcji β-alaniny z kwasu asparaginowego i jest niezbędna do produkcji struktury szkieletowej Destruksyny. Co więcej, mutanty delecyjne DtxS4 były w stanie produkować Destruksynę, gdy do pożywki dodawano β-alaninę. Nawet jeśli DtxS4 nie jest obecny w klastrze Destruxin, A. brassicae posiada jedną funkcjonalną kopię dekarboksylazy asparaginianowej obecnej w innym miejscu genomu i dlatego może prawdopodobnie zapewnić dostawę β-alaniny do syntezy Destruxin B. Historia ewolucyjna produkcji Destruxin B w obrębie rodzaju Metarhizhium ściśle przypominała historię Alternaria. Specjalistyczne patogeny takie jak M. acridum nie posiadają dwóch kluczowych enzymów i dlatego nie produkują Destruxin. Natomiast M. robertsii, generalista o szerszym zakresie żywicieli, wytwarza Destruksyny. Postawiono więc hipotezę, że Destruksyny mogą być odpowiedzialne za powstanie rozszerzonego zakresu żywiciela M. roberstii. Podobna hipoteza może być również prawdziwa w przypadku A. brassicae, który ma szeroki zakres żywicieli i może infekować prawie wszystkie rośliny Brassicaceae. Dalsze doświadczenia mające na celu określenie zakresu żywicielskiego mutantów zerowych Destruksyny A. brassicae mogą pomóc w wyjaśnieniu roli tej ważnej fitotoksyny w rozszerzeniu zakresu żywicielskiego A. brassicae.
Odmienne profile CAZymów A. brassicae i A. brassicicola w obrębie rodzaju Alternaria
CAZymy (Carbohydrate-Active enZymes) są białkami zaangażowanymi w degradację, rearanżację lub syntezę wiązań glikozydowych. Patogeny roślinne wydzielają zróżnicowaną gamę CAZymes, które rozkładają złożone polisacharydy w ścianie komórkowej roślin. Składają się one z pięciu odrębnych klas: hydrolaz glikozydowych (GH), glikozylotransferaz (GT), liaz polisacharydowych (PL), esteraz węglowodanowych (CE) i modułów wiążących węglowodany (CBM). Zidentyfikowaliśmy > 500 CAZymów w sześciu gatunkach Alternaria spp. w tym A. brassicae (plik dodatkowy 5: Tabela S5). Rozmieszczenie CAZymów u A. brassicae i A. brassicicola różniło się od tych u pozostałych Alternaria spp. tworząc oddzielny klaster (Rys. 6). Liczba enzymów o aktywności pomocniczej lub enzymów biorących udział w degradacji ściany komórkowej roślin była bardzo zróżnicowana pomiędzy porównywanymi gatunkami. Blisko 46% CAZymów u A. brassicae było wydzielanych, z czego ~ 17% było przewidywanych jako efektory.

Heatmap of the CAZyme profiles in 16 species with Alternaria genus. Skupienie organizmów ściśle odpowiada filogenezie gatunkowej z wyjątkiem A. brassicae i A. brassicicola, które grupują się oddzielnie z outgrupą Stemphylium lycopersici. CAZymy zostały pogrupowane zgodnie z klasyfikacją CAZy
Rodzina AA9 (dawniej GH61; miedziozależne lityczne monooksygenazy polisacharydowe (LPMO)) jest znacznie powiększona w porównaniu z innymi rodzinami CAZy w rodzaju Alternaria, przy czym każdy gatunek zawiera > 20 kopii genu. Liczba kopii w Alternaria spp. jest znacznie wyższa niż liczba kopii odnotowana dla Botrytis i Fusarium spp. Rodzina AA9 jest zaangażowana w degradację polisacharydów ściany komórkowej i wiadomo, że działa na szereg polisacharydów, w tym skrobię, ksyloglukan, cellodekstryny i glukomannan. Postawiono hipotezę, że LPMO pełnią podwójną rolę – bezpośrednio rozszczepiają polisacharydy ściany komórkowej oraz działają jako generator ROS, przyczyniając się w ten sposób do stresu oksydacyjnego prowadzącego do nekrozy tkanek roślinnych. Uderzające jest to, że 11 z 26 białek AA9 występujących u A. brassicae to efektory wydzielane. Charakterystyka tych CAZymes i ich rola w patogenezie może być przedmiotem dalszych badań.
Pokrywające się profile efektorów w obrębie rodzaju Alternaria
Przewidzieliśmy repertuar efektorów sześciu gatunków Alternaria (Tabela 2), w tym A. brassicae, używając potoku opisanego w sekcji Metody. Większość małych białek wydzielanych, które przewidywano jako efektory, było białkami niescharakteryzowanymi/przewidywanymi, a zatem mogą to być nowe efektory specyficzne dla gatunku lub rodzaju. A. brassicae miała największy udział nieznanych/przewidywanych/hipotetycznych białek w zestawie kandydujących efektorów. Stwierdziliśmy, że większość efektorów pomiędzy sześcioma gatunkami jest wspólna i pokrywa się, co sugeruje, że szerokie mechanizmy patogenezy z udziałem efektorów białkowych mogą być konserwowane w obrębie rodzaju. Stwierdziliśmy jednak, że dwie kopie białka zawierającego fałdy beta/gamma-krystaliny są obecne tylko u A. brassicae i A. brassicicola, a nie u pozostałych czterech gatunków Alternaria. Dalsze wyszukiwanie w bazie danych nr NCBI ujawniło, że białko to było całkowicie nieobecne w rodzaju Alternaria, a najbliższe dopasowania znaleziono w innych patogenach roślinnych viz. Macrophomina i Fusarium. Nie ma jednak dostępnych informacji na temat jego funkcji lub roli w patogenności u któregokolwiek z tych gatunków.
Mogliśmy również ustalić, że niektóre z efektorów u A. brassicae mają potencjał do ewolucji adaptacyjnej, ponieważ były one również znacząco związane z regionami genomu bogatymi w powtórzenia (P-value: 0.0003; Z-score: – 2.8778). Konieczne są zatem analizy na poziomie populacji w celu zidentyfikowania efektorów podlegających selekcji pozytywnej, co mogłoby rzucić światło na ewolucję patogeniczności A. brassicae. Efektory zidentyfikowane w tym badaniu ujawniają szeroki zakres białek, które mogą być zaangażowane w patogenezę A. brassicae. Przewidywano, że 39 z tych efektorów to CAZymy pełniące różne funkcje w degradacji ściany komórkowej i związanych z nią polisacharydów. Genom A. brassicae zawierał dwa białka peptydowe indukujące nekrozę i etylen, które były zaangażowane w patogenezę różnych patogenów, w tym oomycetes i nekrotrofów. Ostatnio wykazano, że Hrip1 (Hypersensitive response inducing protein 1) z A. alternata jest rozpoznawane przez MdNLR16 w klasyczny sposób gen za gen, a delecja Hrip1 z A. alternata zwiększa jej wirulencję. Homolog Hrip1 jest również obecny u A. brassicae, ale nie przewiduje się, że jest on wydzielany poza komórkę, choć wymaga to weryfikacji doświadczalnej. Obecność efektorów, które są rozpoznawane w sposób gen za gen, otwiera możliwość identyfikacji komplementarnych genów R u gospodarza, które mogą być wykorzystane do opracowania odpornych odmian lub kultywarów.