Chaperony molekularne
Chaperony molekularne obejmują szeroki zakres białek odgrywających kluczową rolę w homeostazie komórkowej i są odpowiedzialne za pomoc w składaniu białek, montażu białek multimerycznych, translokacji białek w obrębie i przez komórkę, degradacji niepożądanych lub źle złożonych białek podczas normalnych procesów komórkowych i stabilizacji białek poprzez zapobieganie agregacji i pomoc w ponownym składaniu w warunkach stresu (Lindquist, 1986; Lindquist i Craig, 1988).
Białka zgłoszone do posiadania aktywności chaperonowej zostały początkowo odkryte jako te, które uległy nadekspresji podczas szoku cieplnego i stąd zostały nazwane białkami szoku cieplnego (Hsp). Oprócz szoku cieplnego, inne warunki stresu, takie jak węgiel, azot lub fosforan ograniczające warunki były również znane do indukowania chaperonów molekularnych. Białka te s± klasyfikowane w zależno¶ci od ich masy cz±steczkowej do pięciu głównych rodzin: (a) rodzina Hsp100, (b) rodzina Hsp90, (c) rodzina Hsp70, (d) rodzina Hsp60 oraz (e) rodzina małych białek szoku cieplnego (sHsp) (Bohen i in., 1995; Schirmer i in., 1996; Bukau i Horwich, 1998). Chaperony są również klasyfikowane na podstawie ich sposobu działania na: (a) fałdazy, chaperony, które wspomagają ponowne składanie nieuformowanych białek przy użyciu ATP, np. Hsp70 i Hsp60, (b) hołotazy, chaperony, które wiążą intermediaty fałdowania i zapobiegają agregacji, np. sHsp i Hsp40, oraz (c) dezagregazy, chaperony, które aktywnie dezagregują szkodliwe agregaty białkowe, co może prowadzić do powstania ich małych fragmentów, np. członkowie nadrodziny AAA + ATPase i Hsp100. Ten typ klasyfikacji jest prawdziwy z nielicznymi wyjątkami (Richter i in., 2010; Kim i in., 2013). Znaczna część naszego zrozumienia mechanizmów fałdowania białek wspomaganych przez chaperony pochodzi z prac nad rodzinami chaperonów Hsp60 i Hsp70. Niniejszy przegląd koncentruje się na klasie Hsp60 chaperonów molekularnych, zwracając uwagę na Hsp60 o nietypowej strukturze i funkcji.
Rodzina Hsp60/Chaperoniny
Chaperony o masie 60 kDa tworzą duże pierścienie oligomeryczne i są również określane jako chaperoniny. Chaperoniny mogą być dalej podzielone na dwie grupy na podstawie wymogu współchaperonin i ich lokalizacji komórkowej. Chaperoniny typu I występują w cytoplazmie prokariotów oraz w mitochondrium i chloroplaście eukariotów. Wymagaj± one udziału ko-chaperoniny Hsp10, która pełni rolę czapeczki na pierścieniu. Dobrze zbadana chaperonina typu I znana jest jako system GroEL-GroES w Escherichia coli. Jej homologami s± Cpn60/Cpn20 w chloroplastach oraz mtHsp60/mtHsp10 w mitochondrium (Cheng i in., 1989; Hayer-Hartl i in., 1995; Dickson i in., 2000). Chaperoniny typu II występują w cytoplazmie eukariontów oraz u mikroorganizmów archaebakteryjnych. Posiadają one wbudowaną pokrywę i dlatego do swojej funkcji nie wymagają współchaperonin (Ranson i in., 1998). Przykładem chaperonin typu II jest m.in. eukariotyczna maszyneria TriC/CCT (TCP-1 ring complex/chaperonin containing TCP-1 complex), składająca się z 8 podjednostek oraz termosom u archaebakterii. W przeciwieństwie do chaperonin typu I, niezależne od substratu wychwytywanie chaperonin typu II wymaga udziału homologów prefoldiny i Hsp70 (Iizuka i in., 2004; Cuéllar i in., 2008). Ostatnio opisano trzecią grupę znaną jako chaperoniny typu III, które są strukturalnie podobne do chaperonin typu II, ale mechanistycznie i filogenetycznie różnią się zarówno od chaperonin typu I, jak i typu II, np. chaperonina Carboxydothermus hydrogenoformans (Ch-CPN) (Techtmann i Robb, 2010; An i wsp., 2017; Rysunek 1). Chaperoniny typu I, II i III znane są również jako chaperoniny grupy I, II i III.
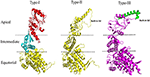
Rysunek 1. Cechy strukturalne chaperonin Typu I, Typu II i Typu III. Porównawcza analiza struktury chaperonin typu I, typu II i typu III. Struktury pobrano z RCSB z kodami PDB odpowiednio: 1AON, 3RUW i 5X9U. Chaperonina typu I podzielona jest na domeny apikalną, pośrednią i równikową, których analogiczne regiony w chaperoninach typu II i III zaznaczono liniami przerywanymi. Chaperonina typu II posiada charakterystyczn± wbudowan± w strukturę pokrywę, która pełni rolę ko-chaperoniny GroES chaperoniny typu I. Chaperoniny typu III są strukturalnie podobne do chaperonin typu II pod względem posiadania wbudowanego wieczka. Jednak sekwencja, struktura i funkcja lidu są odmienne w chaperoninach typu II i typu III (An i in., 2017). Do wygenerowania tego rysunku użyto programu PyMOL (PyMOL Molecular Graphics System, wersja 1.3).
Structure-Function of Type I Chaperonins: Prokaryotic Cytosol
E. coli GroEL-GroES
Strukturalne i funkcjonalne badania nad E. coli GroEL wykazały, że tworzy on strukturę tetradekameryczn± złożon± z dwóch heptamerycznych pierścieni ułożonych jeden na drugim tworz±cych wnękę, która zmienia swój charakter z przeważnie hydrofobowego na hydrofilowy po zwi±zaniu z GroES. Fałdowanie białek substratowych odbywa się w tej wnęce przy udziale ko-chaperoniny GroES, która jest heptameryczną strukturą przypominającą czapeczkę (Mande i in., 1996). Każdy monomer GroES ma wielkość 10 kDa. Monomer GroEL podzielony jest na trzy domeny: apikalną, pośrednią i równikową. Każdy monomer ma rozmiar ~57 kDa.
Istnieją dwa modele zaproponowane dla GroEL-GroES mediowanego składania białek substratowych. Model asymetryczny/sekwencyjny, który jest powszechnie akceptowany. W tym modelu GroEL i GroES s± obecne stechiometrycznie w stosunku 2:1 (stosunek podjednostek 14:7). W drugim modelu znanym jako model symetryczny/symultaniczny, który opiera się na ostatnio zaobserwowanym kompleksie GroEL-GroES, oba pierścienie GroEL są kapturowane przez ko-chaperoninę GroES w stosunku stechiometrycznym 1:1 tj. (GroEL-GroES)2, a stosunek podjednostek 14:14 (Sameshima i in., 2008; Ye i Lorimer, 2013; Fei i in., 2014). Symetryczny kompleks (GroEL-GroES)2 obserwowano zarówno w obecności, jak i przy braku białka substratowego, sugerując przejściowy stan pośredni w cyklu reakcji fałdowania.
Structure-Function of Type I Chaperonins: Endosymbiotic Organelles
Chloroplast and Mitochondrial Chaperonins
Chaperoniny chloroplastowe są typowo określane jako Cpn60 (homologi GroEL) i Cpn10 (homologi GroES). Chaperoniny Cpn60 składają się z wielu podjednostek, które dzielą się na dwa pokrewne, ale odrębne typy α i β (Dickson i in., 2000; Hill i Hemmingsen, 2001). W przeciwieństwie do chaperonin bakteryjnych, które zawierają wiele podjednostek i preferują homo-oligomeryzację (Ojha i in., 2005; Gould i in., 2007), chaperoniny chloroplastowe tworzą hetero-oligomery z dwoma typami podjednostek chaperonin α i β. Heterogenno¶ć występuje również w strukturze ko-chaperonin. Cpn10 jest podobna do standardowej ko-chaperoniny, tworząc heptameryczny pojedynczy pierścień podjednostek o masie 10 kDa (Koumoto i in., 2001; Sharkia i in., 2003). Cpn20 posiada dwie sekwencje polipeptydowe podobne do Cpn10 połączone w tandemie. Oczyszczony Cpn20 istnieje jako tetramer o strukturze pierścieniowej, zawierający podjednostkę 20 kDa. Jest on w pełni funkcjonalny in vitro, wspomagaj±c ponowne składanie zdenaturowanego białka w obecno¶ci zarówno chloroplastowego Cpn60, jak i E. coli GroEL (Tang i in., 2006). Ponadto, w Chlamydomonas reinhardtii Cpn10 wspomaga GroEL tylko w obecności Cpn20 (Tsai i in., 2012). Tak więc, znaczna heterogenność istnieje w oligomerycznym montażu chloroplastowych chaperonin.
Ludzka mitochondrialna chaperonina, mtHsp60 jest znana z mechanizmu składania białka (białko mitochondrialne) różniącego się od systemu GroEL-GroES i wymaga pojedynczego pierścienia heptamerycznego do wykonywania swojej funkcji składania białka wraz ze swoją ko-chaperoniną, mtHsp10 (Viitanen i in., 1992; Nielsen i Cowan, 1998). Jednakże struktura krystaliczna mitochondrialnej chaperoniny w kompleksie z jej ko-chaperoniną, mtHsp60-mtHsp10 przedstawia unikalny etap pośredni, w którym mtHsp60-mtHsp10 tworzy symetryczną dwupierścieniową strukturę przypominającą piłkę nożną, (mtHsp60)14 + 2 (mtHsp10)7.
Type I Chaperonins: Non-canonical Features
Multiple Chaperonins Across Species
Analizy całkowicie zsekwencjonowanych genomów sugerują, że około 30% wszystkich danych sekwencji genomowych posiada wiele kopii sekwencji genów kodujących chaperoniny (Lund, 2009; Kumar i in., 2015). Dystrybucja tych wielokrotnych chaperonin w oparciu o rozległą analizę filogenetyczną sugeruje, że wielokrotne kopie genów chaperonin występują głównie w pięciu filiach, a mianowicie (a) azylu Actinobacteria, (b) azylu Firmicutes, (c) azylu Cyanobacteria, (d) azylu Chlamydia i (e) azylu α-Proteobacteria (Kumar i in., 2015).
Actinobacteria
Actinobacteria są bakteriami Gram-dodatnimi i posiadają wysoką zawartość G + C w swoich genomach, np. Mycobacterium tuberculosis, Mycobacterium leprae i Bifidobacterium longum. Gatunki te posiadają zazwyczaj dwie kopie genów chaperonin, przy czym jedna z nich występuje w strukturze operonowej. Druga kopia Cpn60 istnieje jako niezależny gen, bez udziału genu Cpn10 (Rinke de Wit i in., 1992). Geny aktynobakteryjnych chaperonin s± pod regulacyjn± kontrol± czynnika transkrypcyjnego HrcA, który wi±że się z sekwencj± upstream CIRCE (control inverted repeat of chaperone expression) (Duchêne i in., 1994; Grandvalet i in., 1998). W niektórych przypadkach regulacja odbywa się poprzez wiązanie czynnika transkrypcyjnego HspR z sekwencją HAIR (HspR Associated Inverted Repeat) (Barreiro i in., 2004).
Firmicutes
Firmicutes są bakteriami Gram-dodatnimi i posiadają niską zawartość G + C w swoich genomach, np. Staphylococcus aureus, Desulfitobacterium dehalogenans i C. hydrogenoformans. Wiadomo, że bakterie Firmicutes posiadają zarówno prokariotyczne geny chaperonin typu I, jak i archaiczne geny chaperonin zaliczane do chaperonin typu III. Chaperoniny typu I s± ułożone operonicznie z ko-chaperonin±, natomiast gen chaperoniny typu III znajduje się w operonie dnaK. Zarówno geny chaperonin Typu 1 jak i Typu III są regulowane przez czynnik transkrypcyjny HrcA (Techtmann i Robb, 2010).
Chlamydiae
Chlamydiae są w większości obligatoryjnymi patogenami wewnątrzkomórkowymi, np. Chlamydia trachomatis, Chlamydia pneumonia i Chlamydia psittaci. Gatunki chlamydialne posiadają trzy kopie genów chaperonin (McNally i Fares, 2007). Układ operoniczny sugeruje, że istnieje tylko jedna kopia genów chaperoniny wraz z jej ko-chaperoniną. Jednak inne geny chaperonin s± zlokalizowane oddzielnie. Regulacja chlamydialnych genów chaperoninowych jest złożona. Pierwsza kopia genu chaperoniny jest indukowana przez szok termiczny i regulowana przez system HrcA-CIRCE. Druga kopia genu chaperoniny jest indukowana, gdy Chlamydia znajduj± się w monocytach lub makrofagach (Kol i in., 1999), a trzecia kopia genu chaperoniny jest indukowana, gdy Chlamydia znajduj± się w komórkach Hep-2 (Gérard i in., 2004). Takie rodzaje ekspresji i regulacji genów chaperonin sugerują specyficzne wzorce cyklu życiowego i niezależne role funkcjonalne dla nich.
α-proteobakterie
Rhizobia, należące do klasy α-proteobakterii, są organizmami symbiotycznymi żyjącymi w asocjacji z roślinami strączkowymi w guzkach korzeniowych i biorą udział w wiązaniu azotu, np. Bradyrhizobium japonicum, Rhizobium leguminosarum. Rhizobium zawierają największą liczbę kopii chaperonin. B. japonicum posiada siedem kopii genów chaperonin (Fischer i in., 1993). R. leguminosarum jest dobrze scharakteryzowanym organizmem i posiada trzy kopie genów chaperonin. Układ genów u wszystkich tych organizmów sugeruje, że trzy kopie genów chaperonin tworzą oddzielne operony z odpowiednimi genami ko-chaperonin (George i in., 2004). Jeden z operonów chaperoninowych zlokalizowany jest na wyspie genomowej, która zawiera geny zaangażowane w wiązanie azotu. Jest on regulowany przez czynniki NiF, które reguluj± geny wi±zania azotu (Ogawa i Long, 1995). Druga kopia genu chaperoniny nie jest dobrze zbadana i wiadomo, że jest zaangażowana w właściwości chaperonowe kilku modelowych białek substratowych (George i in., 2004).
Cyanobacteria
Cyanobacteria to w dużej mierze bakterie fotosyntetyzujące, np. Synechococcus platensis, Prochlorococcus marinus i Anabaena variabilis. Około 90% sekwencji genomowych gatunków cyjanobakterii zawiera dwie kopie genów chaperonin, przy czym jeden z nich jest ułożony w operon, podczas gdy drugi gen chaperonin jest kodowany oddzielnie. Niektóre gatunki sinic zawierają trzy kopie genów chaperonin, gdzie dwa z ich genów chaperonin są zlokalizowane z odpowiednimi ko-chaperoninami w operonie, podczas gdy trzecia kopia genów chaperonin jest niezależna (Lund, 2009; Kumar i in., 2015). Geny chaperonin występujące w układzie operonicznym ze swoimi ko-chaperoninami są genami niezbędnymi, natomiast te, które występują niezależnie od ko-chaperonin są genami nieistotnymi (Sato i in., 2008). Dwa geny chaperonin cyjanobakterii s± regulowane pozytywnie przez RpoH i negatywnie przez białka HrcA. Po szoku termicznym jeden z genów chaperonin jest gwałtownie indukowany, podczas gdy drugi gen chaperonin jest indukowany stopniowo (Kojima i Nakamoto, 2007; Rajaram i Apte, 2010). Wiadomo, że gen chaperoniny, który jest stopniowo indukowany na szoku termicznym, jest bezpośrednio zaangażowany w fotosyntezę.
Linia ewolucyjna
W miarę jak coraz więcej sekwencji genomowych staje się dostępnych, analiza genów chaperoniny sugeruje, że dystrybucja i częstotliwość występowania wielu kopii genów chaperoniny w obrębie filii i organizmów nadal wzrasta (Lund, 2009; Kumar i in., 2015). Aby zrozumieć, że przyczyną mnogości genów chaperonin jest albo horyzontalny transfer genów albo duplikacja genów, przeprowadzono analizę filogenetyczną białek GroEL w obrębie gatunków, która ujawniła, że przyczyny istnienia wielu kopii GroEL są niejednolite. W kilku przypadkach mamy do czynienia z duplikacj± genów, po której nast±piła selekcja ewolucyjna, jak to ma miejsce w przypadku myksobakteryjnych GroELs, mykobakteryjnych pierwszych i drugich kopii GroELs oraz kilku rizobakteryjnych GroELs. W przypadku trzeciego mykobakteryjnego homologu GroEL, kilku ryzobialnych GroEL i metanosarcynowych GroEL wystąpił horyzontalny transfer genów (Goyal i in., 2006; Kumar i in., 2015).
W naszym laboratorium zaproponowano wcześniej, że mykobakteryjny GroEL został zduplikowany i przeszedł różne presje selektywne, aby pełnić charakterystyczną rolę strukturalną i funkcjonalną w trakcie ewolucji (Goyal i in., 2006). Badania biofizyczne i biochemiczne rekombinowanych GroEL M. tuberculosis wykazały, że GroEL1 i GroEL2 istnieją jako niższe oligomeryczne gatunki, w przeciwieństwie do tetradecamerycznej struktury GroEL E. coli (Qamra i in., 2004). Struktura krystaliczna M. tuberculosis GroEL2 w jej dimerycznej formie podkreśliła obecność odrębnych reszt w regionie interfejsu, prawdopodobnie odpowiedzialnych za zmianę oligomeryzacji (Rysunek 2; Qamra i Mande, 2004). Badania nad zamianą genów i domen w M. tuberculosis GroEL1 sugerują, że domena równikowa jest odpowiedzialna za nieudaną oligomeryzację. Domena wierzchołkowa może wytrzymać duże insercje i delecje (Kumar i in., 2009). Mniej więcej w tym samym czasie wykazano, że GroEL1 ewoluował, by wiązać kwasy nukleinowe (Basu i in., 2009), a oligomeryzacja jest ułatwiona przez fosforylację reszt serynowych (Kumar i in., 2009). Ponieważ wiadomo, że GroEL2 jest niezbędną chaperoniną w Mycobacteria, podczas gdy oligomeryczne złożenie GroEL1 jest regulowane post-translacyjnie, doniesiono, że tetradecameryczne złożenie i precyzyjna komunikacja między domenami są warunkiem wstępnym dla aktywności chaperonin (Chilukoti i wsp., 2015).
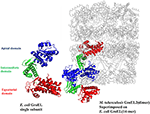
Figura 2. Struktura krystaliczna M. tuberculosis GroEL2 nałożona na strukturę E. coli GroEL-ES. Struktura M. tuberculosis GroEL2 (PDB ID:1SJP) wykazuje niższy stan oligomeryczny (dimer). Kolory niebieski, zielony i czerwony oznaczają odpowiednio domenę apikalną, pośrednią i równikową. W porównaniu do struktury E. coli GroEL (PDB ID: 1AON) pokazanej szarym kolorem, w strukturze M. tuberculosis GroEL2 interakcje między podjednostkami zachodzą poprzez domenę apikalną, podczas gdy w strukturze E. coli GroEL interakcje między podjednostkami zachodzą poprzez domenę równikową. Pojedyncza podjednostka M. tuberculosis GroEL2 jest wyrównana do pierścienia E. coli GroES związanego z GroEL, reprezentującego model asymetryczny. Struktura GroES została usunięta dla uproszczenia. Pojedyncza podjednostka E. coli GroEL została pokazana z domenami oznaczonymi tym samym kolorem w porównaniu do M. tuberculosis GroEL2 w celu analizy porównawczej. Program PyMOL (PyMOL Molecular Graphics System, wersja 1.3) został użyty do wygenerowania tego rysunku.
Różnorodność funkcjonalna
Ważne jest zbadanie, czy obecność wielu kopii chaperonin jest odpowiedzialna za zachowywanie się jako kanoniczne chaperoniny, czy też uległy one dywergencji w celu pełnienia nowych funkcji. Istotne jest również, czy te liczne chaperoniny działają na wspólnych substratach, czy też na odrębnych pulach substratów. GroELs s± wysoce konserwowane w różnych gatunkach i wykazano, że homologi chaperonin z innych bakterii s± zdolne do funkcjonowania w E. coli, co sugeruje nakładanie się białek substratowych i wspólny mechanizm działania GroEL. Oddziaływania białek substratowych z GroEL maj± charakter hydrofobowy, dlatego zmiana konformacyjna polegaj±ca na odsłonięciu domen apikalnych i równikowych we wgłębieniu odgrywa kluczow± rolę w rozpoznawaniu substratów i wspomaganiu fałdowania białek. Wiązanie białek substratowych do GroEL odbywa się poprzez domeny α/β białek nie wykazujących podobieństwa sekwencji (Kerner i in., 2005; Kumar i Mande, 2011), a dalsze badania sugerują, że GroEL selektywnie wiąże substraty globularne, a nie wydłużone polipeptydy (Robinson i in., 1994; Goldberg i in., 1997). Stwierdzono również, że wiele kopii chaperonin w organizmie ewoluowało, by pełnić nowe funkcje. Wykazano, że homolog GroEL w symbioncie owadów, Xenorhabdus nematophila, jest toksyczny dla owadów, co odbywa się poprzez wiązanie się z alfa-chityną. Analiza mutacyjna tych homologów GroEL sugeruje, że aminokwas krytyczny dla tego rodzaju aktywności jest różny od niezbędnego dla chaperoniny (Joshi i in., 2008). W M. tuberculosis, GroEL2 działa jako chaperonina uogólniająca (Hu i in., 2008), podczas gdy GroEL1 jest związana z nukleoidami (Basu i in., 2009). Widać więc, że duplikacja genów groEL doprowadziła do funkcjonalnej różnorodności chaperonin i/lub odrębnego spektrum substratów do wewnątrzkomórkowego składania białek.
Post-translational Modifications/Biofilm Formation
Post-translational modifications in proteins are employed by organisms to modulate their physiological processes and adapt to constantly changing environment (Bernal et al., 2014). Chaperoniny zostały zgłoszone do modyfikacji potranslacyjnych w niektórych organizmach, a ta modyfikacja została zgłoszona do zysku / utraty ich funkcji. Na przykład, frakcjonowanie lizatu komórek M. tuberculosis wykazało, że tetradecameryczna forma GroEL1 jest osiągana tylko po fosforylacji przy resztach serynowych (Kumar i in., 2009). Podobnie w innym doniesieniu wykazano, że fosforylacja zachodzi przy resztach treoninowych (Canova i in., 2009). Obie te obserwacje sugerują, że oligomeryzacja GroEL1 jest wynikiem modyfikacji potranslacyjnej.
Wielkie patogeny unikają wrodzonej odpowiedzi immunologicznej i stają się oporne na antybiotyki poprzez tworzenie biofilmów na komórkach nabłonka (Hall-Stoodley i Stoodley, 2005). Rola GroEL w tworzeniu biofilmu została wyjaśniona u kilku organizmów. Na przykład mutant GroEL1 u M. smegmatis nie tworzy biofilmu. Badania mechanistyczne wykazały, że GroEL1 M. smegmatis oddziałuje z enzymem KasA, który jest krytyczny dla biosyntezy kwasu mykolowego zaangażowanego w tworzenie biofilmu (Ojha et al., 2005). Co ciekawe, ostatnio stwierdzono, że GroEL w patogennym szczepie B. anthracis ulega fosforylacji i w ten sposób moduluje tworzenie biofilmu. Odkrycia te podkreślają, że fosforylacja GroEL ma implikacje funkcjonalne (Arora i in., 2017). Acetylacja jest kolejną modyfikacją posttranslacyjną związaną z chaperoninami E. coli i M. tuberculosis, jednak funkcjonalna rola tej modyfikacji nie została jeszcze przypisana (Liu i in., 2014). Podobnie mitochondrialna ko-chaperonina (mtHsp10) ulega modyfikacji acetylowej i kontroluje składanie białek mitochondrialnych w warunkach nadmiaru składników odżywczych (Lu i in., 2015).
C-Terminal Diversity
W różnych badaniach podkreśla się znaczenie C-końcowych reszt GroEL w ogólnym funkcjonowaniu chaperoniny (Tang i in., 2006; Chen i in., 2013). W przypadkach odnoszących się do wielu kopii chaperonin, mają one różny wzór reszt C-końcowych. Podczas gdy C-terminus GroEL (z E. coli) ma motyw 13 reszt (GGM)4M, homologi GroEL z innych organizmów (które zawierają wiele kopii chaperonin) mają odrębne motywy C-terminalne, takie jak:
a) C-terminal bogaty w histydynę, np. Mycobacteria (Colaco i MacDougall, 2014)
b) C-terminus bez wzoru, np, Rhizobia (George i in., 2004)
c) Podobne powtórzenia (GGM)4M, np. Myxobacteria (Wang i in., 2013)
d) Brak ogona podobnego do GGM, np. Methanosarcina (Figueiredo i in., 2004)
Wyraźnie widać, że wiele paralogów chaperonin w różnych organizmach ma C-terminus podobny do GGM. Widoczny jest szeroki zakres organizacji genomowej tych chaperonin. Ponadto, różnice są również postrzegane w ich koekspresji z co-chaperonin i essentiality ich funkcji. Tak więc, te paralogi są niepokojąco obserwuje się być albo istotne lub nieistotne, współekspresji z ich co-chaperonin lub nie współekspresji, i ewentualnie funkcjonować jako housekeeping chaperonin. Z drugiej strony, chaperoniny nie posiadające C-końca podobnego do GGM prawdopodobnie wyewoluowały w celu pełnienia nowych funkcji (Ojha i in., 2005; Wang i in., 2013; Rysunek 3).

Rysunek 3. Liczne chaperoniny u bakterii wykazujące różnorodność na C-końcu. Wyrównanie sekwencji podkreślające C-końcowe regiony reprezentatywnych bakteryjnych homologów GroEL z GroEL E. coli. Ostatnie reszty C-końcowe wybranych wielu GroEL u różnych bakterii wykazują rozbieżność z kanonicznym motywem (GGM)4M GroEL E. coli pokazanym w czerwonej przerywanej ramce. Sekwencje zostały pobrane z www.uniprot.org i wyrównane w MEGA6 przy użyciu algorytmu MUSCLE (www.megasoftware.net). Formatowanie wyrównanych sekwencji wykonano w przeglądarce wyrównania Jalview (www.jalview.org). Rezydy w wyrównaniu są zgodne z domyślnym schematem kolorów Clustal w Jalview.
Uwagi końcowe
Chaperoniny typu I są ważne ze względu na ich rolę w wewnątrzkomórkowym składaniu białek. Układ GroEL-GroES u bakterii wspomaga składanie około 10-15% białek cytozolowych. Różne struktury GroEL rozwiązane w formie apo-, nukleotydowej oraz w kompleksie z ko-chaperoniną GroES próbują wyjaśnić rolę tych chaperonin w składaniu białek (Saibil i in., 2013). Istnienie wielu chaperonin i ich rola w zróżnicowanych funkcjach wskazuje na presję ewolucyjną w kierunku adaptacji do różnych warunków środowiskowych. Struktura GroEL2 M. tuberculosis podkreśla niższy stan oligomeryczny i bardziej wyeksponowane powierzchnie hydrofobowe, prawdopodobnie w celu zwiększenia puli substratów i zachowania energii (Qamra i Mande, 2004; Qamra i in., 2004; Kumar i Mande, 2011). Ze względu na obecność bogatego w histydynę C-końca w wielu chaperoninach, zaproponowano, że mogą one pełnić alternatywne funkcje biologiczne. U M. smegmatis GroEL1 wiążąc się z żelazem może pomagać w tworzeniu biofilmu (Ojha i in., 2005). Defekt przeżywalności szczepu M. tuberculosis groEL1 knockout w warunkach niskiego napowietrzenia może pomagać w wyczuwaniu tlenu poprzez bezpośrednie wiązanie się z metalami lub pomagać niektórym metaloproteinom w składaniu (Sharma i in., 2016). Struktura innych homologicznych białek chaperoninowych prawdopodobnie odpowie na niezliczoną ilość pytań związanych z nowatorskimi funkcjami homologów chaperonin.
Wkład autorów
Wszyscy wymienieni autorzy wnieśli istotny, bezpośredni i intelektualny wkład w powstanie pracy oraz zatwierdzili ją do publikacji.
Oświadczenie o konflikcie interesów
Autorzy deklarują, że badania zostały przeprowadzone przy braku jakichkolwiek komercyjnych lub finansowych relacji, które mogłyby być interpretowane jako potencjalny konflikt interesów.
Podziękowania
MYA dziękuje za wsparcie finansowe dla Senior Research Fellowship z Departamentu Biotechnologii (DBT), Ministerstwa Nauki i Technologii, Rządu Indii. Autorzy chcieliby z wdzięcznością przyznać wsparcie finansowe dla pracy w laboratorium SCM poprzez granty BT/PR15450/COE/34/46/2016 i BT/PR3260/BRB/10/967/2011 z DBT.
An, Y. J., Rowland, S. E., Na, J. H., Spigolon, D., Hong, S. K., Yoon, Y. J., et al. (2017). Structural and mechanistic characterization of an archaeal-like chaperonin from a thermophilic bacterium. Nat. Commun. 8:827. doi: 10.1038/s41467-017-00980-z
PubMed Abstract | CrossRef Full Text | Google Scholar
Arora, G., Sajid, A., Virmani, R., Singhal, A., Kumar, C. M. S., Dhasmana, N., et al. (2017). Ser/Thr protein kinase PrkC-mediated regulation of GroEL is critical for biofilm formation in Bacillus anthracis. NPJ Biofilms Microbiomes 3:7. doi: 10.1038/s41522-017-0015-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Barreiro, C., González-Lavado, E., Pátek, M., and Martín, J. F. (2004). Transcriptional analysis of the groES-groEL1, groEL2, and dnaK genes in Corynebacterium glutamicum: characterization of heat shock-induced promoters. J. Bacteriol. 186, 4813-4817. doi: 10.1128/JB.186.14.4813-4817.2004
PubMed Abstract | CrossRef Full Text | Google Scholar
Basu, D., Khare, G., Singh, S., Tyagi, A., Khosla, S., and Mande, S. C. (2009). A novel nucleoid-associated protein of Mycobacterium tuberculosis is a sequence homolog of GroEL. Nucleic Acids Res. 37, 4944-4954. doi: 10.1093/nar/gkp502
PubMed Abstract | CrossRef Full Text | Google Scholar
Bernal, V., Castaño-Cerezo, S., Gallego-Jara, J., Écija-Conesa, A., de Diego, T., Iborra, J. L., et al. (2014). Regulacja fizjologii bakterii poprzez acetylację lizyny w białkach. N. Biotechnol. 31, 586-595. doi: 10.1016/j.nbt.2014.03.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Bohen, S. P., Kralli, A., and Yamamoto, K. R. (1995). Hold ’em and fold’ em: chaperones and signal transduction. Science 268, 1303-1304.
PubMed Abstract | Google Scholar
Bukau, B., and Horwich, A. L. (1998). The Hsp70 and Hsp60 chaperone machines. Cell 92, 351-366. doi: 10.1016/S0092-8674(00)80928-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Canova, M. J., Kremer, L., and Molle, V. (2009). The Mycobacterium tuberculosis GroEL1 chaperone is a substrate of Ser/Thr protein kinases. J. Bacteriol. 191, 2876-2883. doi: 10.1128/JB.01569-08
PubMed Abstract | CrossRef Full Text | Google Scholar
Chen, D. H., Madan, D., Weaver, J., Lin, Z., Schröder, G. F., Chiu, W., et al. (2013). Visualizing GroEL/ES in the act of encapsulating a folding protein. Cell 153, 1354-1365. doi: 10.1016/j.cell.2013.04.052
PubMed Abstract | CrossRef Full Text | Google Scholar
Cheng, M. Y., Hartl, F. U., Martin, J., Pollock, R. A., Kalousek, F., Neupert, W., et al. (1989). Mitochondrial heat-shock protein hsp60 is essential for assembly of proteins imported into yeast mitochondria. Nature 337, 620-625. doi: 10.1038/337620a0
PubMed Abstract | CrossRef Full Text | Google Scholar
Chilukoti, N., Kumar, C. M., and Mande, S. C. (2015). GroEL2 of Mycobacterium tuberculosis reveals the importance of structural pliability in chaperonin function. J. Bacteriol. 198, 486-497. doi: 10.1128/JB.00844-15
PubMed Abstract | CrossRef Full Text | Google Scholar
Colaco, C. A., and MacDougall, A. (2014). Mycobacterial chaperonins: the tail wags the dog. FEMS Microbiol. Lett. 350, 20-24. doi: 10.1111/1574-6968.12276
PubMed Abstract | CrossRef Full Text | Google Scholar
Cuéllar, J., Martín-Benito, J., Scheres, S. H., Sousa, R., Moro, F., López-Viñas, E., et al. (2008). The structure of CCT-Hsc70 NBD suggests a mechanism for Hsp70 delivery of substrates to the chaperonin. Nat. Struct. Mol. Biol. 15, 858-864. doi: 10.1038/nsmb.1464
PubMed Abstract | CrossRef Full Text | Google Scholar
Dickson, R., Weiss, C., Howard, R. J., Alldrick, S. P., Ellis, R. J., Lorimer, G., et al. (2000). Reconstitution of higher plant chloroplast chaperonin 60 tetradecamers active in protein folding. J. Biol. Chem. 275, 11829-11835. doi: 10.1074/jbc.275.16.11829
PubMed Abstract | CrossRef Full Text | Google Scholar
Duchêne, A. M., Thompson, C. J., and Mazodier, P. (1994). Transcriptional analysis of groEL genes in Streptomyces coelicolor A3(2). Mol. Gen. Genet. 245, 61-68. doi: 10.1007/BF00279751
PubMed Abstract | CrossRef Full Text | Google Scholar
Fei, X., Ye, X., LaRonde, N. A., and Lorimer, G. H. (2014). Formacja i struktury GroEL:Piłki nożne chaperoniny GroES2, forma funkcjonalna do składania białka. Proc. Natl. Acad. Sci. U.S.A. 111, 12775-12780. doi: 10.1073/pnas.1412922111
PubMed Abstract | CrossRef Full Text | Google Scholar
Figueiredo, L., Klunker, D., Ang, D., Naylor, D. J., Kerner, M. J., Georgopoulos, C., et al. (2004). Functional characterization of an archaeal GroEL/GroES chaperonin system: significance of substrate encapsulation. J. Biol. Chem. 279, 1090-1099. doi: 10.1074/jbc.M310914200
PubMed Abstract | CrossRef Full Text | Google Scholar
Fischer, H. M., Babst, M., Kaspar, T., Acuña, G., Arigoni, F., and Hennecke, H. (1993). One member of a gro-ESL-like chaperonin multigene family in Bradyrhizobium japonicum is co-regulated with symbiotic nitrogen fixation genes. EMBO J. 12, 2901-2912.
PubMed Abstract | Google Scholar
George, R., Kelly, S. M., Price, N. C., Erbse, A., Fisher, M., and Lund, P. A. (2004). Three GroEL homologues from Rhizobium leguminosarum have distinct in vitro properties. Biochem. Biophys. Res. Commun. 324, 822-828. doi: 10.1016/j.bbrc.2004.09.140
PubMed Abstract | CrossRef Full Text | Google Scholar
Gérard, H. C., Whittum-Hudson, J. A., Schumacher, H. R., and Hudson, A. P. (2004). Differential expression of three Chlamydia trachomatis hsp60-encoding genes in active vs. persistent infections. Microb. Pathog. 36, 35-39. doi: 10.1016/j.micpath.2003.08.005
PubMed Abstract | CrossRef Full Text | Google Scholar
Goldberg, M. S., Zhang, J., Sondek, S., Matthews, C. R., Fox, R. O., and Horwich, A. L. (1997). Native-like structure of a protein-folding intermediate bound to the chaperonin GroEL. Proc. Natl. Acad. Sci. U.S.A. 94, 1080-1085. doi: 10.1073/pnas.94.4.1080
PubMed Abstract | CrossRef Full Text | Google Scholar
Gould, P. S., Burgar, H. R., and Lund, P. A. (2007). Homologous cpn60 genes in Rhizobium leguminosarum are not functionally equivalent. Cell Stress Chaperones 12, 123-131. doi: 10.1379/CSC-227R.1
CrossRef Full Text | Google Scholar
Goyal, K., Qamra, R., and Mande, S. C. (2006). Wielokrotne powielanie genów i szybka ewolucja w genie groEL: implikacje funkcjonalne. J. Mol. Evol. 63, 781-787. doi: 10.1007/s00239-006-0037-7
PubMed Abstract | CrossRef Full Text | Google Scholar
Grandvalet, C., Rapoport, G., and Mazodier, P. (1998). hrcA, encoding the repressor of the groEL genes in Streptomyces albus G, is associated with a second dnaJ gene. J. Bacteriol. 180, 5129-5134.
Google Scholar
Hall-Stoodley, L., and Stoodley, P. (2005). Tworzenie i rozprzestrzenianie się biofilmów oraz przenoszenie patogenów przez człowieka. Trends Microbiol. 13, 7-10. doi: 10.1016/j.tim.2004.11.004
PubMed Abstract | CrossRef Full Text | Google Scholar
Hayer-Hartl, M. K., Martin, J., and Hartl, F. U. (1995). Asymmetrical interaction of GroEL and GroES in the ATPase cycle of assisted protein folding. Science 269, 836-841. doi: 10.1126/science.7638601
PubMed Abstract | CrossRef Full Text | Google Scholar
Hill, J. E., and Hemmingsen, S. M. (2001). Arabidopsis thaliana typ I i II chaperonin. Cell Stress Chaperones 6, 190-200. doi: 10.1379/1466-1268(2001)006<0190:ATTIAI>2.0.CO;2
PubMed Abstract | CrossRef Full Text | Google Scholar
Hu, Y., Henderson, B., Lund, P. A., Tormay, P., Ahmed, M. T., Gurcha, S. S., et al. (2008). A Mycobacterium tuberculosis mutant lacking the groEL homologue cpn60.1 is viable but fails to induce an inflammatory response in animal models of infection. Infect. Immun. 76, 1535-1546. doi: 10.1128/IAI.01078-07
PubMed Abstract | CrossRef Full Text | Google Scholar
Iizuka, R., So, S., Inobe, T., Yoshida, T., Zako, T., Kuwajima, K., et al. (2004). Role of the helical protrusion in the conformational change and molecular chaperone activity of the archaeal group II chaperonin. J. Biol. Chem. 279, 18834-18839. doi: 10.1074/jbc.M400839200
PubMed Abstract | CrossRef Full Text | Google Scholar
Joshi, M. C., Sharma, A., Kant, S., Birah, A., Gupta, G. P., Khan, S. R., et al. (2008). An insecticidal GroEL protein with chitin binding activity from Xenorhabdus nematophila. J. Biol. Chem. 283, 28287-28296. doi: 10.1074/jbc.M804416200
PubMed Abstract | CrossRef Full Text | Google Scholar
Kerner, M. J., Naylor, D. J., Ishihama, Y., Maier, T., Chang, H. C., Stines, A. P., et al. (2005). Proteome-wide analysis of chaperonin-dependent protein folding in Escherichia coli. Cell 122, 209-220. doi: 10.1016/j.cell.2005.05.028
PubMed Abstract | CrossRef Full Text | Google Scholar
Kim, Y. E., Hipp, M. S., Bracher, A., Hayer-Hartl, M., and Hartl, F. U. (2013). Molecular chaperone functions in protein folding and proteostasis. Annu. Rev. Biochem. 82, 323-355. doi: 10.1146/annurev-biochem-060208-092442
PubMed Abstract | CrossRef Full Text | Google Scholar
Kojima, K., and Nakamoto, H. (2007). A novel light- and heat-responsive regulation of the groE transcription in the absence of HrcA or CIRCE in cyanobacteria. FEBS Lett. 581, 1871-1880. doi: 10.1016/j.febslet.2007.03.084
PubMed Abstract | CrossRef Full Text | Google Scholar
Kol, A., Bourcier, T., Lichtman, A. H., and Libby, P. (1999). Chlamydial and human heat shock protein 60s activate human vascular endothelium, smooth muscle cells, and macrophages. J. Clin. Invest. 103, 571-577. doi: 10.1172/JCI5310
PubMed Abstract | CrossRef Full Text | Google Scholar
Koumoto, Y., Shimada, T., Kondo, M., Hara-Nishimura, I., and Nishimura, M. (2001). Chloroplasts have a novel Cpn10 in addition to Cpn20 as co-chaperonins in Arabidopsis thaliana. J. Biol. Chem. 276, 29688-29694. doi: 10.1074/jbc.M102330200
PubMed Abstract | CrossRef Full Text | Google Scholar
Kumar, C. M., Khare, G., Srikanth, C. V., Tyagi, A. K., Sardesai, A. A., and Mande, S. C. (2009). Facilitated oligomerization of mycobacterial GroEL: evidence for phosphorylation-mediated oligomerization. J. Bacteriol. 191, 6525-6538. doi: 10.1128/JB.00652-09
PubMed Abstract | CrossRef Full Text | Google Scholar
Kumar, S., and Mande, S. (2011). Protein chaperones and non-protein substrates: on substrate promiscuity of GroEL. Curr. Sci. 100, 1646-1653.
Google Scholar
Kumar, C. M., Mande, S. C., and Mahajan, G. (2015). Multiple chaperonins in bacteria-novel functions and non-canonical behaviors. Cell Stress Chaperones 20, 555-574. doi: 10.1007/s12192-015-0598-8
CrossRef Full Text | Google Scholar
Lindquist, S. (1986). The heat-shock response. Annu. Rev. Biochem. 55, 1151-1191. doi: 10.1146/annurev.bi.55.070186.005443
PubMed Abstract | CrossRef Full Text | Google Scholar
Lindquist, S., and Craig, E. A. (1988). The heat-shock proteins. Annu. Rev. Genet. 22, 631-677. doi: 10.1146/annurev.ge.22.120188.003215
PubMed Abstract | CrossRef Full Text | Google Scholar
Liu, F., Yang, M., Wang, X., Yang, S., Gu, J., Zhou, J., et al. (2014). Acetylome analysis reveals diverse functions of lysine acetylation in Mycobacterium tuberculosis. Mol. Cell. Proteomics 13, 3352-3366. doi: 10.1074/mcp.M114.041962
PubMed Abstract | CrossRef Full Text | Google Scholar
Lu, Z., Chen, Y., Aponte, A. M., Battaglia, V., Gucek, M., and Sack, M. N. (2015). Prolonged fasting identifies heat shock protein 10 as a Sirtuin 3 substrate elucidating a new mechanism linking mitochondrial protein acetylation to fatty acid oxidation enzyme folding and function. J. Biol. Chem. 290, 2466-2476. doi: 10.1074/jbc.M114.606228
PubMed Abstract | CrossRef Full Text | Google Scholar
Lund, P. A. (2009). Wiele chaperonin u bakterii – dlaczego tak wiele? FEMS Microbiol. Rev. 33, 785-800. doi: 10.1111/j.1574-6976.2009.00178.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Mande, S. C., Mehra, V., Bloom, B. R., and Hol, W. G. (1996). Structure of the heat shock protein chaperonin-10 of Mycobacterium leprae. Science 271, 203-207. doi: 10.1126/science.271.5246.203
PubMed Abstract | CrossRef Full Text | Google Scholar
McNally, D., and Fares, M. A. (2007). In silico identification of functional divergence between the multiple groEL gene paralogs in Chlamydiae. BMC Evol. Biol. 7:81. doi: 10.1186/1471-2148-7-81
PubMed Abstract | CrossRef Full Text | Google Scholar
Nielsen, K. L., and Cowan, N. J. (1998). A single ring is sufficient for productive chaperonin-mediated folding in vivo. Mol. Cell 2, 93-99. doi: 10.1016/S1097-2765(00)80117-3
PubMed Abstract | CrossRef Full Text | Google Scholar
Ogawa, J., and Long, S. R. (1995). The Rhizobium meliloti groELc locus is required for regulation of early nod genes by the transcription activator NodD. Genes Dev. 9, 714-729. doi: 10.1101/gad.9.6.714
PubMed Abstract | CrossRef Full Text | Google Scholar
Ojha, A., Anand, M., Bhatt, A., Kremer, L., Jacobs, W. R., and Hatfull, G. F. (2005). GroEL1: dedykowany chaperon zaangażowany w biosyntezę kwasu mykolowego podczas tworzenia biofilmu u prątków gruźlicy. Cell 123, 861-873. doi: 10.1016/j.cell.2005.09.012
PubMed Abstract | CrossRef Full Text | Google Scholar
Qamra, R., and Mande, S. C. (2004). Crystal structure of the 65-kilodalton heat shock protein, chaperonin 60.2, of Mycobacterium tuberculosis. J. Bacteriol. 186, 8105-8113. doi: 10.1128/JB.186.23.8105-8113.2004
PubMed Abstract | CrossRef Full Text | Google Scholar
Qamra, R., Srinivas, V., and Mande, S. C. (2004). Mycobacterium tuberculosis GroEL homologi nietypowo istnieją jako niższe oligomery i zachowują zdolność do tłumienia agregacji białek substratowych. J. Mol. Biol. 342, 605-617. doi: 10.1016/j.jmb.2004.07.066
PubMed Abstract | CrossRef Full Text | Google Scholar
Rajaram, H., and Apte, S. K. (2010). Differential regulation of groESL operon expression in response to heat and light in Anabaena. Arch. Microbiol. 192, 729-738. doi: 10.1007/s00203-010-0601-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Ranson, N. A., White, H. E., and Saibil, H. R. (1998). Chaperonins. Biochem. J. 333(Pt 2), 233-242. doi: 10.1042/bj3330233
PubMed Abstract | CrossRef Full Text | Google Scholar
Richter, K., Haslbeck, M., and Buchner, J. (2010). Odpowiedź na szok termiczny: życie na granicy śmierci. Mol. Cell 40, 253-266. doi: 10.1016/j.molcel.2010.10.006
PubMed Abstract | CrossRef Full Text | Google Scholar
Rinke de Wit, T. F., Bekelie, S., Osland, A., Miko, T. L., Hermans, P. W., van Soolingen, D., et al. (1992). Mycobacteria contain two groEL genes: the second Mycobacterium leprae groEL gene is arranged in an operon with groES. Mol. Microbiol. 6, 1995-2007. doi: 10.1111/j.1365-2958.1992.tb01372.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Robinson, C. V., Gross, M., Eyles, S. J., Ewbank, J. J., Mayhew, M., Hartl, F. U., et al. Conformation of GroEL-bound alpha-lactalbumin probed by mass spectrometry. Nature 372, 646-651. doi: 10.1038/372646a0
PubMed Abstract | CrossRef Full Text | Google Scholar
Saibil, H. R., Fenton, W. A., Clare, D. K., and Horwich, A. L. (2013). Structure and allostery of the chaperonin GroEL. J. Mol. Biol. 425, 1476-1487. doi: 10.1016/j.jmb.2012.11.028
PubMed Abstract | CrossRef Full Text | Google Scholar
Sameshima, T., Ueno, T., Iizuka, R., Ishii, N., Terada, N., Okabe, K., et al. (2008). Football- and bullet-shaped GroEL-GroES complexes coexist during the reaction cycle. J. Biol. Chem. 283, 23765-23773. doi: 10.1074/jbc.M802541200
PubMed Abstract | CrossRef Full Text | Google Scholar
Sato, S., Ikeuchi, M., and Nakamoto, H. (2008). Expression and function of a groEL paralog in the thermophilic cyanobacterium Thermosynechococcus elongatus under heat and cold stress. FEBS Lett. 582, 3389-3395. doi: 10.1016/j.febslet.2008.08.034
PubMed Abstract | CrossRef Full Text | Google Scholar
Schirmer, E. C., Glover, J. R., Singer, M. A., and Lindquist, S. (1996). HSP100/Clp proteins: a common mechanism explains diverse functions. Trends Biochem. Sci. 21, 289-296. doi: 10.1016/S0968-0004(96)10038-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Sharkia, R., Bonshtien, A. L., Mizrahi, I., Weiss, C., Niv, A., Lustig, A., et al. (2003). On the oligomeric state of chloroplast chaperonin 10 and chaperonin 20. Biochim. Biophys. Acta 1651, 76-84. doi: 10.1016/S1570-9639(03)00237-1
PubMed Abstract | CrossRef Full Text | Google Scholar
Sharma, A., Rustad, T., Mahajan, G., Kumar, A., Rao, K. V., Banerjee, S., et al. (2016). Towards understanding the biological function of the unusual chaperonin Cpn60.1 (GroEL1) of Mycobacterium tuberculosis. Tuberc. Edinb. Scotl. 97, 137-146. doi: 10.1016/j.tube.2015.11.003
PubMed Abstract | CrossRef Full Text | Google Scholar
Tang, Y. C., Chang, H. C., Roeben, A., Wischnewski, D., Wischnewski, N., Kerner, M. J., et al. (2006). Structural features of the GroEL-GroES nano-cage required for rapid folding of encapsulated protein. Cell 125, 903-914. doi: 10.1016/j.cell.2006.04.027
PubMed Abstract | CrossRef Full Text | Google Scholar
Techtmann, S. M., and Robb, F. T. (2010). Archaeal-like chaperonins in bacteria. Proc. Natl. Acad. Sci. U.S.A. 107, 20269-20274. doi: 10.1073/pnas.1004783107
PubMed Abstract | CrossRef Full Text | Google Scholar
Tsai, Y. C., Mueller-Cajar, O., Saschenbrecker, S., Hartl, F. U., and Hayer-Hartl, M. (2012). Chaperonin cofactors, Cpn10 and Cpn20, of green algae and plants function as hetero-oligomeric ring complexes. J. Biol. Chem. 287, 20471-20481. doi: 10.1074/jbc.M112.365411
PubMed Abstract | CrossRef Full Text | Google Scholar
Viitanen, P. V., Lorimer, G. H., Seetharam, R., Gupta, R. S.Oppenheim, J., Thomas, J. O., et al. (1992). Mammalian mitochondrial chaperonin 60 funkcjonuje jako pojedynczy pierścień toroidalny. J. Biol. Chem. 267, 695-698.
PubMed Abstract | Google Scholar
Wang, Y., Zhang, W. Y., Zhang, Z., Li, J., Li, Z. F., Tan, Z. G., et al. (2013). Mechanisms involved in the functional divergence of duplicated GroEL chaperonins in Myxococcus xanthus DK1622. PLoS Genet. 9:e1003306. doi: 10.1371/journal.pgen.1003306
PubMed Abstract | CrossRef Full Text | Google Scholar
Ye, X., and Lorimer, G. H. (2013). Substrate protein switches GroE chaperonins from asymmetric to symmetric cycling by catalyzing nucleotide exchange. Proc. Natl. Acad. Sci. U.S.A. 110, E4289-E4297. doi: 10.1073/pnas.1317702110
PubMed Abstract | CrossRef Full Text | Google Scholar
.