Co to jest ChIP?
Imunoprecypitacja chromatyny, lub ChIP, jest technologią opartą na przeciwciałach, używaną do selektywnego wzbogacania specyficznych białek wiążących DNA wraz z ich celami DNA. ChIP jest używany do badania konkretnej interakcji białko-DNA, kilku interakcji białko-DNA lub interakcji w całym genomie lub podzbiorze genów.
ChIP wykorzystuje przeciwciała, które selektywnie rozpoznają i wiążą białka, w tym histony, modyfikacje histonów, czynniki transkrypcyjne i kofaktory, aby dostarczyć informacji o stanach chromatyny i transkrypcji genów. Połączenie analizy proteomicznej i technik biologii molekularnej stosowanych w ChIP pozwala na zrozumienie ekspresji i regulacji genów w komórkach lub tkankach będących przedmiotem zainteresowania.
Kiedy stosuje się ChIP?
Typowo, ChIP jest stosowany do identyfikacji względnej obfitości specyficznego białka lub specyficznej modyfikacji białka w określonym regionie genomu. ChIP może być używany do odpowiedzi na wiele pytań naukowych związanych z interakcją białek i chromatyny. Na przykład, ChIP może być użyty do porównania obecności pewnych białek w różnych loci, mapowania różnych białek w interesującym nas regionie genomu lub ilościowego określenia wiązania białek do indukowalnego genu w odpowiedzi na bodziec w czasie.
Jak działa ChIP?
Zasada działania ChIP jest stosunkowo prosta i polega na użyciu przeciwciała do wyizolowania lub wytrącenia określonego białka, histonu, czynnika transkrypcyjnego lub kofaktora i związanej z nim chromatyny z mieszaniny białek, która została wyekstrahowana z komórek lub tkanek. Stąd nazwa tej techniki: Chromatin Immunoprecipitation. W ChIP-PCR lub ChIP-seq, wzbogacone immunologicznie fragmenty DNA są następnie identyfikowane i oznaczane ilościowo przy użyciu powszechnie dostępnych odczynników PCR lub qPCR oraz technologii sekwencjonowania następnej generacji (NGS).
Co to jest natywne ChIP (N-ChIP) vs usieciowane ChIP (X-ChIP)?
Istnieją 2 rodzaje technik ChIP, które mogą być wykonywane w zależności od pytania eksperymentalnego i materiału wyjściowego do eksperymentu: 1) natywne ChIP (N-ChIP) i 2) usieciowane ChIP (X-ChIP). Oba typy ChIP mają wady i zalety:
- W N-ChIP nie stosuje się środka utrwalającego do usieciowania białek do chromatyny. Zamiast tego, natywna chromatyna jest izolowana z jąder komórkowych, które są trawione nukleazą. Ponieważ przeciwciała są hodowane przeciwko nieutrwalonym antygenom, N-ChIP oferuje korzyść w postaci lepszego rozpoznawania i wiązania przeciwciał do ich docelowych antygenów. PCR może nie być wymagane do dalszej analizy ze względu na wysoką liczebność białek histonowych. Chociaż te zalety sprawiają, że N-ChIP jest atrakcyjną metodą, może być ona stosowana tylko do wykrywania histonów. Dodatkowo, utrata wiązania białka podczas trawienia chromatyny i etapów immunoprecypitacji może zniekształcać dane lub utrudniać właściwe analizy.
- W X-ChIP, chemiczne utrwalacze takie jak formaldehyd są używane do usieciowania interesującego nas białka do DNA, a fragmentacja chromatyny jest osiągana poprzez sonikację lub trawienie nukleazą. Zaletą X-ChIP jest to, że może być stosowany z białkami histonowymi i niehistonowymi i generalnie wymaga mniej komórkowego materiału wyjściowego niż N-ChIP. X-ChIP minimalizuje również szanse na utratę białek chromatyny podczas ekstrakcji, co pozwala na wykrywanie przejściowych interakcji białkowych. Jednakże etap wytrącania jest mniej wydajny, a amplifikacja DNA przez PCR jest konieczna do dalszych analiz.
Jakie są różne typy badań ChIP?
Po zakończeniu immunoprecypitacji chromatyny można przeprowadzić kilka dalszych analiz oczyszczonej chromatyny i związanych z nią białek, histonów, czynników transkrypcyjnych i kofaktorów. Najpopularniejszymi metodami analizy pojedynczego genu i całego genomu są odpowiednio qPCR i ChIP-seq. PCR i ChIP-chip są również opcjami dla analizy downstream.
5.1 Jakie są zalety ChIP-PCR?
ChIP-PCR jest wykonywany w celu analizy modyfikacji histonów i/lub wiązania białek do znanego podzbioru docelowych loci w genomie. W ChIP-PCR, wzbogacone immunologicznie fragmenty DNA są identyfikowane i oznaczane ilościowo przy użyciu powszechnie dostępnych odczynników i technologii PCR lub qPCR. Szybkie i ilościowe porównanie specyficznych regionów w genomie w wielu próbkach może być osiągnięte przy użyciu ChIP-qPCR. Jest to tańsze i bardziej efektywne czasowo niż metody sekwencjonowania całego genomu.
5.2 Jakie są zalety ChIP-chip?
Technologia ChIP-chip odnosi się do wykorzystania mikroukładu mikromacierzy DNA do analizy fragmentów DNA wzbogaconych immunoglobuliną ChIP. Wykorzystanie technologii mikromacierzy kafelkowania genomu pozwala na analizę całego genomu białek, które są związane z izolowanym DNA i generuje genomową mapę o wysokiej rozdzielczości wiązania białek i modyfikacji białek. ChIP-chip ma wiele zastosowań w badaniach podstawowych, jak również w badaniach nad chorobami. Na przykład, może być użyty do identyfikacji miejsc wiązania czynników transkrypcyjnych, enhancerów i represorów oraz do porównania tych typów wiązania białek w próbkach kontrolnych i patologicznych. Ponieważ jednak koszt NGS znacznie się zmniejszył, a podobne wyniki uzyskuje się przy użyciu ChIP-seq, więcej osób decyduje się na wykonanie ChIP-seq zamiast ChIP-chip.
5.3 Jakie są zalety ChIP-seq?
Podobnie jak ChIP-chip, ChIP-seq dostarcza informacji o wiązaniu białek w całym genomie. Jednak w przeciwieństwie do ChIP-chip, ChIP-seq wykorzystuje technologię NGS do identyfikacji fragmentów DNA i mapowania ich względem całego genomu.
Nowocześniejsza technologia amplifikacji DNA pozwala na przeprowadzenie solidnej analizy w ciągu kilku dni przy niewielkiej ilości wejściowego DNA. Gdy materiał wyjściowy jest rzadki, te postępy technologiczne w metodach przygotowywania bibliotek umożliwiły eksperymenty ChIP-seq.
Dodatkowo, nowa technologia, w której próbki DNA są jednoznacznie znakowane krótkimi sekwencjami, znanymi jako kody kreskowe, pozwala obecnie na łączenie pojedynczych fragmentów w pojedynczy pas sekwencjonowania do analizy multipleksowej. To znacznie zwiększyło wydajność i obniżyło koszt eksperymentów sekwencjonowania DNA, dodatkowo wspierając zastosowania ChIP-seq.
W sumie, dzięki postępowi w technologii sekwencjonowania DNA, zaletą ChIP-seq jest to, że duża liczba próbek DNA wzbogaconych w ChIP może być sekwencjonowana tanio w stosunkowo krótkim czasie z wyższą czułością i dokładnością niż ChIP-chip.
Jakie są różne kroki w badaniu ChIP?
Badania ChIP przebiegają zgodnie z ogólnym protokołem:
- Sieciowanie białek do DNA tylko dla X-ChIP
- Liza komórek
- Fragmentacja chromatyny przez trawienie (dla X-ChIP i N-ChIP) lub ścinanie oparte na sonikacji (dla X-ChIP i N-ChIP).ścinanie (tylko dla X-ChIP)
- Immunoprecypitacja przy użyciu specyficznych przeciwciał
- Czyszczenie DNA do dalszych analiz
- Analiza DNA poprzez PCR, qPCR, mikromacierz, lub NGS
Ważne, pozytywne i negatywne kontrole na każdym etapie są integralne dla określenia, czy eksperyment ChIP zakończył się sukcesem.
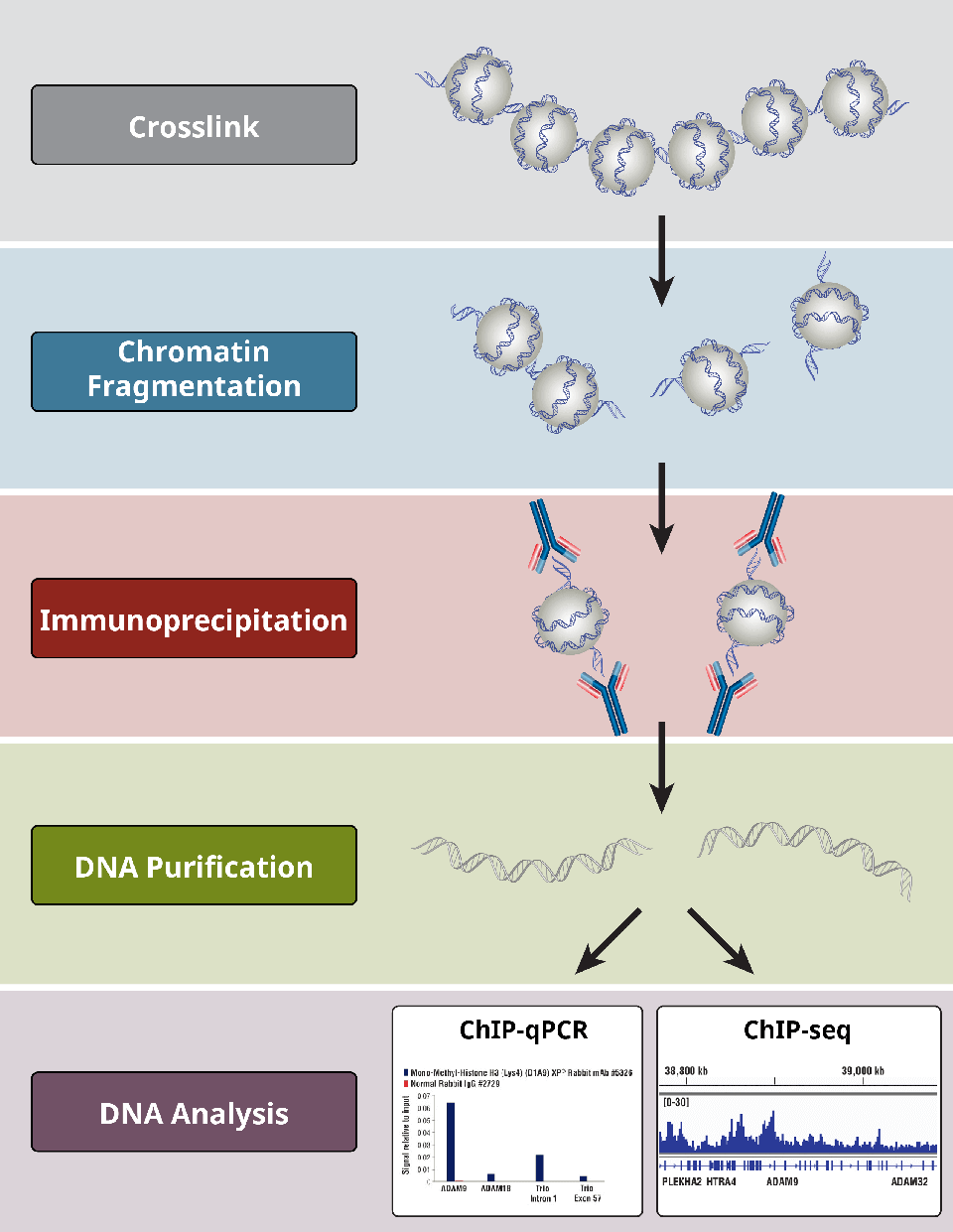
Przegląd najbardziej krytycznych etapów protokołu ChIP.
Przegląd najbardziej krytycznych etapów protokołu ChIP.
6.1 Jak usieciować komórki i tkanki do ChIP?
Odczynniki sieciujące są używane do „przytwierdzania” białek do wiązanego przez nie DNA. Odczynniki na bazie formaldehydu są zwykle używane do osiągnięcia tego wiązania. Komórki i tkanki są ogólnie utrwalane w podobny sposób, ale tkanki wymagają dłuższego czasu utrwalania i szybszego dostarczania utrwalacza w celu szybkiego przeniknięcia do tkanki docelowej, zanim zacznie się degenerować.
Nadmierne utrwalenie chromatyny może zmniejszyć wydajność fragmentacji przez sonikację, oprócz hamowania wiązania przeciwciał z ich docelowymi białkami. Dlatego czas utrwalania powinien być ustalony empirycznie, aby umożliwić maksymalne wiązanie przeciwciała z antygenem przy jednoczesnym osiągnięciu idealnego usieciowania białek z ich docelowym DNA.
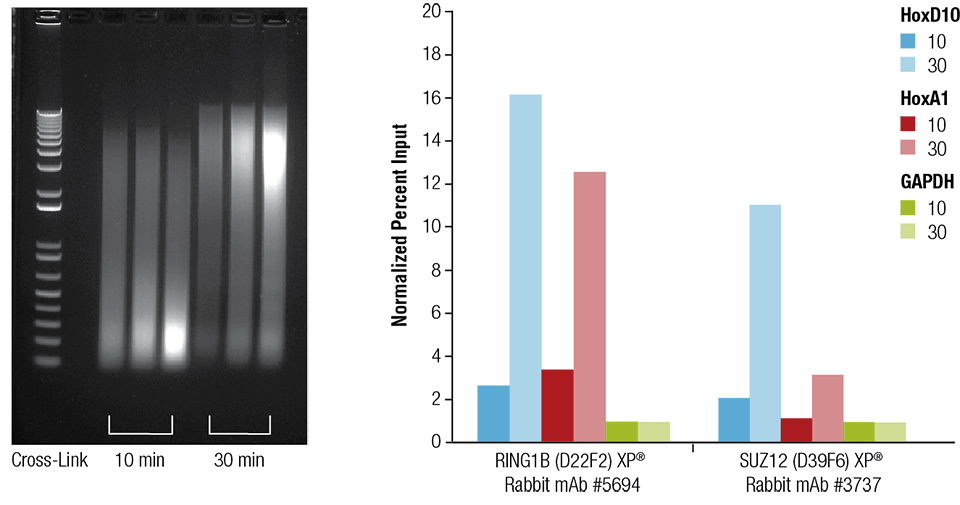
Serce myszy (H), mózg (B) i wątroba (L) zostały usieciowane przez 10 lub 30 minut, jak wskazano (lewy panel). Chromatyna została przygotowana i poddana sonikacji przez 4 minuty. ChIP przeprowadzono przy użyciu chromatyny przygotowanej z tkanki serca ze wskazanymi przeciwciałami przy użyciu zestawu SimpleChIP® Plus Sonication Chromatin IP Kit #56383, a wzbogacony DNA był kwantyfikowany przez PCR w czasie rzeczywistym przy użyciu primerów do wskazanych genów (prawy panel). Ilość immunoprecypitowanego DNA w każdej próbce jest przedstawiona jako znormalizowany sygnał do negatywnego loci GAPDH, który jest równy 1.
Serce myszy (H), mózg (B) i wątrobę (L) sieciowano przez 10 lub 30 minut, jak wskazano (lewy panel). Chromatyna została przygotowana i poddana sonikacji przez 4 minuty. ChIP przeprowadzono przy użyciu chromatyny przygotowanej z tkanki serca ze wskazanymi przeciwciałami przy użyciu zestawu SimpleChIP® Plus Sonication Chromatin IP Kit #56383, a wzbogacony DNA był kwantyfikowany przez PCR w czasie rzeczywistym przy użyciu primerów do wskazanych genów (prawy panel). Ilość immunoprecypitowanego DNA w każdej próbce jest przedstawiona jako znormalizowany sygnał do negatywnego loci GAPDH, który jest równy 1.
6.2 Jak fragmentować chromatynę?
Fragmentacja chromatyny jest niezbędna dla powodzenia eksperymentu ChIP. Fragmentacja chromatyny jest niezbędna do jej solubilizacji i pozwala na jej koprecypitację. Ponadto, rozdzielczość badania ChIP zależy od fragmentacji chromatyny, ponieważ wielkość fragmentu DNA określa rozdzielczość badania ChIP.
Trawienie enzymatyczne wykorzystuje nukleazę mikrokalkową (MNase), która rozszczepia dwuniciowy DNA pomiędzy nukleosomami w celu wygenerowania fragmentów chromatyny. Podczas gdy pełne trawienie MNazą generuje fragmenty DNA o długości 150 par zasad (mononukleosomy), niekompletne trawienie generuje fragmenty DNA o długości od 150 do 750 par zasad (mono-, di- i tri-nukleosomy). Sonikacja wykorzystuje siłę mechaniczną do fragmentacji chromatyny. Sonikacja fragmentuje chromatynę pomiędzy i wewnątrz nukleosomów, generując zakres fragmentów chromatyny pomiędzy 150 a 1000 par zasad.
W przypadku X-ChIP, do ścinania chromatyny stosuje się trawienie enzymatyczne lub sonikację. Warunki sonikacji w protokole sonikacji ChIP powinny być ustalane empirycznie, ponieważ różnią się one w zależności od typu komórki i warunków eksperymentalnych. Warunki trawienia są bardziej spójne w różnych typach komórek i tkanek, ale rozmiar fragmentu chromatyny powinien być nadal analizowany przed IP.
Dla N-ChIP, nukleazy są używane do fragmentacji chromatyny w celu utrzymania wiązania białka w nieutrwalonych próbkach. Fragmentacja nukleaz powinna być również ustalona empirycznie, aby zminimalizować nadmierne trawienie chromatyny.
6.3 Dlaczego stosować trawienie enzymatyczne do ChIP?
Trawienie nukleazami musi być stosowane do N-ChIP, ponieważ białka nie są usieciowane z DNA, a trudne warunki związane z fragmentacją opartą na sonikacji spowodowałyby dysocjację białek chromatyny od DNA. N-ChIP jest idealny do analizy interakcji białko histonowe-DNA, ponieważ wiązanie histon-DNA jest bardzo silne i stabilne. Jednakże, N-ChIP nie działa dobrze w przypadku analizy wiązania czynnika transkrypcyjnego i kofaktora chromatyny.
Do fragmentacji chromatyny w X-ChIP można użyć trawienia enzymatycznego lub sonikacji. Korzyści z trawienia enzymatycznego obejmują spójność fragmentacji i łagodne warunki fragmentacji (niższa temperatura i detergent), które lepiej zachowują integralność chromatyny i epitopów przeciwciał, co skutkuje zwiększonym wzbogaceniem immunologicznym chromatyny związanej z czynnikiem transkrypcyjnym i kofaktorem.
6.4 Dlaczego warto używać sonikacji do fragmentacji chromatyny do ChIP?
W przeciwieństwie do fragmentacji chromatyny uzyskanej przez trawienie enzymatyczne, sonikacja opiera się na siłach mechanicznych do fragmentacji chromatyny na mniejsze kawałki. Idealny rozmiar fragmentów chromatyny do wzbogacania immunologicznego wynosi od 200 do 1000 par zasad. Sonikacja jest tradycyjną metodą stosowaną do fragmentacji chromatyny i może być wykonywana przy użyciu tradycyjnego sonikatora z sondą lub bardziej zaawansowanych sonikatorów z łaźnią wodną, które zapewniają bardziej ukierunkowaną sonikację. Sonikacja generuje prawdziwie randomizowane fragmenty chromatyny; wymaga jednak szerokiej optymalizacji dla różnych linii komórkowych i tkanek oraz jest trudna do odtworzenia z eksperymentu na eksperyment. Wymóg stosowania buforów o wysokiej zawartości detergentów oraz ciepło generowane podczas sonikacji mogą uszkodzić integralność zarówno chromatyny, jak i epitopów przeciwciał na białkach chromatyny.
6.5 Optymalizacja sonikacji chromatyny dla ChIP
Fragmentacja chromatyny oparta na sonikacji tradycyjnie wykorzystuje bufory o wysokiej zawartości detergentów i generuje ciepło, z których oba mogą uszkodzić integralność chromatyny i epitopów przeciwciał. Dlatego ilość sonikacji używanej do fragmentacji chromatyny musi być ustalona doświadczalnie dla różnych linii komórkowych i tkanek. Należy zidentyfikować i użyć minimalnej ilości sonikacji wymaganej do wygenerowania fragmentów DNA o długości od 150 do 1000 par zasad w celu zminimalizowania uszkodzeń chromatyny.
Przed rozpoczęciem pełnego badania ChIP z dalszą analizą przez qPCR, DNA chip, lub NGS, elektroforeza żelowa powinna być użyta do analizy próbek chromatyny poddanych sonikacji przez różny czas. Wielkość fragmentu zależy od czasu sonikacji – wielkość fragmentu zmniejsza się wraz z wydłużaniem czasu sonikacji. Jednak dane sugerują, że dłuższy czas sonikacji nie prowadzi do lepszych wyników. W związku z tym, przeprowadzenie oczyszczonego immunoprecypitowanego DNA na żelu i określenie idealnego rozmiaru fragmentu jest prostą metodą określenia minimalnej ilości sonikacji wymaganej do uzyskania pożądanego rozmiaru DNA i uniknięcia niepotrzebnego uszkodzenia chromatyny.

Trawiona enzymem chromatyna została przeprowadzona na żelu agarozowym. Pas 1 pokazuje chromatynę, która jest niedostatecznie strawiona. Lane 2 pokazuje prawidłowo strawioną chromatynę, a Lane 3 pokazuje chromatynę, która jest nadtrawiona.
Chromatyna trawiona enzymem była prowadzona na żelu agarozowym. Pas 1 pokazuje chromatynę, która jest niedostatecznie strawiona. Pas 2 pokazuje prawidłowo strawioną chromatynę, a pas 3 pokazuje chromatynę, która jest nadtrawiona.
6.6 Jak wybrać przeciwciało do ChIP?
Wybór odpowiedniego przeciwciała do eksperymentu ChIP jest integralną częścią jego sukcesu. Przeciwciała używane w eksperymencie ChIP powinny być specyficzne dla interesującego nas białka i mieć wysokie powinowactwo do antygenu. Najlepszym wyborem przeciwciała do eksperymentu ChIP lub ChIP-seq jest przeciwciało zwalidowane pod kątem ChIP- lub ChIP-seq. Jeśli dla danego genu nie jest dostępne przeciwciało zwalidowane metodą ChIP, następnym najlepszym wyborem jest przeciwciało, które zostało zwalidowane metodą IP. Należy zauważyć, że nie wszystkie przeciwciała zwalidowane w IP działają w ChIP i nie wszystkie zwalidowane w ChIP działają w ChIP-seq. Ponadto, im więcej przeciwciał jest zwalidowanych w innych zastosowaniach, takich jak western, IP, IF, flow i IHC, tym większą pewność można mieć co do wydajności i specyficzności przeciwciała. Po walidacji przeciwciała musi nastąpić empiryczne określenie optymalnego stężenia przeciwciała wraz z warunkami płukania IP.
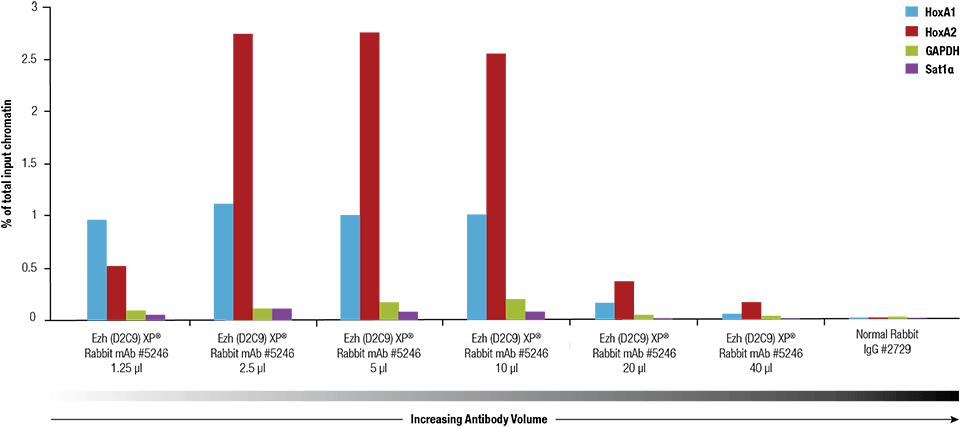
Ezh2 (D2C9) XP® Rabbit mAb #5246 miareczkowano przy użyciu SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 na usieciowanej chromatynie przygotowanej z 4 x 106 komórek NCCIT.
Ezh2 (D2C9) XP® Rabbit mAb #5246 miareczkowano przy użyciu zestawu SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 na usieciowanej chromatynie przygotowanej z 4 x 106 komórek NCCIT.
6.7 Jak wykonać immunoprecypitację
Przeciwciała są używane do wychwytywania interesującego nas białka i związanego z nim DNA. Stężenie przeciwciał powinno być ustalane empirycznie, z ogólnym punktem wyjścia 0,5-2,0 μg przeciwciał używanych na 10 μg chromatynowego DNA (odpowiada to w przybliżeniu 4 x 106 komórkom). Surowość buforów i czasy płukania powinny być również ustalane empirycznie, ponieważ zależą one od powinowactwa przeciwciała do antygenu docelowego. Zazwyczaj, inkubacje przeciwciało:chromatyna są wykonywane przez 2 godziny do nocy.
Kompleksy przeciwciało-antygen (+DNA) są wychwytywane przez powinowactwo na żywicy wiążącej przeciwciało. W eksperymentach ChIP żywica ta składa się zwykle z kulek magnetycznych, sefarozowych lub agarozowych klasy ChIP, sprzężonych z białkiem A i/lub białkiem B. Przeciwciała wiążą kulki białka A i/lub białka G z różnym powinowactwem, w zależności od gatunku, w którym zostały opracowane i podtypu IgG ich łańcucha ciężkiego. Kulki są zazwyczaj inkubowane z przeciwciałem:chromatyna przez 2 do 4 godzin.
Potrzebne są etapy płukania, aby usunąć chromatynę niezwiązaną z przeciwciałem, a następnie odwrócenie wiązań krzyżowych (dla X-ChIP) i oczyszczenie DNA. Dodatkowo, należy przeprowadzić kontrolne IP IgG w celu określenia tła (sygnał:szum). Przeciwciała kontroli pozytywnej (np. całkowity histon H3) i/lub primery qPCR kontroli pozytywnej (dla znanych pozytywnych i negatywnych loci wiążących białka docelowe) muszą być również włączone w celu określenia wiązania niespecyficznego. Aby uzyskać optymalne wyniki, QC chromatyny IP przez qPCR należy przeprowadzić przed dalszą analizą NGS.
6.8 Jak eluować chromatynę z kulek białka A/G
Chromatyna jest eluowana z kulek białka A/G przy użyciu detergentu i ciepła. Niska prędkość „worteksowania” lub mieszania jest wymagana do utrzymania kulek w zawiesinie i zwiększenia elucji chromatyny.
6.9 Jak odwrócić wiązania krzyżowe chromatyny
Wiązania krzyżowe są odwracane przez wysokie ciepło i wysoką sól (oba te składniki są niezbędne). Proteinaza K jest również dodawana do trawienia powiązanych białek chromatyny i dodawanych przeciwciał, co pozwala na bardziej wydajne oczyszczanie DNA.
6.10 Jak oczyszczać DNA
Po usunięciu wiązań krzyżowych chromatyny, DNA jest oczyszczane za pomocą klasycznych metod fenolowo-chloroformowych, po których następuje wytrącanie etanolem lub za pomocą zestawów do oczyszczania DNA na kolumnach.
Jak analizować wzbogacone DNA
Po oczyszczeniu DNA można przeprowadzić kilka analiz downstream, w tym ChIP-PCR, ChIP-qPCR, ChIP-chip i ChIP-seq.
7.1 Analiza ChIP-PCR i ChIP-qPCR
Analizy ChIP-PCR i ChIP-qPCR są najlepsze do analizy pojedynczych genów i mogą być stosowane do amplifikacji i kwantyfikacji specyficznych fragmentów DNA w szybki i opłacalny sposób.
7.2 Analiza ChIP-chip
Analiza ChIP-chip wykorzystująca mikroukłady mikromacierzy DNA w celu stworzenia obejmującej cały genom, wysokiej rozdzielczości mapy wiązania białek i ich modyfikacji.
7.3 Analiza ChIP-seq
Analiza ChIP-seq wykorzystuje standardową technologię NGS do zestrojenia oczyszczonego DNA z uprzednio zanotowanymi całymi genomami w celu identyfikacji profili wiązania białek obejmujących cały genom.