Características genômicas de A. brassicae e dois outros co-infectantes A. alternata isolates
Sequenciamos os genomas de dois isolados de A. alternata (PN1 e PN2) que estavam co-infectando B. juncea com A. brassicae. A montagem de A. brassicae foi previamente descrita . Em resumo, a montagem consistiu de nove cromossomos completos e um cromossomo com repetições teloméricas ausentes em uma das extremidades. Além desses cromossomos, havia seis contíguos dos quais um deles tinha ~ 1 Mb de tamanho, que juntos podem constituir um cromossomo dispensável (Fig. 1). O N50 da montagem de A. brassicae era de 2,98 Mb (Tabela 1). Os dois isolados co-infectados B. juncea foram identificados como A. alternata com base nas suas sequências ITS e GAPDH. As montagens A. alternata Aat_PN1 e Aat_PN2 consistiram de 14 contigs totalizando 33,77 Mb, e 15 contigs totalizando 33,53 Mb, respectivamente (Tabela 1). Seis contigs em cada um dos dois conjuntos continham repetições teloméricas em ambas as extremidades e, portanto, são mais prováveis de representar moléculas cromossômicas completas. Quatro outras contíguas em ambas as montagens continham repetições teloméricas em uma das extremidades, mas eram de tamanho similar às moléculas cromossômicas completas, como descrito em A. solani . Portanto, as montagens do genoma de A. alternata isolados representaram dez cromossomos quase completos de cada um dos dois isolados.
Alinhamentos genômicos inteiros com Alternaria spp. relacionados mostraram uma sintonia geral entre os genomas com rearranjos menores (Fig. 2). Adicionalmente, seqüências mitocondriais também foram obtidas a partir dos dados de seqüenciamento dos dois isolados de A. alternata. Os genomas mitocondriais das linhagens A. alternata foram aproximadamente 49.783 bp e 50.765 bp, respectivamente, e mostraram alta similaridade com os genomas mitocondriais anteriormente publicados do genoma A. alternata .
Previsão do gene após mascaramento repetido resultou na identificação de 11593, 11495 e 11387 genes nos genomas A. brassicae, A. alternata PN1, e PN2, respectivamente. Isto foi comparável aos números de genes estimados em outras Alternaria spp. (Tabela 1). A análise BUSCO mostrou que os modelos de genes previstos nos três genomas cobriam 98% dos genes fúngicos conservados em uma única cópia, indicando a quase totalidade das assembléias. Os genes previstos foram anotados de forma abrangente usando uma combinação de bases de dados como descrito na seção Métodos (Fig. 1). Além dos três genomas, nós também previmos genes de novo nas assembléias genômicas de três outras espécies de Alternaria que foram sequenciadas usando tecnologias de leitura longa, como A. brassicicola (abra43), A. alternata (ATCC34957), e A. solani (altNL03003) (Tabela 1). Estes seis genomas e suas predições genéticas foram usados para a análise comparativa de grupos de genes codificadores de metabolitos secundários e genes efetores-codificadores.

Sumário de A. genoma de Brassicae, (De trilhas circulares externas para internas) a pseudocromossomos/esqueletos, b genes codificadores de proteínas, c Elementos repetidos, d Elementos transponíveis (DNA e LTR), e grupos de metabólitos secundários previstos, f Proteínas secretas, g efetores previstos
Análise filogenômica atribui um clade separado para o Brassica-infectante A. brassicae e A. brassicicola dentro dos gêneros Alternaria
Para reconstruir com precisão a divergência e relação entre A. brassicae, os dois isolados A. alternata (PN1 e PN2), e as outras espécies Alternaria, realizamos análises filogenômicas usando 29 ortologenos de cópia única que tiveram o maior sinal filogenético calculado pelo programa Mirlo. A seleção de genes com maior sinal filogenético leva a filogenias que são mais congruentes com a árvore da espécie. A filogenia resultante mostrou que as espécies de Alternaria de grande porte e Alternaria de pequeno porte se agruparam separadamente em dois clades diferentes (Fig. 3). Curiosamente, os dois principais agentes patogênicos das Brassicas, A. brassicae e A. brassicola se agruparam separadamente de todas as outras espécies de Alternaria, possivelmente indicando uma trajetória evolutiva diferente com base nas preferências comuns dos hospedeiros dessas duas espécies.
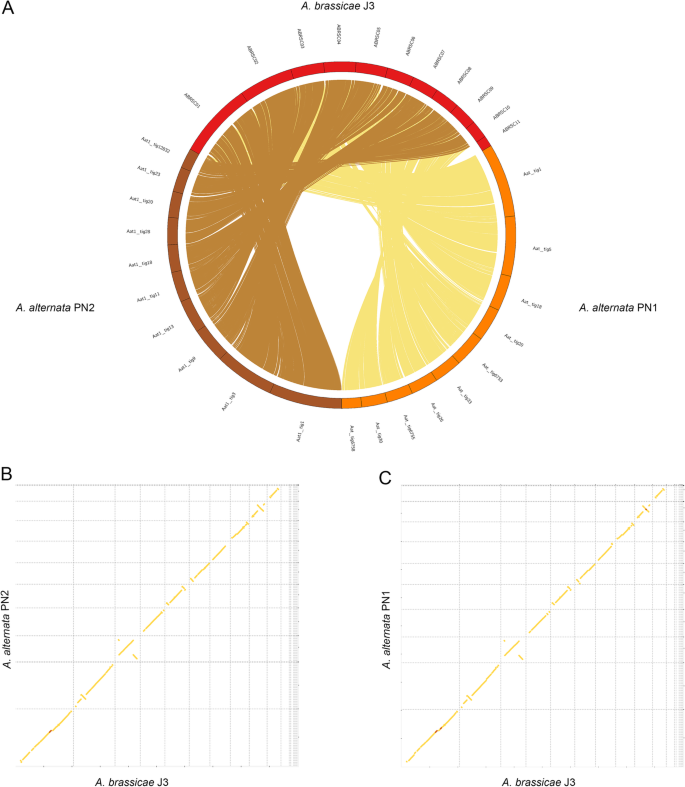
Alinhamentos de A. alternata PN1 e PN2 com A. brassicae. um gráfico Circos mostrando a macrossimilhança de A. alternata PN1 e PN2 com A. brassicola. brassicae em todos os contíguos exceto os contíguos dispensáveis (ABRSC11, scaffold13,17,18,19), b e c Lotes sintéticos de A. brassicae com A. alternata PN1 e PN2
Análises comparativas de A. alternata PN1 e PN2 com A. brassicae. alternata isolados obtidos de diferentes hospedeiros
Comparamos os genomas de A. alternata PN1 e PN2 (isolados de B. juncea) com o de A. alternata ATCC34957 (isolados de sorgo) para identificar quaisquer diferenças em seu conteúdo genômico que possam permitir que estes infectem duas espécies muito diferentes. Alinhamentos genómicos inteiros de A. alternata PN1 e PN2 com o de A. alternata ATCC34957 revelaram níveis muito elevados de sintonia e a ausência de quaisquer regiões específicas da espécie. Identificamos 719, 152 e 586 genes isolados específicos entre os três isolados de A. alternata, respectivamente (arquivo adicional 1: Tabela S1). Mais de dois terços dos genes específicos isolados nos três isolados eram proteínas não caracterizadas ou não tinham anotações. Notavelmente, todos os três isolados não continham nenhum cromossoma dispensável que pudesse conferir patogenicidade, como foi relatado para os isolados de A. alternata que infectam muitas das culturas fruteiras, tais como citrinos, peras e maçãs. Os repertórios genéticos dos três isolados também consistiram de um número e tipo similar de efetores, CAZymes e grupos de metabólitos secundários (Tabela 2). Além disso, os dois isolados PN1 e PN2 não causam por si só sintomas de infecção em B. juncea sob condições epifitóticas (dados não mostrados). Nossos resultados sugerem que estes isolados de A. alternata (PN1 e PN2) podem ser patógenos facultativos que levam a um estilo de vida saprófito e podem mudar para um estilo de vida patogênico sob certas condições ambientais.
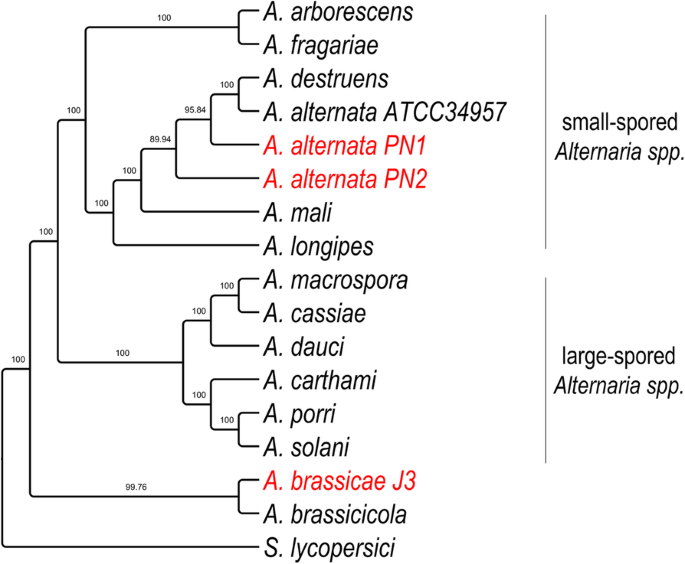
Árvore filogenética de espécies Alternaria com S. lycopersici como um grupo externo. A árvore foi construída utilizando 29 ortologias de cópia única, que tiveram o maior sinal filogenético, conforme calculado em Mirlo. Os valores dos suportes de ramo de 1000 réplicas de bootstrap são mostrados
Uma abundância de regiões ricas em repetição e elementos transponíveis em A. brassicae
Patógenos vegetais filamentosos tendem a ter uma arquitetura do genoma distinta com maior conteúdo de repetição. A estimativa de conteúdo de repetição e mascaramento usando RepeatModeler e RepeatMasker revelou que o genoma A. brassicae consistia de ~ 9,33% de repetições em comparação com 2,43 e 2,64% de repetições nos genomas A. alternata. O genoma de A. brassicae abriga o maior conteúdo de repetição (~ 9,33%) entre todas as espécies Alternaria sequenciadas até a data. Nossa análise mostrou que o conteúdo de repetição difere significativamente entre os isolados de A. alternata e as outras espécies de Alternaria patogênicas. As espécies patogênicas Alternaria especialmente A. brassicae e A. brassicola tinham um repertório consideravelmente maior de elementos LTR/Gypsy e LTR/Copia (> 8X) em comparação com os outros A. alternata isolados (patogênicos e não patogênicos) (Fig. 4). Os genomas A. brassicola e A. brassicola também tiveram uma super-representação dos transpositores de DNA, que totalizaram ~ 5% do genoma, em comparação com < 1% nas outras espécies Alternaria (Fig. 4).

Comparação de conteúdo repetido em seis espécies Alternaria. O tamanho das bolhas corresponde à (a) percentagem de elementos transponíveis (ET) no genoma, b número de cópia da ET no genoma
>
Esta proliferação de ADN repetitivo e subsequente evolução dos genes que se sobrepõem a estas regiões pode ser a chave para o sucesso evolutivo onde estes patogénios conseguiram persistir ao longo de gerações de conflito co-evolucionário com os seus hospedeiros. A proximidade com ET expõe potencialmente os genes a mutações de pontos induzidos por repetição (RIP) e, portanto, a evolução acelerada. A recombinação ectópica entre ETs similares também pode resultar em novas combinações de genes e assim aumentar a diversidade de proteínas ou metabolitos.
Presença de um cromossoma dispensável no A. brassicae de grandes dimensões
Cromossomos específicos de linhagem (LS) ou cromossomos dispensáveis (DC) foram relatados de várias espécies fitopatogênicas, incluindo A. alternata. Os DCs em A. alternata são conhecidos por conferirem virulência e especificidade do hospedeiro ao isolado. O alinhamento do genoma inteiro de A. brassicae com outras Alternaria spp. revelou que um contigente de aproximadamente 1 Mb junto com outros contigentes menores (66-366 kb) era específico para A. brassicae e não mostrava sintenização para nenhuma região nas outras Alternaria spp. No entanto, a sintenização parcial foi observada quando o contigente foi alinhado com as seqüências de outros cromossomos dispensáveis relatados em Alternaria spp. . Isto nos levou a supor que estes contigentes juntos podem representar uma DC de A. brassicae. Para confirmar isto, nós procuramos nas contigs pela presença de AaMSAS e ALT1genes, que são genes marcadores conhecidos para cromossomos dispensáveis em Alternaria spp. . Encontramos duas cópias do gene AaMSAS como parte de dois aglomerados biossintéticos do metabolito secundário no contigente 1 Mb. Entretanto, não encontramos nenhum homólogo do gene ALT1. Além disso, o conteúdo repetido dos contigentes (ABRSC11, andaime 13, 17, 18, e 19) foi comparado com todo o genoma. O conteúdo gênico das contigs específicas da linhagem foi significativamente inferior ao dos cromossomos do núcleo (Tabela 3). Por outro lado, os contíguos DC foram altamente enriquecidos em conteúdo de ET quando comparados aos cromossomos do núcleo (Tabela 3).
Embora a DC não tenha sido enriquecida com genes que codificam proteínas secretadas, a proporção de genes efetores secretados foi 30% maior quando comparada aos cromossomos do núcleo. Todas as evidências acima apontam para o fato de que A. brassicae pode de fato abrigar uma DC. As DCs em Alternaria spp. têm sido relatadas até agora apenas das Alternaria spp. de esporos pequenos e nenhuma espécie de Alternaria de esporos grandes tem sido conhecida por abrigar DCs. Resta saber se a CD contribui para a virulência de A. brassicae. Estudos futuros envolveriam a caracterização do cromossomo dispensável em A. brassicae e correlacionar sua presença com a patogenicidade de diferentes isolados.
Análise ortográfica revela genes específicos de espécies com papéis putativos na virulência
Diferenças no conteúdo e diversidade de genes dentro dos genes contribuem para a adaptação, crescimento e patogenicidade. A fim de catalogar as diferenças no conteúdo gênico dentro do gênero Alternaria e dos Dothideomycetes, realizamos uma análise ortográfica sobre o conjunto combinado de 3.60.216 proteínas de 30 espécies diferentes (incluindo 16 espécies Alternaria) pertencentes aos Dothideomycetes (arquivo adicional 2: Tabela S2) das quais 3.45.321 proteínas poderiam ser atribuídas pelo menos a um dos grupos ortogonais. Identificamos 460 genes específicos de A. brassicae que estavam presentes em A. brassicae mas ausentes em todas as outras espécies de Alternaria (Arquivo adicional 3: Tabela S3). Estes genes específicos da espécie incluíam 35 genes codificadores de proteínas secretadas, dos quais 11 foram previstos como sendo efetores. Além disso, 20 destes genes específicos das espécies estavam presentes na CD. Um grande número destas proteínas pertencia à categoria de proteínas não caracterizadas sem função conhecida. A fim de testar se estes genes específicos das espécies são o resultado da evolução adaptativa que ocorre nas regiões ricas em repetição do genoma, realizamos um teste de permutação para comparar a sobreposição de regiões ricas em repetição e elementos transponíveis com um gene aleatório contra a sobreposição destes genes específicos das espécies. Verificamos que estes genes específicos das espécies se sobrepuseram significativamente com regiões ricas em repetição (P-valor: 9,99e-05; Z-score: – 4,825) e elementos transponíveis (P-valor: 0,0460; Z-score: 2,539) no genoma.
Perfil metabólico secundário de A. brassicae e sua associação com elementos transponíveis (ETs)
Os gêneros de Alternaria e Cochliobolus são conhecidos por serem os maiores produtores de toxinas metabólicas secundárias específicas do hospedeiro. Alternaria spp. especialmente são conhecidos pela produção de metabolitos secundários quimicamente diversos, que incluem as toxinas específicas do hospedeiro (HSTs) e não-HSTs. Estes metabólitos secundários são geralmente gerados por peptídeos sintéticos não ribo-somais (NRPS) e poliquetas sintéticas (PKS). Identificamos cinco grupos de genes NRPS tipo SM, 12 grupos de genes tipo PKS e sete grupos de genes do tipo terpeno em A. brassicae (arquivo adicional 4: Tabela S4). Dos cinco grupos de NRPS, pudemos identificar três grupos que produzem metabólitos secundários conhecidos: Destruxina B, HC-toxina e dimetilcoprogênio (sideróforo) com papéis conhecidos na virulência.
O grupo de genes responsável pela produção de dimetilcoprogênio (sideróforo) em A. brassicae consiste de 22 genes, incluindo os principais genes biossintéticos, oxidoreductases e transportadores de sideróforo. Os sideróforos são compostos de ferro, usados por fungos para adquirir ferro férrico extracelular e têm sido relatados como envolvidos na virulência fúngica. A identificação do cluster genético responsável pela síntese de sideróforos permitiria o estudo dos sideróforos e seu papel na patogenicidade em A. brassicae. Adicionalmente, também foi identificado um cluster do tipo PKS constituído de 12 genes, responsáveis pela produção de melanina (arquivo adicional 4: Tabela S4). O aglomerado biossintético da melanina foi descrito anteriormente para A. alternata . Também, o fator de transcrição Amr1, que induz a produção de melanina, foi caracterizado em A. brassicicola e é conhecido por suprimir a virulência . Contudo, o papel da melanina na virulência é ambíguo e específico da espécie .
Os patógenos vegetais pertencentes ao gênero Alternaria parecem ter uma capacidade dinâmica para adquirir novo potencial metabólico secundário para colonizar novos nichos ecológicos. A explicação mais parcimoniosa para essa aquisição dinâmica do potencial de metabólitos secundários é a transferência horizontal de genes dentro do gênero Alternaria e possivelmente com outros gêneros. Há uma extensa evidência na literatura que grande parte dos HSTs de Alternaria são carregados nos cromossomos dispensáveis e a troca desses cromossomos pode ampliar a especificidade do hospedeiro. Também identificamos um cluster NRPS, possivelmente codificando para HC-toxina em uma das CDs (andaime 18) (arquivo adicional 4: Tabela S4). O HC-toxina é um determinante conhecido da virulência do patógeno vegetal Cochliobolus carbonum, que infecta genótipos do milho que não possuem uma cópia funcional do HM1, uma carbonil redutase que desintoxica a toxina . Um relatório recente mostrou que A. jesenskae também poderia produzir HC-toxina, tornando-o o único outro fungo além de C. carbonum a produzir a toxina . A presença do cluster de genes HC-toxina, um determinante de virulência em C. carbonum, em uma DC de A. brassicae aponta para o fato de que a transferência horizontal de genes entre espécies pode ser mais comum do que o esperado.
Parte da transferência horizontal de genes, duplicação rápida, divergência e perda dos genes SM também podem contribuir para a evolução do patógeno e novas capacidades metabólicas. Estes processos de duplicação e divergência podem muito bem ser auxiliados pela proximidade dos aglomerados de metabólitos secundários aos elementos de repetição que os tornam propensos a mutações RIP. Portanto, nós testamos se os clusters de metabólitos secundários também foram associados a regiões ricas em repetição. Um teste de permutação foi usado para comparar a sobreposição de regiões ricas em repetição com um conjunto de genes aleatórios contra a sobreposição de genes de clusters de metabólitos secundários. Os clusters de metabólitos secundários sobrepuseram significativamente as regiões ricas em repetição em comparação com o conjunto de genes aleatórios (valor P: 0,0017; Z-score: – 2,7963). Além disso, estes clusters sobrepuseram-se significativamente com elementos transponíveis entre as regiões ricas em repetição (valor P: 0,0087; Z-score: 2,9871). Isto mostra que ambos os mecanismos descritos acima para a aquisição de novo potencial metabólico secundário podem ser possíveis no caso de A. brassicae. Análises em escala populacional a nível de espécie e gênero podem lançar luz sobre a prevalência desses mecanismos dentro do gênero Alternaria.
Análise de Synteny revela a base genética da exclusividade da produção de Destruxin B por A. brassicae dentro do gênero Alternaria
Destruxina B representa uma classe de depsipeptídeos cíclicos que é conhecida por ser um dos principais fatores de patogenicidade de A. brassicae e tem sido relatada como sendo uma toxina hospedeira específica de A. brassicae . Destruxina B não foi reportada como sendo produzida por nenhuma das outras espécies de Alternaria. Aqui relatamos pela primeira vez os grupos de genes biossintéticos responsáveis pela produção de Destruxin B em A. brassicae. O cluster consiste em 10 genes, incluindo a principal enzima biossintética codificada por um gene NRPS (DtxS1) e a enzima limitadora de taxa, DtxS3 (aldo-keto reductase) (arquivo adicional 4: Tabela S4). Curiosamente, a análise sintética deste cluster entre as seis espécies de Alternaria mostrou que estes dois genes não estavam presentes em nenhuma das outras Alternaria spp. embora a síntese global do cluster tenha sido mantida em todas estas espécies (Fig. 5). A ausência dos genes-chave codificadores das enzimas DtxS1 e DtxS3 no aglomerado Destruxin B nas outras espécies de Alternaria explica a ausência de Destruxin B nessas espécies.

Synteny do aglomerado Destruxin B nas seis espécies de Alternaria. DtxS1 e DtxS3 estão marcados em vermelho (rotulados com os respectivos IDs genéticos) e estão ausentes de todas as outras espécies de Alternaria
Destruxina B foi relatada a partir do entomopatógeno Metarhizium robertsii , e Ophiosphaerella herpotricha , a causa da mancha morta de bermudagrass, além de A. brassicae. Wang et al. descreveram o aglomerado biossintético do metabolito secundário responsável pela produção de Destruxin em M. robertsii . O aglomerado de Destruxin na espécie Metarhizhium consiste em dois genes chave adicionais, DtxS2 e DtxS4. DtxS2 é responsável pela conversão de Destruxin B em Destruxin A, C, D, e E. Não encontramos nenhum gene sintético no cluster que corresponde a DtxS2 ou DtxS4 em A. brassicae. A. brassicae é conhecido por produzir apenas Destruxin B e não as outras formas de Destruxin, o que poderia possivelmente explicar a ausência de DtxS2 em A. brassicae. DtxS4 é uma descarboxilase de aspartato que é necessária para a produção de β-alanina a partir do ácido aspártico e é essencial para a produção da estrutura da espinha dorsal da Destruxina. Além disso, os mutantes de eliminação do DtxS4 foram capazes de produzir Destruxin quando foram suplementados com β-alanina no meio . Embora o DtxS4 não esteja presente no aglomerado Destruxin, A. brassicae tem uma cópia funcional de descarboxilase de aspartato presente em outros lugares do genoma e, portanto, pode possivelmente garantir o fornecimento de β-alanina para a síntese de Destruxin B. A história evolutiva da produção de Destruxin B dentro do gênero Metarhizhium se assemelhava muito à de Alternaria. Os patógenos especializados como o M. acridum não possuem as duas principais enzimas e, portanto, não produzem Destruxinas. Contudo, M. robertsii, um generalista com uma gama de hospedeiros mais ampla, produz Destruxinas . Por isso, foi feita a hipótese de que Destruxins poderia ser responsável pelo estabelecimento da gama de hospedeiros alargada de M. roberstii. Uma hipótese semelhante pode também ser verdadeira no caso de A. brassicae, que tem uma ampla gama de hospedeiros e pode infectar quase todos os Brassicaceae. Experimentos adicionais para determinar a gama de hospedeiros de Destruxin mutantes nulos de A. brassicae podem ajudar a esclarecer o papel dessa importante fitotoxina na extensão da gama de hospedeiros de A. brassicae.
Distinto perfil CAZyme de A. brassicae e A. brassicola dentro do gênero Alternaria
CAZymes (Carbohidrato – Enzimas Ativas) são proteínas envolvidas na degradação, rearranjo, ou síntese de ligações glicosídicas. Os patógenos vegetais secretam uma gama diversificada de CAZymes que decompõem os complexos polissacáridos na parede celular da planta. Eles consistem em cinco classes distintas: hidrolases de glicosídeos (GH), glicosiltransferases (GT), lisases de polissacarídeos (PL), esterases de carboidratos (CE), e módulos de ligação de carboidratos (CBM). Identificamos > 500 CAZymes nas seis Alternaria spp. incluindo A. brassicae (Arquivo adicional 5: Tabela S5). A distribuição CAZyme de A. brassicae e A. brassicicola variou das outras Alternaria spp. formando assim um aglomerado separado (Fig. 6). O número de enzimas de atividade auxiliar ou as enzimas envolvidas na degradação da parede celular das plantas variou consideravelmente entre as diferentes espécies em comparação. Quase 46% das CAZymes em A. brassicae foram secretadas, das quais ~ 17% foram previstas como efetoras.

Mapa de aquecimento dos perfis CAZyme em 16 espécies com o gênero Alternaria. O agrupamento dos organismos se assemelha muito ao da filogenia das espécies, exceto A. brassicae e A. brassicicola, que se agrupam separadamente com o grupo Stemphylium lycopersici. As CAZymes foram agrupadas de acordo com a classificação CAZy
A família AA9 (anteriormente GH61; monooxigenases polissacarídeo lítico dependente do cobre (LPMO)) é significativamente aumentada em comparação com as outras famílias CAZy no gênero Alternaria com cada espécie contendo > 20 cópias do gene. Os números de cópias no gênero Alternaria spp. são muito maiores que os números de cópias relatados para Botrytis e Fusarium spp. . A família AA9 está envolvida na degradação dos polissacarídeos da parede celular e é conhecida por atuar em uma gama de polissacarídeos incluindo amido, xiloglucano, celodextrinas, e glucomanano. LPMOs têm sido hipotéticos a ter um duplo papel – clivar diretamente os polissacarídeos da parede celular, e atuar como um gerador de ROS e, assim, contribuir para o estresse oxidativo que leva à necrose dos tecidos vegetais. De forma impressionante, 11 das 26 proteínas AA9 presentes em A. brassicae estão previstas como agentes secretores de efeito. A caracterização dessas CAZymes e seu papel na patogênese poderia ser objeto de estudos adicionais.
Perfil de efector de sobreposição dentro do gênero Alternaria
Prevemos o repertório de seis espécies de Alternaria (Tabela 2) incluindo A. brassicae usando o pipeline descrito na seção de métodos. A maioria das pequenas proteínas secretadas previstas para serem efetores foram proteínas não caracterizadas/previstas e, portanto, podem ser novas espécies ou efetores específicos do gênero. A. brassicae tinha a maior proporção de proteínas desconhecidas/previstas/hipotéticas no conjunto de efectores candidatos. Descobrimos que a maioria dos efectores entre as seis espécies são comuns e sobrepostos, sugerindo que os amplos mecanismos de patogénese envolvendo efectores proteináceos podem ser conservados dentro do género. No entanto, encontramos duas cópias de uma dobra beta/gama-cristalina contendo proteína a estar presente apenas em A. brassicae e A. brassicicola e não nas outras quatro espécies de Alternaria. Uma pesquisa adicional através da base de dados nr do NCBI revelou que esta proteína estava completamente ausente no gênero Alternaria e as combinações mais próximas foram encontradas em outros patógenos vegetais, como Macrophomina e Fusarium. Entretanto, nenhuma informação está disponível quanto à sua função ou papel na patogenicidade em qualquer das espécies.
Pudemos também estabelecer que alguns dos efetores em A. brassicae têm o potencial de evoluir de forma adaptativa, uma vez que também foram significativamente associados com as regiões ricas em repetição do genoma (P-valor: 0,0003; Z-score: – 2,8778). Portanto, análises a nível populacional são necessárias para identificar os efetores sob seleção positiva, o que poderia esclarecer a evolução da patogenicidade em A. brassicae. Os efetores identificados neste estudo revelam a ampla gama de proteínas que podem estar envolvidas na patogênese de A. brassicae. 39 destes efetores foram preditos como CAZymes tendo vários papéis na degradação da parede celular e polissacarídeos associados. O genoma de A. brassicae continha duas necroses e proteínas peptídeas indutoras de etileno, que têm sido implicadas na patogênese de vários patógenos, incluindo oomicetos e necrotrofos. Hrip1 (Hypersensitive response inducing protein 1) from A. alternata has recently been shown to be recognised by MdNLR16 in a classical gene-for-gene manner, and delete of Hrip1 from A. alternata enhances its virulence . Um homólogo Hrip1 também está presente em A. brassicae, mas não está previsto que seja secretado fora da célula, embora isso precise ser verificado experimentalmente. A presença de efectores que são reconhecidos de forma gene a gene abre a possibilidade de identificação de genes R complementares no hospedeiro que podem ser utilizados para o desenvolvimento de variedades ou cultivares resistentes.