Genomiska egenskaper hos A. brassicae och två andra saminfekterande A. alternata-isolat
Vi sekvenserade genomet hos två isolat av A. alternata (PN1 och PN2) som saminfekterade B. juncea med A. brassicae. A. brassicae-samlingen har tidigare beskrivits . I korthet bestod den av nio kompletta kromosomer och en kromosom där telomeriska upprepningar saknas i en av ändarna. Förutom dessa kromosomer fanns det sex kontigs, varav en var ~ 1 Mb stor, som tillsammans kan utgöra en överflödig kromosom (fig. 1). N50 för A. brassicae-samlingen var 2,98 Mb (tabell 1). De två isolaten som saminfekterade B. juncea identifierades som A. alternata baserat på deras ITS- och GAPDH-sekvenser. A. alternata-sammansättningarna Aat_PN1 och Aat_PN2 bestod av 14 contigs på totalt 33,77 Mb respektive 15 contigs på totalt 33,53 Mb (tabell 1). Sex contigs i var och en av de två sammansättningarna innehöll telomeriska upprepningar i båda ändarna och representerar därför sannolikt fullständiga kromosomala molekyler. Fyra andra contigs i båda sammansättningarna innehöll telomeriska upprepningar i ena änden men var av liknande storlek som fullständiga kromosommolekyler enligt beskrivningen i A. solani . Därför representerade genomsammansättningarna för A. alternata-isolat tio nästan kompletta kromosomer för vart och ett av de två isolaten.
Helgenomanpassningar med besläktade Alternaria spp. visade på en övergripande syntes mellan genomerna med mindre omarrangemang (fig. 2). Dessutom erhölls mitokondriella sekvenser från sekvenseringsdata för de två isolaten av A. alternata. Mitokondriegenomerna för A. alternata-stammarna var ungefär 49 783 bp respektive 50 765 bp stora och uppvisade stor likhet med det tidigare publicerade mitokondriegenomet av A. alternata.
Genprediktion efter upprepningsmaskering resulterade i identifiering av 11593, 11495 och 11387 gener i A. brassicae-, A. alternata PN1- respektive PN2-genomkomplexen. Detta var jämförbart med det uppskattade antalet gener i andra Alternaria spp. (tabell 1). BUSCO-analysen visade att de genmodeller som förutspåddes i de tre genomerna täckte 98 % av de konserverade svampgenerna i enstaka kopior, vilket tyder på att sammansättningarna är nästan kompletta. De förutsagda generna annoterades utförligt med hjälp av en kombination av databaser enligt beskrivningen i avsnittet Metoder (fig. 1). Förutom de tre genomerna förutspådde vi också gener de novo i genomsamlingar av tre andra Alternaria-arter som sekvenserades med hjälp av teknik med långa läsningar, nämligen A. brassicicola (abra43), A. alternata (ATCC34957) och A. solani (altNL03003) (tabell 1). Dessa sex genomer och deras genprediktioner användes för jämförande analyser av sekundärmetabolitkodande genkluster och effektorkodande gener.

Sammanfattning av A. brassicae-genom, (från yttre till inre cirkulära spår) a pseudokromosomer/scaffolds, b proteinkodande gener, c repetitiva element, d transposabla element (DNA och LTR), e förutspådda kluster av sekundära metaboliter, f utsöndrade proteiner, g förutspådda effektorer
Fylogenomisk analys tilldelar en separat klad för den Brassica-infekterande A. Brassicae och A. brassicicola inom Alternaria-släktet
För att noggrant rekonstruera divergensen och förhållandet mellan A. brassicae, de två A. alternata-isolaten (PN1 och PN2) och de andra Alternaria-arterna utförde vi fylogenomiska analyser med hjälp av 29 ortologer i enstaka kopior som hade den högsta fylogenetiska signalen beräknad av programmet Mirlo. Val av gener med högre fylogenetiska signaler leder till fylogenier som är mer kongruenta med artträdet . Den resulterande fylogenin visade att de storspåriga Alternaria-arterna och de småspåriga Alternaria-arterna samlades separat i två olika klasser (fig. 3). Intressant är att de två största patogenerna av Brassica, nämligen A. brassicae och A. brassicicola, grupperade sig separat från alla andra Alternaria-arter, vilket möjligen tyder på att de två arterna har en annan evolutionär utveckling som bygger på deras gemensamma värdpreferenser.
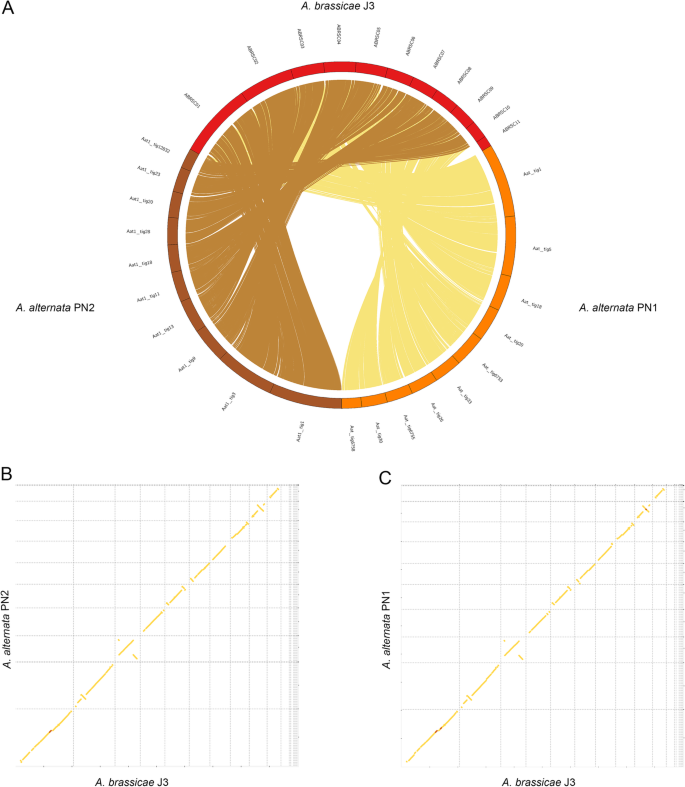
Helgenomanpassningar av A. alternata PN1 och PN2 till A. brassicae. a Cirkosplot som visar makrosyntesen av A. alternata PN1 och PN2 med A. brassicae. brassicae över alla contigs utom de överflödiga contigs (ABRSC11, scaffold13,17,18,19), b och c Syntenic dotplots av A. brassicae med A. alternata PN1 och PN2
Komparativa analyser av A. brassicae med A. alternata PN1 och PN2
. alternata-isolat från olika värdar
Vi jämförde genomet av A. alternata PN1 och PN2 (isolerade från B. juncea) med genomet av A. alternata ATCC34957 (isolerad från sorghum) för att identifiera eventuella skillnader i genominnehållet som skulle kunna göra det möjligt för dessa att infektera två mycket olika arter. En anpassning av hela genomet från A. alternata PN1 och PN2 till A. alternata ATCC34957 avslöjade mycket höga nivåer av syntes och avsaknad av artspecifika regioner. Vi identifierade 719, 152 och 586 isolatspecifika gener mellan de tre isolaten av A. alternata (Additional file 1: Table S1). Mer än två tredjedelar av de isolatspecifika generna i alla tre isolaten var okarakteriserade proteiner eller hade inga annoteringar. Det är anmärkningsvärt att alla tre isolaten inte innehöll några överflödiga kromosomer som kan ge patogenitet, vilket har rapporterats för A. alternata-isolat som infekterar många fruktgrödor, t.ex. citrusfrukter, päron och äpplen. Genrepertoaren hos de tre isolaten bestod också av liknande antal och typ av effektorer, CAZymer och kluster av sekundära metaboliter (tabell 2). Dessutom orsakar de två isolaten PN1 och PN2 inga egna infektionssymptom hos B. juncea under epifytotiska förhållanden (data visas inte). Våra resultat tyder på att dessa isolat av A. alternata (PN1 och PN2) kan vara fakultativa patogener som leder en saprofytisk livsstil och kan övergå till en patogen livsstil under vissa miljöförhållanden.
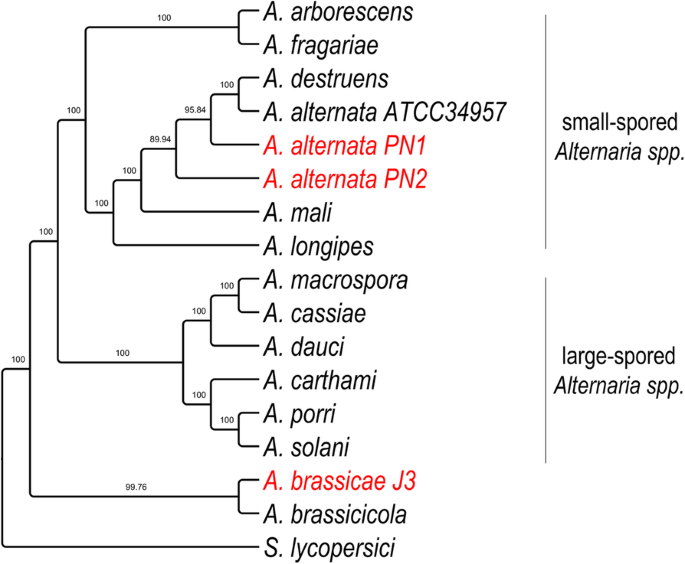
Fyloogenetiskt träd för Alternaria-arter med S. lycopersici som en outgroup. Trädet konstruerades med hjälp av 29 ortologer med en enda kopia, som hade den högsta fylogenetiska signalen enligt beräkningen i Mirlo. Grenstödsvärden från 1000 bootstrapreplikat visas
Ett överflöd av repetitionsrika regioner och transposabla element i A. brassicae
Filamentösa växtpatogener tenderar att ha en distinkt genomarkitektur med högre innehåll av upprepningar. Uppskattning av upprepningsinnehållet och maskering med hjälp av RepeatModeler och RepeatMasker visade att A. brassicae-genomet bestod av ~ 9,33 % upprepningar jämfört med 2,43 och 2,64 % upprepningar i A. alternata-genomen. A. brassicae-genomet har det högsta innehållet av upprepningar (~ 9,33 %) bland alla Alternaria-arter som hittills har sekvenserats. Vår analys visade att upprepningsinnehållet skiljer sig avsevärt mellan A. alternata-isolaten och de andra patogena Alternaria-arterna. De patogena Alternaria-arterna, särskilt A. brassicae och A. brassicicola, hade en betydligt större repertoar av LTR/Gypsy- och LTR/Copia-element (> 8X) i jämförelse med övriga A. alternata-isolat (patogena och icke-patogena) (fig. 4). Genomen från A. brassicae och A. brassicicola hade också en överrepresentation av DNA-transposoner, som uppgick till ~ 5 % av genomet, jämfört med < 1 % i de andra Alternaria-arterna (fig. 4).

Genomjämförelse av upprepningsinnehållet i sex Alternaria-arter. Bubblornas storlek motsvarar a) procentandel transposabla element (TE) i genomet, b) TE:s kopieringsnummer i genomet
Denna utbredning av repetitivt DNA och den efterföljande evolutionen av gener som överlappar dessa regioner kan vara nyckeln till evolutionär framgång där dessa patogener har lyckats överleva under generationer av samevolutionära konflikter med sina värddjur. Närheten till TE:s utsätter potentiellt generna för repetitionsinducerade punktmutationer (RIP) och därmed för en accelererad utveckling. Ektopisk rekombination mellan liknande TEs kan också resultera i nya kombinationer av gener och därmed öka mångfalden av proteiner eller metaboliter.
Förekomst av en överflödig kromosom hos den storspåriga A. brassicae
Linjespecifika kromosomer (LS) eller överflödiga kromosomer (DC) har rapporterats från flera fytopatogena arter, inklusive A. alternata. DC i A. alternata är kända för att ge isolatet virulens och värdspecificitet. Vid anpassningen av A. brassicae till andra Alternaria spp. i hela arvsmassan visade det sig att en kontig på ca 1 Mb tillsammans med andra mindre kontigs (66-366 kb) var specifik för A. brassicae och att den inte uppvisade någon syntes med någon region i andra Alternaria spp. Däremot observerades en partiell syntes när kontiget anpassades till sekvenser av andra dispenserbara kromosomer som rapporterats hos Alternaria spp. Detta fick oss att anta att dessa contigs tillsammans kan representera en DC av A. brassicae. För att bekräfta detta sökte vi i kontigarna efter förekomst av AaMSAS- och ALT1-generna, som är kända markörgener för dispenserbara kromosomer hos Alternaria spp. Vi hittade två kopior av AaMSAS-genen som en del av två biosyntetiska kluster av sekundära metaboliter på kontigen på 1 Mb. Vi hittade dock ingen homolog till ALT1-genen. Dessutom jämfördes upprepningsinnehållet i contigs (ABRSC11, scaffold 13, 17, 18 och 19) med hela genomet. Geninnehållet i de linjespecifika contigs var betydligt lägre än i kärnkromosomerna (tabell 3). Omvänt var DC-kontiggarna starkt berikade i TE-innehåll jämfört med kärnkromosomerna (tabell 3).
Och även om DC inte var berikad med gener som kodar för sekretoriska proteiner var andelen sekretoriska effektorgener 30 % högre jämfört med kärnkromosomerna. Alla ovanstående bevis pekar på att A. brassicae faktiskt kan hysa en DC. DC i Alternaria spp. har hittills endast rapporterats från småspåriga Alternaria spp. och inga storspåriga Alternaria-arter är kända för att hysa DC. Det återstår att se om DC bidrar till virulensen hos A. brassicae. Framtida studier skulle innefatta karakterisering av den dispenserbara kromosomen hos A. brassicae och korrelera dess närvaro med patogeniciteten hos olika isolat.
Ortologianalys avslöjar artspecifika gener med förmodade roller i virulens
Differenser i geninnehåll och diversitet inom generna bidrar till anpassning, tillväxt och patogenicitet. För att katalogisera skillnaderna i geninnehållet inom Alternaria-släktet och Dothideomyceterna utförde vi en ortologianalys på den kombinerade uppsättningen av 3 60 216 proteiner från 30 olika arter (inklusive 16 Alternaria-arter) som tillhör Dothideomyceterna (Additional file 2: Table S2) varav 3 45 321 proteiner kunde hänföras till minst en av ortogrupperna. Vi identifierade 460 A. brassicae-specifika gener som fanns i A. brassicae men som saknades i alla andra Alternaria-arter (Additional file 3: Table S3). Dessa artspecifika gener omfattade 35 proteinkodande gener för sekretade proteiner, av vilka 11 förutspåddes vara effektorer. Dessutom fanns 20 av dessa artspecifika gener på DC. Ett stort antal av dessa proteiner tillhörde kategorin okarakteriserade proteiner utan känd funktion. För att testa om dessa artspecifika gener är ett resultat av adaptiv evolution som äger rum i genomets repetitionsrika regioner utförde vi ett permutationstest för att jämföra överlappningen av repetitionsrika regioner och transposabla element med en slumpmässig genuppsättning med överlappningen av dessa artspecifika gener. Vi fann att dessa artspecifika gener överlappade signifikant med upprepningsrika regioner (P-värde: 9,99e-05; Z-score: – 4,825) och transposerbara element (P-värde: 0,0460; Z-score: 2,539) i genomet.
Sekundärmetabolitprofil hos A. brassicae och dess samband med transposabla element (TEs)
Släktena Alternaria och Cochliobolus är kända för att vara de största producenterna av värdspecifika sekundärmetabolitgifter. Alternaria spp. är särskilt kända för produktion av kemiskt varierande sekundära metaboliter, som inkluderar värdspecifika toxiner (HST) och icke-HST. Dessa sekundära metaboliter genereras vanligen av icke-ribosomala peptidsynteser (NRPS) och polyketidsynteser (PKS). Vi identifierade fem SM-genkluster av NRPS-typ, 12 genkluster av PKS-typ och sju terpenliknande genkluster i A. brassicae (Additional file 4: Table S4). Av de fem NRPS-klustren kunde vi identifiera tre kluster som producerar kända sekundära metaboliter, nämligen Destruxin B, HC-toxin och dimetylkoprogen (siderofor) med kända roller i virulens.
Genklustret som är ansvarigt för produktionen av dimetylkoprogen (siderofor) i A. brassicae består av 22 gener, inklusive de viktigaste biosyntetiska generna, oxidoreduktaserna och siderofortransportörerna. Siderophorer är järnkällande föreningar som används av svampar för att få extracellulärt järn och har rapporterats vara involverade i svampens virulens. Identifieringen av det genkluster som ansvarar för syntesen av sideroforer skulle göra det möjligt att studera sideroforer och deras roll i patogeniteten hos A. brassicae. Dessutom identifierades ett kluster av PKS-typ bestående av 12 gener som ansvarar för melaninproduktionen (Additional file 4: Table S4). Det melaninbiosyntetiska klustret har tidigare beskrivits för A. alternata. Dessutom har transkriptionsfaktorn Amr1, som inducerar melaninproduktion, karakteriserats i A. brassicicola och är känd för att undertrycka virulens . Melaninets roll för virulens är dock tvetydig och artspecifik .
Växtpatogenerna som tillhör släktet Alternaria verkar ha en dynamisk förmåga att förvärva nya sekundära metaboliter för att kolonisera nya ekologiska nischer. Den mest förenklade förklaringen till detta dynamiska förvärv av sekundärmetabolitpotential är horisontell genöverföring inom släktet Alternaria och eventuellt med andra släkten. Det finns omfattande belägg i litteraturen för att en stor del av de horisontella generna hos Alternaria finns på de överflödiga kromosomerna och att utbyte av dessa kromosomer kan bredda värdspecificiteten. Vi identifierade också ett NRPS-kluster som eventuellt kodar för HC-toxin i en av DC:erna (scaffold 18) (Additional file 4: Table S4). HC-toxin är en känd virulensdeterminant hos växtpatogenen Cochliobolus carbonum, som infekterar majsgenotyper som saknar en funktionell kopia av HM1, ett karbonylreduktas som avgiftar toxinet . En färsk rapport visade att A. jesenskae också kan producera HC-toxin, vilket gör den till den enda andra svampen förutom C. carbonum som producerar toxinet . Närvaron av HC-toxin-genklustret, en virulensdeterminant hos C. carbonum, i en DC av A. brassicae pekar på att horisontell genöverföring mellan arter kan vara vanligare än förväntat.
Avstånd från horisontell genöverföring, snabb duplicering, divergens och förlust av SM-gener kan också bidra till att patogenen utvecklar nya metaboliska förmågor. Dessa duplicerings- och divergensprocesser kan mycket väl underlättas av de sekundära metabolitklustrenas närhet till de upprepade elementen, vilket gör dem känsliga för RIP-mutationer. Därför testade vi om kluster av sekundära metaboliter också var förknippade med upprepningsrika regioner. Ett permutationstest användes för att jämföra överlappningen av repetitionsrika regioner med en slumpmässig genuppsättning med överlappningen av sekundärmetabolitklustergener. De sekundära metabolitklustren överlappade signifikant upprepningsrika regioner jämfört med den slumpmässiga genuppsättningen (P-värde: 0,0017; Z-score: – 2,7963). Dessa kluster överlappade också signifikant med transposabla element bland de repetitionsrika regionerna (P-värde: 0,0087; Z-score: 2,9871). Detta visar att båda de mekanismer som beskrivs ovan för förvärv av ny potential för sekundära metaboliter kan vara möjliga hos A. brassicae. Analyser i populationsskala på art- och släktnivå kan kasta ljus över förekomsten av dessa mekanismer inom Alternaria-släktet.
Syntesanalys avslöjar den genetiska grunden för exklusiviteten i produktionen av Destruxin B hos A. brassicae inom Alternaria-släktet
Destruxin B representerar en klass av cykliska depsipeptider som är känd som en av de viktigaste patogenicitetsfaktorerna hos A. brassicae och har rapporterats vara ett värdspecifikt toxin hos A. brassicae . Destruxin B har inte rapporterats produceras av någon av de andra Alternaria-arterna. Här rapporterar vi för första gången de biosyntetiska genkluster som är ansvariga för produktionen av Destruxin B hos A. brassicae. Klustret består av 10 gener, inklusive det viktigaste biosyntetiska enzymet som kodas av en NRPS-gen (DtxS1) och det hastighetsbegränsande enzymet DtxS3 (aldo-ketoreduktas) (Additional file 4: Table S4). Intressant nog visade syntesanalysen av detta kluster bland de sex Alternaria-arterna att båda dessa gener inte fanns i någon av de andra Alternaria spp. även om klustrets övergripande syntes bibehölls i alla dessa arter (fig. 5). Frånvaron av nyckelgenerna som kodar för enzymerna DtxS1 och DtxS3 i Destruxin B-klustret hos de andra Alternaria-arterna förklarar avsaknaden av Destruxin B hos dessa arter.

Syntesen av Destruxin B-klustret hos de sex Alternaria-arterna. DtxS1 och DtxS3 är markerade i rött (märkta med respektive gen-ID) och saknas i alla andra Alternaria-arter
Destruxin B har rapporterats från entomopatogenen Metarhizium robertsii , och Ophiosphaerella herpotricha , som är orsaken till vårdöd fläck i bermudagräs, förutom från A. brassicae. Wang et al. beskrev det biosyntetiska kluster av sekundära metaboliter som är ansvarigt för produktionen av Destruxin i M. robertsii . Destruxin-klustret i Metarhizhium-arter består av ytterligare två nyckelgener, nämligen DtxS2 och DtxS4. DtxS2 ansvarar för omvandlingen av Destruxin B till Destruxin A, C, D och E. Vi kunde inte hitta någon syntesgen i klustret som motsvarar DtxS2 eller DtxS4 i A. brassicae. Det är känt att A. brassicae endast producerar Destruxin B och inte de andra formerna av Destruxin, vilket möjligen kan förklara avsaknaden av DtxS2 i A. brassicae. DtxS4 är ett aspartatdekarboxylas som krävs för produktion av β-alanin från asparaginsyra och är nödvändigt för produktionen av Destruxins ryggradstruktur. Dessutom kunde deletionsmutanter av DtxS4 producera Destruxin när de kompletterades med β-alanin i mediet . Även om DtxS4 inte finns i Destruxin-klustret har A. brassicae en funktionell kopia av aspartatdekarboxylas som finns någon annanstans i arvsmassan och som därför möjligen kan säkerställa tillförseln av β-alanin för syntesen av Destruxin B. Den evolutionära historiken för produktionen av Destruxin B inom Metarhizhium-släktet liknade i hög grad den för Alternaria. De specialiserade patogenerna, såsom M. acridum, saknar de två nyckelenzymerna och producerar därför inte Destruxiner. M. robertsii, en generalist med ett bredare värdområde, producerar dock Destruxiner . Hypotesen var därför att Destruxiner skulle kunna vara ansvariga för att M. roberstii har ett utvidgat värdområde. En liknande hypotes kan också gälla för A. brassicae, som har ett brett värdområde och kan infektera nästan alla Brassicaceae. Ytterligare experiment för att bestämma värdområdet för Destruxin-nollmutanter av A. brassicae kan bidra till att klargöra den roll som detta viktiga fytotoxin spelar för att utvidga värdområdet för A. brassicae.
Distinct CAZyme profiles of A. brassicae and A. brassicicola within the Alternaria genus
CAZymes (Carbohydrate-Active enZymes) är proteiner som är involverade i nedbrytningen, omarrangemanget eller syntesen av glykosidiska bindningar. Växtpatogener utsöndrar en mängd olika CAZymes som bryter ner de komplexa polysackariderna i växternas cellväggar. De består av fem olika klasser, nämligen glykosidhydrolaser (GH), glykosyltransferaser (GT), polysackaridlyaser (PL), kolhydratesteraser (CE) och kolhydratbindande moduler (CBM). Vi identifierade > 500 CAZymes i de sex Alternaria spp. inklusive A. brassicae (Additional file 5: Table S5). CAZymfördelningen hos A. brassicae och A. brassicicola varierade från de andra Alternaria spp. och bildar därmed ett separat kluster (fig. 6). Antalet enzymer med hjälpaktivitet eller de enzymer som är involverade i nedbrytning av växtcellväggar varierade avsevärt mellan de olika jämförda arterna. Nästan 46 % av CAZymerna i A. brassicae utsöndrades varav ~ 17 % förutspåddes vara effektorer.

Heatmap över CAZyme-profilerna i 16 arter med Alternaria-släktet. Organismernas klusterbildning liknar i hög grad den i artfylogenin utom A. brassicae och A. brassicicola, som klustrar separat med outgroupen Stemphylium lycopersici. CAZymerna grupperades enligt CAZy-klassificeringen
Familjen AA9 (tidigare GH61; kopparberoende lytiska polysackaridmonooxygenaser (LPMO)) är betydligt förstorad jämfört med de andra CAZy-familjerna i Alternaria-släktet med varje art som innehåller > 20 kopior av genen. Antalet kopior i Alternaria spp. är mycket högre än det antal kopior som rapporterats för Botrytis och Fusarium spp. AA9-familjen är involverad i nedbrytningen av cellväggens polysackarider och är känd för att verka på en rad polysackarider, inklusive stärkelse, xyloglukan, cellodextriner och glukomannan. Man har antagit att LPMO:erna har en dubbel roll – dels att de direkt klyver cellväggens polysackarider, dels att de agerar som ROS-generator och därmed bidrar till den oxidativa stress som leder till nekros i växtvävnaden. Det är slående att 11 av de 26 AA9-proteiner som finns i A. brassicae förutspås vara sekretoriska effektorer. Karaktärisering av dessa CAZymes och deras roll i patogenesen kan bli föremål för ytterligare studier.
Överlappande effektorprofiler inom Alternaria-släktet
Vi förutspådde effektorrepertoaren hos sex Alternaria-arter (tabell 2), inklusive A. brassicae, med hjälp av den pipeline som beskrivs i avsnittet om metoder. De flesta av de små sekretoriska proteiner som förutspåddes vara effektorer var okaraktäriserade/förutsägda proteiner och kan därför vara nya art- eller genusspecifika effektorer. A. brassicae hade den största andelen okända/förutsagda/hypotetiska proteiner i uppsättningen av potentiella effektorer. Vi fann att de flesta effektorer mellan de sex arterna var gemensamma och överlappande, vilket tyder på att de breda mekanismerna för patogenes som involverar proteinskapande effektorer kan vara bevarade inom släktet. Vi fann dock att två kopior av ett protein som innehåller ett beta/gamma-kristallinveck fanns endast hos A. brassicae och A. brassicicola och inte hos de andra fyra Alternaria-arterna. En ytterligare sökning i NCBI:s nr-databas visade att detta protein helt saknades i Alternaria-släktet och att de närmaste överensstämmelserna hittades i andra växtpatogener, nämligen Macrophomina- och Fusarium-arter. Det finns dock ingen information om dess funktion eller roll i patogeniteten hos någon av arterna.
Vi kunde också fastställa att vissa av effektorerna i A. brassicae har potential att utvecklas adaptivt eftersom de också var signifikant förknippade med de upprepningsrika regionerna i genomet (P-värde: 0,0003; Z-score: – 2,8778). Det krävs därför analyser på populationsnivå för att identifiera de effektorer som utsätts för positivt urval, vilket skulle kunna belysa evolutionen av patogenicitet hos A. brassicae. De effektorer som identifierades i den här studien avslöjar det breda spektrum av proteiner som kan vara involverade i A. brassicaes patogenes. 39 av dessa effektorer förutspåddes vara CAZymes som har olika roller i nedbrytningen av cellväggen och tillhörande polysackarider. Genomet hos A. brassicae innehöll två nekros- och etyleninducerande peptidproteiner, som har varit inblandade i patogenesen hos olika patogener, inklusive oomyceter och nekrotrofer . Hrip1 (Hypersensitive response inducing protein 1) från A. alternata har nyligen visats kännas igen av MdNLR16 på ett klassiskt gen-för-gen-sätt, och borttagning av Hrip1 från A. alternata ökar dess virulens . En Hrip1-homolog finns också i A. brassicae, men den förväntas inte utsöndras utanför cellen, även om detta måste verifieras experimentellt. Förekomsten av effektorer som känns igen på ett gen-för-gen-sätt öppnar möjligheten att identifiera kompletterande R-gener i värden som kan användas för att utveckla resistenta sorter eller odlingar.