Genomické vlastnosti A. brassicae a dvou dalších souběžně infikujících izolátů A. alternata
Sekvenovali jsme genomy dvou izolátů A. alternata (PN1 a PN2), které společně s A. brassicae infikovaly B. juncea. Sestava A. brassicae byla popsána již dříve . Stručně řečeno, sestava se skládala z devíti kompletních chromozomů a jednoho chromozomu s chybějícími telomerickými repeticemi na jednom z konců. Kromě těchto chromozomů existovalo šest kontigů, z nichž jeden měl velikost ~ 1 Mb, které mohou dohromady tvořit postradatelný chromozom (obr. 1). N50 sestavy A. brassicae byla 2,98 Mb (tab. 1). Dva izoláty spoluinfikující B. juncea byly na základě sekvencí ITS a GAPDH identifikovány jako A. alternata. Sestavy A. alternata Aat_PN1 a Aat_PN2 se skládaly ze 14 kontigů o celkové velikosti 33,77 Mb, resp. 15 kontigů o celkové velikosti 33,53 Mb (tabulka 1). Šest kontigů v každé z obou sestav obsahovalo telomerické repetice na obou koncích, a proto s největší pravděpodobností představují celé chromozomální molekuly. Čtyři další kontigy v obou sestavách obsahovaly telomerické repetice na jednom konci, ale měly podobnou velikost úplných chromozomových molekul, jak bylo popsáno u A. solani . Proto sestavy genomů izolátů A. alternata představovaly deset téměř úplných chromozomů každého z obou izolátů.
Zarovnání celých genomů s příbuznými Alternaria spp. ukázala celkovou syntézu mezi genomy s menšími přeskupeními (obr. 2). Kromě toho byly ze sekvenačních dat pro dva izoláty A. alternata získány také mitochondriální sekvence. Mitochondriální genomy kmenů A. alternata měly velikost přibližně 49 783 bp a 50 765 bp a vykazovaly vysokou podobnost s dříve publikovaným mitochondriálním genomem A. alternata .
Předpověď genů po maskování opakování vedla k identifikaci 11593, 11495 a 11387 genů v sestavách genomu A. brassicae, A. alternata PN1 a PN2. To bylo srovnatelné s počtem genů odhadnutým u jiných druhů rodu Alternaria (tabulka 1). BUSCO analýza ukázala, že genové modely předpovězené ve třech genomech pokrývají 98 % konzervovaných genů hub s jednou kopií, což ukazuje na téměř úplnost sestav. Předpovězené geny byly komplexně anotovány pomocí kombinace databází, jak je popsáno v části Metody (obr. 1). Kromě těchto tří genomů jsme také předpověděli geny de novo v sestavách genomů tří dalších druhů Alternaria, které byly sekvenovány pomocí technologií dlouhých čtení, a to A. brassicicola (abra43), A. alternata (ATCC34957) a A. solani (altNL03003) (tabulka 1). Těchto šest genomů a jejich genové predikce byly použity pro srovnávací analýzy genových klastrů kódujících sekundární metabolity a efektorové geny.

Souhrn genů A. brassicae genomu, (od vnější po vnitřní kruhovou stopu) a pseudochromozomy/skládky, b geny kódující proteiny, c opakující se elementy, d transponovatelné elementy (DNA a LTR), e předpokládané klastry sekundárních metabolitů, f sekretované proteiny, g předpokládané efektory
Fylogenomická analýza přiřazuje samostatný klad pro brassicae infikující A. brassicae a A. brassicicola v rámci rodu Alternaria
Pro přesnou rekonstrukci divergence a vztahů mezi A. brassicae, dvěma izoláty A. alternata (PN1 a PN2) a ostatními druhy rodu Alternaria jsme provedli fylogenomickou analýzu s použitím 29 ortologů s jednou kopií, které měly nejvyšší fylogenetický signál vypočítaný programem Mirlo. Výběr genů s vyššími fylogenetickými signály vede k fylogenezi, která se více shoduje s druhovým stromem . Výsledná fylogeneze ukázala, že druhy Alternaria s velkými výtrusy a Alternaria s malými výtrusy se shlukují odděleně do dvou různých kladů (obr. 3). Zajímavé je, že dva hlavní patogeny brukvovitých, a to A. brassicae a A. brassicicola, se shlukovaly odděleně od všech ostatních druhů rodu Alternaria, což pravděpodobně naznačuje odlišnou evoluční trajektorii založenou na společných hostitelských preferencích těchto dvou druhů.
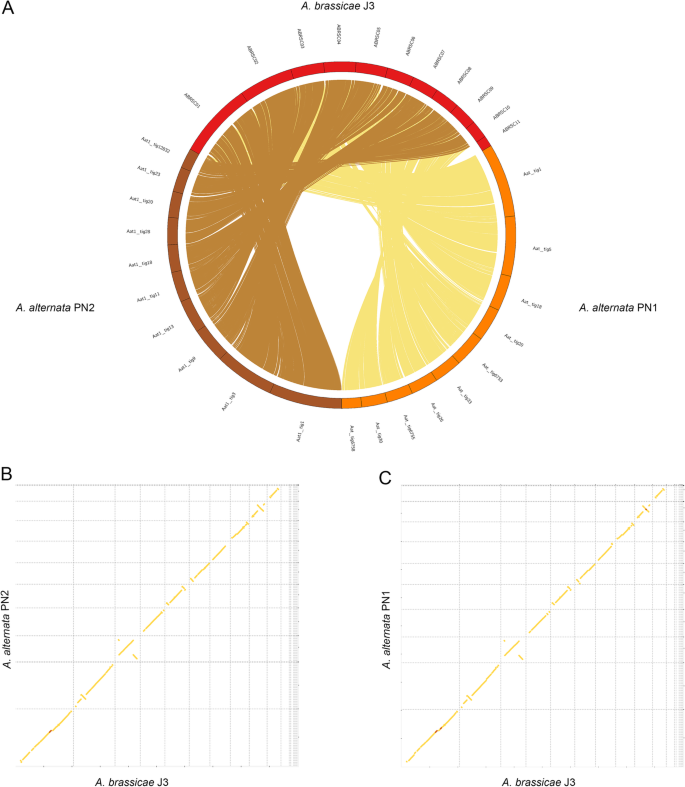
Celogenomové zarovnání A. alternata PN1 a PN2 s A. brassicae. a Circosův graf zobrazující makrosyntézu A. alternata PN1 a PN2 s A. brassicae napříč všemi kontigy kromě dispenzabilních kontigů (ABRSC11, scaffold13,17,18,19), b a c Syntenic dotplots of A. brassicae with A. alternata PN1 and PN2
Srovnávací analýzy A. alternata získaných z různých hostitelů
Srovnávali jsme genomy A. alternata PN1 a PN2 (izolovaných z B. juncea) s genomem A. alternata ATCC34957 (izolovaného z čiroku), abychom zjistili případné rozdíly v jejich genomickém obsahu, které by jim umožnily infikovat dva velmi odlišné druhy. Zarovnání celých genomů A. alternata PN1 a PN2 s genomem A. alternata ATCC34957 odhalilo velmi vysokou úroveň syntézy a absenci jakýchkoli druhově specifických oblastí. Mezi třemi izoláty A. alternata jsme identifikovali 719, 152 a 586 izolátově specifických genů (doplňkový soubor 1: tabulka S1). Více než dvě třetiny izolátově specifických genů u všech tří izolátů byly necharakterizované proteiny nebo neměly žádné anotace. Pozoruhodné je, že všechny tři izoláty neobsahovaly žádné dispenzibilní chromozomy, které by mohly propůjčovat patogenitu, jak bylo zaznamenáno u izolátů A. alternata infikujících mnoho ovocných plodin, jako jsou citrusy, hrušky a jabloně . Genový repertoár tří izolátů se rovněž skládal z podobného počtu a typu efektorů, CAZymů a skupin sekundárních metabolitů (tabulka 2). Dva izoláty PN1 a PN2 navíc samy o sobě nevyvolávají příznaky infekce u B. juncea v epifytických podmínkách (údaje nejsou uvedeny). Naše výsledky naznačují, že tyto izoláty A. alternata (PN1 a PN2) mohou být fakultativními patogeny, které vedou saprofytický způsob života a za určitých podmínek prostředí mohou přejít na patogenní způsob života.
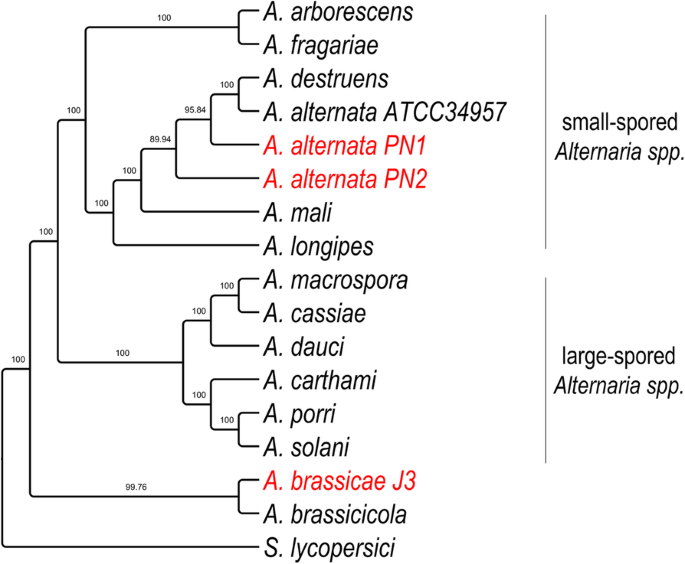
Fylogenetický strom druhů Alternaria s S. lycopersici jako outgroup. Strom byl sestaven s použitím 29 ortologů s jednou kopií, které měly nejvyšší fylogenetický signál vypočtený v programu Mirlo. Jsou uvedeny hodnoty podpory větví z 1000 bootstrapových replikátů
Hojný výskyt oblastí bohatých na repetice a transponovatelných elementů u A. brassicae
Vláknité rostlinné patogeny mají tendenci mít odlišnou architekturu genomu s vyšším obsahem opakování. Odhad obsahu opakování a maskování pomocí RepeatModeler a RepeatMasker odhalil, že genom A. brassicae se skládá z ~ 9,33 % opakování ve srovnání s 2,43 a 2,64 % opakování v genomech A. alternata. Genom A. brassicae obsahuje nejvíce repetic (~ 9,33 %) ze všech dosud sekvenovaných druhů rodu Alternaria. Naše analýza ukázala, že obsah repetic se výrazně liší mezi izoláty A. alternata a ostatními patogenními druhy rodu Alternaria. Patogenní druhy Alternaria, zejména A. brassicae a A. brassicicola, měly výrazně větší repertoár LTR/Gypsy a LTR/Copia elementů (> 8X) ve srovnání s ostatními izoláty A. alternata (patogenními i nepatogenními) (obr. 4). Genomy A. brassicae a A. brassicicola měly také nadměrné zastoupení DNA transpozonů, které činilo ~ 5 % genomu ve srovnání s < 1 % u ostatních druhů Alternaria (obr. 4).

Srovnání obsahu repetic u šesti druhů Alternaria. Velikost bublin odpovídá a) procentuálnímu zastoupení transponovatelných elementů (TE) v genomu, b počtu kopií TE v genomu
Toto rozmnožení repetitivní DNA a následná evoluce genů překrývajících tyto oblasti může být klíčem k evolučnímu úspěchu, kdy se těmto patogenům podařilo přetrvat po generace v koevolučním konfliktu se svými hostiteli. Blízkost TE potenciálně vystavuje geny opakováním indukovaným bodovým mutacím (RIP), a tím urychluje evoluci . Ektopická rekombinace mezi podobnými TE může také vést ke vzniku nových kombinací genů, a tím ke zvýšení diverzity proteinů nebo metabolitů.
Přítomnost dispenzibilního chromozomu u velkoplodé A. brassicae
Chromozomy specifické pro linii (LS) nebo dispenzibilní chromozomy (DC) byly zaznamenány u několika fytopatogenních druhů včetně A. alternata. Je známo, že DC u A. alternata propůjčují izolátu virulenci a hostitelskou specifitu. Zarovnání celého genomu A. brassicae s jinými druhy Alternaria spp. ukázalo, že kontig o velikosti přibližně 1 Mb spolu s dalšími menšími kontigy (66-366 kb) je specifický pro A. brassicae a nevykazuje syntézu s žádnou oblastí u jiných druhů Alternaria spp. Nicméně částečná syntéza byla pozorována, když byl kontig zarovnán se sekvencemi jiných dispenzibilních chromozomů uváděných u Alternaria spp. To nás vedlo k hypotéze, že tyto kontigy mohou společně představovat DC A. brassicae. Abychom to potvrdili, hledali jsme v kontigenech přítomnost genů AaMSAS a ALT1, což jsou známé markerové geny pro dispenzabilní chromozomy u Alternaria spp. Našli jsme dvě kopie genu AaMSAS jako součást dvou biosyntetických klastrů sekundárních metabolitů na 1 Mb kontigu. Nenalezli jsme však žádný homolog genu ALT1. Dále byl porovnán obsah opakování v kontigu (ABRSC11, scaffold 13, 17, 18 a 19) s celým genomem. Obsah genů v kontigu specifickém pro danou linii byl výrazně nižší než v jádrových chromozomech (tabulka 3). Naopak kontigy DC byly ve srovnání s jádrovými chromozomy vysoce obohaceny o obsah TE (tabulka 3).
Ačkoli DC nebyl obohacen o geny kódující vylučované proteiny, podíl vylučovaných efektorových genů byl ve srovnání s jádrovými chromozomy o 30 % vyšší. Všechny výše uvedené důkazy ukazují na skutečnost, že A. brassicae může skutečně skrývat DC. DC u Alternaria spp. byly dosud zaznamenány pouze u malosporulujících Alternaria spp. a není známo, že by se u velkosporulujících druhů Alternaria vyskytovaly DC. Zbývá zjistit, zda DC přispívá k virulenci A. brassicae. Budoucí studie by měly zahrnovat charakterizaci dispenzibilního chromozomu u A. brassicae a korelaci jeho přítomnosti s patogenitou různých izolátů.
Orthologická analýza odhaluje druhově specifické geny s předpokládanou rolí ve virulenci
Rozdíly v obsahu genů a diverzita uvnitř genů přispívají k adaptaci, růstu a patogenitě. Abychom katalogizovali rozdíly v obsahu genů v rámci rodu Alternaria a Dothideomycetes, provedli jsme ortologickou analýzu kombinovaného souboru 3 60 216 proteinů z 30 různých druhů (včetně 16 druhů Alternaria) patřících do Dothideomycetes (doplňkový soubor 2: tabulka S2), z nichž 3 45 321 proteinů bylo možné přiřadit alespoň k jedné z ortoskupin. Identifikovali jsme 460 genů specifických pro A. brassicae, které byly přítomny u A. brassicae, ale chyběly u všech ostatních druhů Alternaria (Additional file 3: Table S3). Tyto druhově specifické geny zahrnovaly 35 genů kódujících sekreční proteiny, z nichž 11 bylo předpovězeno jako efektory. Kromě toho bylo 20 z těchto druhově specifických genů přítomno na DC. Velký počet těchto proteinů patřil do kategorie necharakterizovaných proteinů bez známé funkce. Abychom otestovali, zda jsou tyto druhově specifické geny výsledkem adaptivní evoluce probíhající v oblastech genomu bohatých na repetice, provedli jsme permutační test, abychom porovnali překrytí oblastí bohatých na repetice a transpozibilních elementů s náhodným souborem genů s překrytím těchto druhově specifických genů. Zjistili jsme, že tyto druhově specifické geny se významně překrývají s oblastmi bohatými na opakování (hodnota P: 9,99e-05; Z-skóre: – 4,825) a transponovatelnými elementy (hodnota P: 0,0460; Z-skóre: 2,539) v genomu.
Profil sekundárních metabolitů A. brassicae a jeho souvislost s transponovatelnými elementy (TE)
Rody Alternaria a Cochliobolus jsou známy jako hlavní producenti hostitelsky specifických sekundárních metabolitů toxinů. Zejména Alternaria spp. jsou známy produkcí chemicky rozmanitých sekundárních metabolitů, které zahrnují hostitelsky specifické toxiny (HST) a non-HST. Tyto sekundární metabolity jsou obvykle generovány neribozomálními peptidovými syntázami (NRPS) a polyketidovými syntázami (PKS). U A. brassicae jsme identifikovali pět genových klastrů typu NRPS SM, 12 genových klastrů typu PKS a sedm genových klastrů podobných terpenům (Additional file 4: Table S4). Z pěti shluků NRPS jsme mohli identifikovat tři shluky, které produkují známé sekundární metabolity, tj. destruxin B, HC-toxin a dimetylkoprogen (siderofor) se známou rolí ve virulenci.
Genový shluk odpovědný za produkci dimetylkoprogenu (sideroforu) u A. brassicae se skládá z 22 genů, včetně hlavních biosyntetických genů, oxidoreduktáz a přenašečů sideroforu. Siderofory jsou sloučeniny chelatující železo, které houby využívají k získávání extracelulárního železa a které se podle zpráv podílejí na virulenci hub. Identifikace shluku genů odpovědných za syntézu sideroforů by umožnila studium sideroforů a jejich úlohy v patogenitě u A. brassicae. Kromě toho byl identifikován také klastr typu PKS sestávající z 12 genů, který je zodpovědný za produkci melaninu (Additional file 4: Table S4). Melaninový biosyntetický klastr byl u A. alternata popsán již dříve . Také transkripční faktor Amr1, který indukuje produkci melaninu, byl charakterizován u A. brassicicola a je známo, že potlačuje virulenci . Úloha melaninu ve virulenci je však nejednoznačná a druhově specifická .
Zdá se, že rostlinné patogeny patřící do rodu Alternaria mají dynamickou schopnost získávat nový potenciál sekundárních metabolitů pro kolonizaci nových ekologických nik. Nejjednodušším vysvětlením tohoto dynamického získávání potenciálu sekundárních metabolitů je horizontální přenos genů v rámci rodu Alternaria a případně s jinými rody. V literatuře existují rozsáhlé důkazy o tom, že velká část HST Alternaria je nesena na dispenzibilních chromozomech a výměna těchto chromozomů může rozšířit hostitelskou specifitu . Identifikovali jsme také klastr NRPS, který pravděpodobně kóduje HC-toxin v jednom z DC (scaffold 18) (Additional file 4: Table S4). HC-toxin je známým determinantem virulence rostlinného patogenu Cochliobolus carbonum, který infikuje genotypy kukuřice postrádající funkční kopii HM1, karbonylreduktázy, která tento toxin detoxikuje . Nedávná zpráva ukázala, že A. jesenskae může rovněž produkovat HC-toxin, což z ní činí jedinou další houbu kromě C. carbonum, která tento toxin produkuje . Přítomnost shluku genů pro HC-toxin, determinantu virulence u C. carbonum, v DC A. brassicae poukazuje na skutečnost, že mezidruhový horizontální přenos genů může být častější, než se očekávalo.
Kromě horizontálního přenosu genů může k vývoji nových metabolických schopností patogenu přispívat také rychlá duplikace, divergence a ztráta SM genů. Tyto procesy duplikace a divergence mohou být dobře podpořeny blízkostí shluků sekundárních metabolitů k opakujícím se elementům, která je činí náchylnými k RIP-mutacím. Proto jsme testovali, zda jsou klastry sekundárních metabolitů také spojeny s oblastmi bohatými na repetice. K porovnání překryvu oblastí bohatých na opakování s náhodným souborem genů s překryvem genů klastrů sekundárních metabolitů byl použit permutační test. Klastry sekundárních metabolitů se významně překrývaly s oblastmi bohatými na opakování ve srovnání s náhodným souborem genů (hodnota P: 0,0017; Z-skóre: – 2,7963). Tyto shluky se také významně překrývaly s transpozibilními elementy mezi oblastmi bohatými na opakování (hodnota P: 0,0087; Z-skóre: 2,9871). To ukazuje, že oba výše popsané mechanismy získávání potenciálu nových sekundárních metabolitů mohou být v případě A. brassicae možné. Analýzy v populačním měřítku na úrovni druhů a rodů mohou vrhnout světlo na převahu těchto mechanismů v rámci rodu Alternaria.
Syntetická analýza odhaluje genetický základ exkluzivity produkce destruxinu B u A. Brassica. brassicae v rámci rodu Alternaria
Destruxin B představuje třídu cyklických depsipeptidů, o nichž je známo, že jsou jedním z klíčových faktorů patogenity A. brassicae a že jsou hostitelsky specifickým toxinem A. brassicae . Destruxin B nebyl hlášen u žádného z ostatních druhů rodu Alternaria. Zde poprvé uvádíme biosyntetické genové klastry odpovědné za produkci destruxinu B u A. brassicae. Klastr se skládá z 10 genů, včetně hlavního biosyntetického enzymu kódovaného genem NRPS (DtxS1) a enzymem omezujícím rychlost, DtxS3 (aldo-keto reduktasa) (Additional file 4: Table S4). Zajímavé je, že analýza syntézy tohoto klastru mezi šesti druhy Alternaria ukázala, že oba tyto geny nebyly přítomny u žádného z ostatních druhů Alternaria, ačkoli celková syntéza klastru byla u všech těchto druhů zachována (obr. 5). Nepřítomnost klíčových genů kódujících enzymy DtxS1 a DtxS3 v klastru Destruxin B u ostatních druhů Alternaria vysvětluje nepřítomnost Destruxinu B u těchto druhů.

Syntéza klastru Destruxin B u šesti druhů Alternaria. DtxS1 a DtxS3 jsou vyznačeny červeně (označeny příslušnými ID genů) a chybí u všech ostatních druhů rodu Alternaria
Destruxin B byl kromě A. brassicae zaznamenán u entomopatogenu Metarhizium robertsii , a Ophiosphaerella herpotricha , původce jarní mrtvolné skvrnitosti bobovitých trav. Wang a kol. popsali biosyntetický klastr sekundárních metabolitů odpovědný za produkci destruxinu u M. robertsii . Destruxinový klastr u druhu Metarhizhium se skládá ze dvou dalších klíčových genů, a to DtxS2 a DtxS4. DtxS2 je zodpovědný za přeměnu Destruxinu B na Destruxin A, C, D a E. V klastru jsme nenašli žádný syntenický gen, který by odpovídal DtxS2 nebo DtxS4 u A. brassicae. Je známo, že A. brassicae produkuje pouze Destruxin B a ne ostatní formy Destruxinu, což by mohlo vysvětlovat absenci DtxS2 u A. brassicae. DtxS4 je aspartátdekarboxyláza, která je nezbytná pro produkci β-alaninu z kyseliny asparagové a je nezbytná pro produkci páteřní struktury destruxinu. Navíc deleční mutanti DtxS4 byli schopni produkovat Destruxin, když jim byl do média přidán β-alanin. Přestože DtxS4 není přítomen v Destruxinovém klastru, A. brassicae má jednu funkční kopii aspartátdekarboxylázy přítomnou na jiném místě genomu, a proto může pravděpodobně zajistit přísun β-alaninu pro syntézu Destruxinu B. Evoluční historie produkce Destruxinu B v rámci rodu Metarhizhium se velmi podobala historii Alternaria. Specializované patogeny, jako je M. acridum, postrádají oba klíčové enzymy, a proto neprodukují destruxiny. M. robertsii, generalista s širším hostitelským spektrem, však Destruxiny produkuje. Proto byla vyslovena hypotéza, že Destruxiny mohou být zodpovědné za rozšíření hostitelského areálu M. roberstii. Podobná hypotéza může platit i v případě A. brassicae, která má široký hostitelský areál a může infikovat téměř všechny Brassicaceae. Další experimenty zaměřené na určení hostitelského areálu Destruxin null mutantů A. brassicae mohou pomoci objasnit roli tohoto důležitého fytotoxinu v rozšíření hostitelského areálu A. brassicae.
Rozlišné CAZymové profily A. brassicae a A. brassicicola v rámci rodu Alternaria
CAZymes (Carbohydrate-Active enZymes) jsou proteiny zapojené do degradace, přeskupování nebo syntézy glykosidických vazeb. Rostlinné patogeny vylučují rozmanitou škálu CAZymů, které rozkládají složité polysacharidy v rostlinné buněčné stěně. Skládají se z pěti různých tříd, a to z glykosidhydrolas (GH), glykosyltransferas (GT), polysacharidlyas (PL), karbohydrátesteras (CE) a modulů vázajících karbohydráty (CBM). U šesti druhů Alternaria včetně A. brassicae jsme identifikovali > 500 CAZymů (Additional file 5: Table S5). Distribuce CAZymů u A. brassicae a A. brassicicola se lišila od distribuce u ostatních Alternaria spp. a tvořila tak samostatný klastr (obr. 6). Počet enzymů s pomocnou aktivitou nebo enzymů podílejících se na degradaci rostlinné buněčné stěny se mezi jednotlivými porovnávanými druhy značně lišil. Téměř 46 % CAZymů u A. brassicae bylo vylučováno, z toho ~ 17 % bylo předpokládáno jako efektory.

Heatmapa profilů CAZymů u 16 druhů s rodem Alternaria. Shlukování organismů se velmi podobá fylogenezi druhů s výjimkou A. brassicae a A. brassicicola, které se shlukují samostatně s outgroup Stemphylium lycopersici. CAZy byly seskupeny podle klasifikace CAZy
Rodina AA9 (dříve GH61; měď dependentní lytické polysacharidové monooxygenázy (LPMO)) je ve srovnání s ostatními rodinami CAZy v rodu Alternaria výrazně rozšířená, přičemž každý druh obsahuje > 20 kopií genu. Počty kopií u Alternaria spp. jsou mnohem vyšší než počty kopií uváděné u Botrytis a Fusarium spp. Rodina AA9 se podílí na rozkladu polysacharidů buněčné stěny a je známo, že působí na řadu polysacharidů včetně škrobu, xyloglukanu, celulodextrinů a glukomannanu. Předpokládá se, že LPMO mají dvojí úlohu – jednak přímo štěpí polysacharidy buněčné stěny, jednak působí jako generátor ROS, a tím přispívají k oxidačnímu stresu vedoucímu k nekróze v rostlinných pletivech. Je pozoruhodné, že 11 z 26 proteinů AA9 přítomných u A. brassicae jsou předpokládané sekretované efektory. Charakterizace těchto CAZymů a jejich role v patogenezi by mohla být předmětem dalších studií.
Překrývající se profily efektorů v rámci rodu Alternaria
Předpověděli jsme repertoár efektorů šesti druhů Alternaria (tabulka 2) včetně A. brassicae pomocí pipeline popsané v části o metodách. Většina malých vylučovaných proteinů předpovězených jako efektory byly necharakterizované/předpovězené proteiny, a proto se může jednat o nové druhově nebo rodově specifické efektory. A. brassicae měl v souboru kandidátních efektorů největší podíl neznámých/předpokládaných/hypotetických proteinů. Zjistili jsme, že většina efektorů mezi šesti druhy je společná a překrývá se, což naznačuje, že široké mechanismy patogeneze zahrnující proteinové efektory mohou být v rámci rodu konzervovány. Zjistili jsme však, že dvě kopie proteinu obsahujícího beta/gama krystalinovou složku jsou přítomny pouze u A. brassicae a A. brassicicola a nikoli u ostatních čtyř druhů rodu Alternaria. Další vyhledávání v databázi nr NCBI ukázalo, že tento protein v rodu Alternaria zcela chybí a nejbližší shody byly nalezeny u jiných rostlinných patogenů, tj. u druhů Macrophomina a Fusarium. Nejsou však k dispozici žádné informace o jeho funkci nebo roli v patogenitě u žádného z druhů.
Mohli jsme také zjistit, že některé efektory u A. brassicae mají potenciál vyvíjet se adaptivně, protože byly také významně spojeny s oblastmi genomu bohatými na opakování (P-hodnota: 0,0003; Z-skóre: – 2,8778). K identifikaci efektorů pod pozitivní selekcí, která by mohla objasnit evoluci patogenity u A. brassicae, jsou proto nutné analýzy na úrovni populace. Efektory identifikované v této studii odhalují širokou škálu proteinů, které se mohou podílet na patogenezi A. brassicae. Předpokládá se, že 39 z těchto efektorů jsou CAZymy, které mají různé úlohy při rozkladu buněčné stěny a souvisejících polysacharidů. Genom A. brassicae obsahoval dva peptidové proteiny indukující nekrózu a etylen, které byly zapojeny do patogeneze různých patogenů včetně oomycet a nekrotrofů . Nedávno bylo prokázáno, že Hrip1 (Hypersensitive response inducing protein 1) z A. alternata je rozpoznáván MdNLR16 klasickým způsobem gen za gen a delece Hrip1 z A. alternata zvyšuje jeho virulenci . Homolog Hrip1 je přítomen také u A. brassicae, ale nepředpokládá se, že by byl vylučován mimo buňku, i když to je třeba experimentálně ověřit. Přítomnost efektorů, které jsou rozpoznávány způsobem gen za gen, otevírá možnost identifikace komplementárních R-genů v hostiteli, které lze využít pro vývoj rezistentních odrůd nebo kultivarů.
.