Genomic features of A. Bassicae. brassicaeと他の2つの共感染A. alternata分離株
我々はB. junceaにA. brassicaeと共感染したA. alternata分離株2株(PN1、PN2)のゲノムを配列決定した。 A. brassicaeのアセンブリについては既報の通りである. この染色体は、9本の完全な染色体と、テロメアリピートの末端が欠落している1本の染色体から構成されている。 これらの染色体とは別に、6本のコンティグが存在し、そのうち1本は1Mb程度の大きさであることから、これらは合わせて不要な染色体である可能性がある(図1)。 A. brassicaeのアセンブリのN50は2.98 Mbであった(表1)。 B. junceaに共存する2株は、ITSとGAPDHの配列からA. alternataであると同定された。 A. alternataのアセンブリAat_PN1とAat_PN2は、それぞれ14コンティグ(合計33.77Mb)と15コンティグ(合計33.53Mb)で構成されていた(表1)。 2つの集合体のそれぞれで6つのコンティグが両端にテロメアリピートを含んでおり、完全な染色体分子である可能性が最も高いと考えられる。 また、他の4つのコンティグは片方の端にテロメアリピートを持つが、A. solaniで報告されているような完全な染色体分子と同じ大きさであった。
Alternaria spp.と全ゲノムアラインメントしたところ、ゲノム間には小さな再配列があるが全体としてシンテニーを示した (Fig.2). さらに、A. alternataの2つの分離株のシークエンスデータからミトコンドリア配列も取得した。 A. alternata株のミトコンドリアゲノムはそれぞれ約49,783 bpと50,765 bpの大きさで、これまでに発表されたA. alternataのミトコンドリアゲノムと高い類似性を示した。
リピートマスキング後の遺伝子予測により、A. brassicae, A. alternata PN1, PN2ゲノム集合体にそれぞれ11593、11495、11387個の遺伝子が同定された。 これは、他のAlternaria属で推定される遺伝子数と同程度であった(表1)。 BUSCO解析の結果、3つのゲノムで予測された遺伝子モデルは、1コピー保存された真菌遺伝子の98%をカバーしており、アセンブリがほぼ完全であることが示された。 予測された遺伝子は、方法論の項で述べたように、データベースを組み合わせて包括的なアノテーションを行った(図1)。 この3種のゲノムに加え、ロングリード技術で配列決定された他の3種のAlternaria、すなわちA. brassicicola (abra43), A. alternata (ATCC34957), A. solani (altNL03003) のゲノムアセンブリにおいてもde novo遺伝子を予測した(Table 1)。 これらの6つのゲノムとその遺伝子予測を用いて、二次代謝産物をコードする遺伝子群やエフェクターをコードする遺伝子の比較解析を行った。 brassicaeゲノム概要、(外側の円形トラックから内側の円形トラックへ)a 擬染色体・足場、b タンパク質コード遺伝子、c 繰り返し要素、d トランスポーザブル・エレメント(DNAおよびLTR)、e 予測二次代謝物クラスター、f 分泌タンパク質、g 予想エフェクター
系統解析により別系統が割り当てられA Brassica感染型 A. brassicaeは、そのうちの1つの系統である。 brassicaeとA. brassicicolaのAlternaria属内での分岐と関係を正確に再構築するために、プログラムMirloで計算した最も高い系統的シグナルを持つ29の単一コピーオルソログを使用して系統解析を実施しました。 系統発生シグナルの高い遺伝子を選択することで、より種樹に一致した系統を導き出すことができた 。 その結果、大胞子性アルテルナリアと小胞子性アルテルナリアは、2つの異なるクレードに分かれていることがわかった(図3)。 興味深いことに、アブラナ科植物の主要病原菌であるA. brassicaeとA. brassicicolaは、他のすべてのAlternaria種とは別にクラスタリングされており、おそらくこれら2種の共通の宿主選好性に基づく異なる進化の軌跡を示していると思われる。
Fig. 2
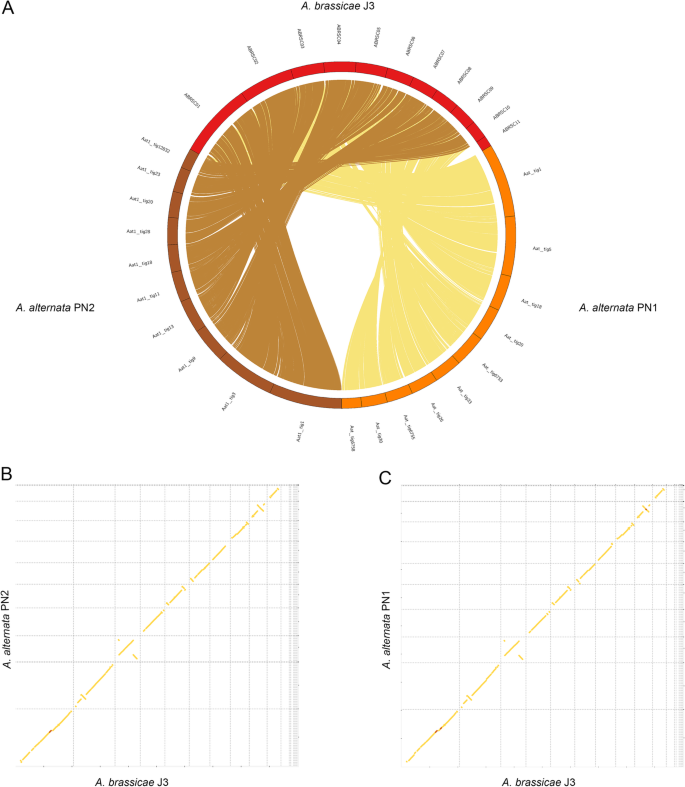
A. alternata PN1 および PN2 と A. brassicae のホールゲノム・アラインメント。 A. alternata PN1およびPN2とA. brassicaeのマクロシンテンシーを示すCircos plot、dispensable contig (ABRSC11, scaffold13,17,18,19) を除くすべてのcontig、bおよびc Syntenic dotplot A. brassicae と A. alternata PN1 および PN2
Comparing analyses of A. A. brassicae, A. alternata PN1 および PN2 の比較研究。 alternata PN1およびPN2(B. junceaから分離)とA. alternata ATCC34957(ソルガムから分離)のゲノムを比較し、これらが全く異なる2つの種に感染することを可能にするかもしれないゲノム内容の相違を明らかにした。 A. alternata PN1およびPN2とA. alternata ATCC34957の全ゲノム配列を比較したところ、非常に高いシンテニーを示し、種特異的な領域は存在しないことが判明した。 また、A. alternataの3つの分離株の間で、それぞれ719、152、586の分離特異的な遺伝子を同定した(Additional file 1: Table S1)。 また、3つの菌株の分離特異的遺伝子のうち2/3以上は、未特定タンパク質または注釈のない遺伝子であった。 また、柑橘類、梨、りんごなどの果樹類に感染するA. alternata分離株で報告されているような病原性を付与する可能性のある分散型染色体を3株とも含まなかった。 また、3つの菌株の遺伝子レパートリーは、エフェクター、CAZymes、二次代謝産物の数および種類がほぼ同じであった(表2)。 さらに、PN1およびPN2の2株は、着生条件下でB. junceaに単独で感染症状を引き起こさない(データは示していない)。 これらの結果は,A. alternataのこれらの分離株(PN1およびPN2)は,腐生生活を送るfacultative pathogensであり,特定の環境条件下で病原生活に切り替わる可能性を示唆している。
Fig. 3 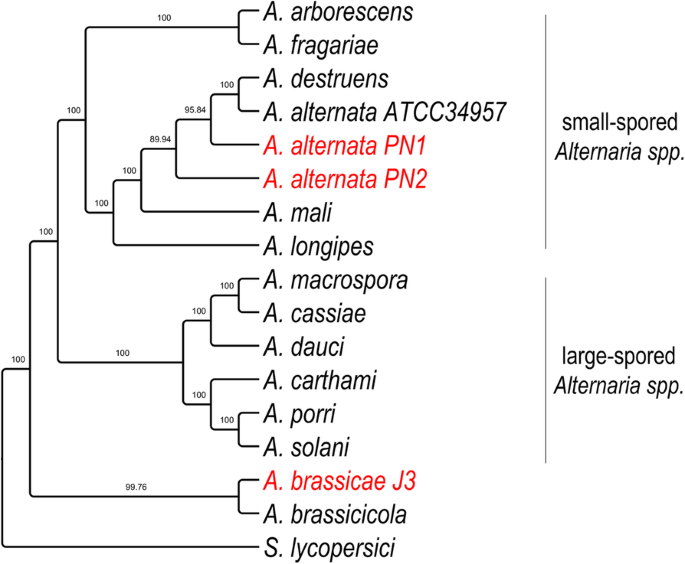
アウトグループとしての S. lycopersici と Alternaria 種の系統樹. Mirloで計算した最も高い系統的シグナルを持つ29の単一コピーオーソログを用いて樹を構築した。 表2 ほぼ完全な6種のAlternariaゲノム配列のタンパク質レパートリーと機能分類
A.A.における豊富なリピートリッチ領域とトランスポーザブルエレメント

A. alternata PN1 および PN2 と A. brassicae のホールゲノム・アラインメント。 A. alternata PN1およびPN2とA. brassicaeのマクロシンテンシーを示すCircos plot、dispensable contig (ABRSC11, scaffold13,17,18,19) を除くすべてのcontig、bおよびc Syntenic dotplot A. brassicae と A. alternata PN1 および PN2
Comparing analyses of A. A. brassicae, A. alternata PN1 および PN2 の比較研究。 alternata PN1およびPN2(B. junceaから分離)とA. alternata ATCC34957(ソルガムから分離)のゲノムを比較し、これらが全く異なる2つの種に感染することを可能にするかもしれないゲノム内容の相違を明らかにした。 A. alternata PN1およびPN2とA. alternata ATCC34957の全ゲノム配列を比較したところ、非常に高いシンテニーを示し、種特異的な領域は存在しないことが判明した。 また、A. alternataの3つの分離株の間で、それぞれ719、152、586の分離特異的な遺伝子を同定した(Additional file 1: Table S1)。 また、3つの菌株の分離特異的遺伝子のうち2/3以上は、未特定タンパク質または注釈のない遺伝子であった。 また、柑橘類、梨、りんごなどの果樹類に感染するA. alternata分離株で報告されているような病原性を付与する可能性のある分散型染色体を3株とも含まなかった。 また、3つの菌株の遺伝子レパートリーは、エフェクター、CAZymes、二次代謝産物の数および種類がほぼ同じであった(表2)。 さらに、PN1およびPN2の2株は、着生条件下でB. junceaに単独で感染症状を引き起こさない(データは示していない)。 これらの結果は,A. alternataのこれらの分離株(PN1およびPN2)は,腐生生活を送るfacultative pathogensであり,特定の環境条件下で病原生活に切り替わる可能性を示唆している。
Fig. 3 
アウトグループとしての S. lycopersici と Alternaria 種の系統樹. Mirloで計算した最も高い系統的シグナルを持つ29の単一コピーオーソログを用いて樹を構築した。 表2 ほぼ完全な6種のAlternariaゲノム配列のタンパク質レパートリーと機能分類
A.A.における豊富なリピートリッチ領域とトランスポーザブルエレメント

アウトグループとしての S. lycopersici と Alternaria 種の系統樹. Mirloで計算した最も高い系統的シグナルを持つ29の単一コピーオーソログを用いて樹を構築した。 表2 ほぼ完全な6種のAlternariaゲノム配列のタンパク質レパートリーと機能分類
A.A.における豊富なリピートリッチ領域とトランスポーザブルエレメント
糸状菌の植物病原菌は、リピート含量の高い明確なゲノム構造を持つ傾向がある。 RepeatModelerとRepeatMaskerを用いたリピート量の推定とマスキングの結果、A. brassicaeゲノムは〜9.33%のリピートで構成されており、A. alternataゲノムの2.43%と2.64%のリピートと比べ、高いリピート量であることが判明した。 A. brassicaeゲノムは、これまでに解読されたAlternaria種の中で最も高い繰り返し頻度(〜9.33%)を有していた。 その結果、A. alternataと他の病原性Alternariaとの間で、繰り返し配列の含有率が大きく異なることが明らかになった。 特にA. brassicaeとA. brassicicolaは、他のA. alternata分離株(病原性および非病原性)と比較して、LTR/GypsyおよびLTR/Copia要素のレパートリーがかなり多かった(> 8X)(図4)。 また、A. brassicaeとA. brassicicolaのゲノムにはDNAトランスポゾンが過剰に存在し、他のAlternaria種では<1%であるのに対し、ゲノムの〜5%に達した(図4)。

6 種の Alternaria における繰り返し含有量の比較。 泡の大きさは、(a)ゲノム中のトランスポーザブル・エレメント(TE)の割合、bゲノム中のTEのコピー数
この繰り返しDNAの増殖と、その領域に重なる遺伝子の進化は、これらの病原体が宿主との共進化的競合の世代を超えて持続することができた進化の成功の鍵である可能性がある。 TEに近接した遺伝子は、繰り返し誘発点突然変異(RIP)にさらされる可能性があり、その結果、進化が加速される。
A. brassicaeにおける分散型染色体の存在
A. alternataを含むいくつかの植物病原体種から、系統特異的染色体(LS)または分散型染色体(DC)が報告されている。 A. alternataのDCは,病原性や宿主特異性を付与することが知られている. しかし、このコンティグをアルテルナリア属で報告されている他の不感蒸泄性染色体の配列にアラインメントしたところ、部分的にシンテニーが認められた。 このことから、これらのコンティグを合わせて、A. brassicaeのDCを表している可能性が考えられた。 このことを確認するために、Alternaria属の分散型染色体のマーカー遺伝子として知られているAaMSAS遺伝子とALT1遺伝子の存在をコンティグで検索した。 その結果、1Mbのコンティグ上に2つの二次代謝産物生合成クラスターの一部として、AaMSAS遺伝子のコピーを2つ発見した。 しかし、ALT1遺伝子のホモログは見出せなかった。 さらに、コンティグ(ABRSC11、scaffold 13、17、18、19)のリピート含有量を全ゲノムと比較したところ、ABRSC11、scaffold 13、17、18、19のリピート含有量は、全ゲノムと同じであった。 系統特異的コンティグの遺伝子含量は、コア染色体の遺伝子含量より有意に低かった(表3)。 表3 コア染色体とディセンサブル染色体の比較
DCはコア染色体に比べて分泌タンパク質をコードする遺伝子に富んでいなかったが、分泌エフェクター遺伝子の割合はコア染色体に比べ30%高かった。 以上のことから、A. brassicaeは確かにDCを保有している可能性があることがわかった。 これまで、Alternaria属のDCは、小胞子性Alternaria属のみから報告されており、大胞子性Alternaria属にはDCを保有するものは知られていない。 DCがA. brassicaeの毒性に寄与しているかどうかはまだ不明である。
Orthology analysis reveals species-specific genes with putative roles in virulence
Differential gene content and diversity within genes contributions to adaptation, growth, and pathogenicity. そこで、Alternaria属とDothideomycetes属の遺伝子内容の違いをカタログ化するために、Dothideomycetes属に属する30種(Alternaria16種を含む)のタンパク質3,60,216個の集合に対してオーソロジー解析を行い(追加ファイル2:表S2)、そのうち少なくとも一つのオーソログループに割り当てられた3,45321個のタンパク質を明らかにしました。 A. brassicaeに存在し、他のAlternaria属には存在しないA. brassicae特異的遺伝子を460個同定した(Additional file 3: Table S3)。 これらの種特異的遺伝子には、35個の分泌タンパク質コード化遺伝子が含まれ、そのうち11個はエフェクターであると予測された。 さらに、これらの種特異的遺伝子のうち20個はDC上に存在した。 これらのタンパク質の多くは、機能未知のタンパク質に属していた。 これらの種特異的遺伝子がゲノムのリピートリッチ領域で起こった適応進化の結果であるかどうかを検証するために、リピートリッチ領域とトランスポーザブルエレメントのランダムな遺伝子セットとの重なりを、これらの種特異的遺伝子の重なりと比較する順列検定を実施した。 その結果、これらの種特異的遺伝子は、ゲノム中のリピートに富んだ領域(P値: 9.99e-05; Zスコア: – 4.825)およびトランスポーザブル・エレメント(P値: 0.0460; Zスコア: 2.539)と有意に重複していることがわかった。
A. brassicaeの二次代謝産物プロファイルとトランスポゾーン(TE)との関連
Alternaria属とCochliobolus属は宿主特異的二次代謝産物毒素の主要産地として知られています。 特にAlternaria属は化学的に多様な二次代謝産物を生産することが知られており、その中には宿主特異的毒素(HSTs)と非HSTsが含まれる。 これらの二次代謝産物は通常、非リボソームペプチド合成酵素(NRPS)やポリケタイド合成酵素(PKS)により生成される。 我々は、A. brassicaeに5つのNRPS型SM遺伝子クラスタ、12のPKS型遺伝子クラスタ、7つのテルペン様遺伝子クラスタを同定した(Additional file 4: Table S4)。 このうち、Destruxin B、HC-toxin、ジメチルコプロゲン(シデロフォア)という既知の二次代謝産物を生産する3つのクラスターを同定した(4613)
A. brassicaeのジメチルコプロゲンを生産する遺伝子群は、主要生合成遺伝子、酸化還元酵素、シデロフォア輸送体などの22遺伝子からなる。 シデロフォアは、真菌が細胞外の鉄を獲得するために用いる鉄キレート化合物で、真菌の病原性に関与することが報告されている。 シデロフォア合成を担う遺伝子群の同定は、A. brassicaeのシデロフォアとその病原性における役割の研究を可能にするものである。 さらに、メラニン産生に関わる 12 の遺伝子からなる PKS 型クラスターも同定した(Additional file 4: Table S4)。 このメラニン生合成クラスターは、A. alternataについて以前に報告されている。 また、A. brassicicolaでは、メラニン産生を誘導する転写因子Amr1が特徴的であり、病原性を抑制することが知られている . しかし、メラニンの毒性における役割は曖昧であり、種特異的である。
アルタナリア属に属する植物病原菌は、新しい生態的ニッチを植民地化するために、新しい二次代謝産物の潜在能力を獲得する動的な能力を持っているようである。 この二次代謝産物ポテンシャルの動的な獲得の最も簡潔な説明は、Alternaria属内およびおそらく他の属との水平的な遺伝子移動である。 AlternariaのHSTの多くがdispensable染色体上に存在し、これらの染色体を交換することで宿主特異性を拡大できることを示す文献が多数存在する。 我々はまた、DCの1つ(scaffold 18)にHC-toxinをコードしていると思われるNRPSクラスターを同定した(Additional file 4: Table S4)。 HC-毒素は植物病原菌Cochliobolus carbonumの毒性決定因子として知られており、この毒素を無毒化するカルボニル還元酵素HM1の機能コピーを欠いたトウモロコシの遺伝子型に感染する。 最近、A. jesenskaeもHC-毒素を産生することが示され、C. carbonum以外にこの毒素を産生する唯一の真菌となった。 C. carbonumの毒性決定因子であるHC-toxin遺伝子群がA. brassicaeのDCに存在することは、種間水平遺伝子伝達が予想以上に多いことを示唆している。
水平遺伝子伝達とは別に、SM遺伝子の急速な重複、分岐、喪失も病原体が新しい代謝能力を進化させることに寄与していると考えられる。 このような重複と分岐のプロセスは、二次代謝産物クラスターがリピート要素に近接しているため、RIP-mutationが起こりやすいことが寄与している可能性がある。 そこで、二次代謝産物クラスターがリピートに富む領域と関連しているかどうかを検証した。 順列検定を用い、リピートリッチ領域とランダムな遺伝子セットの重なりを、二次代謝産物クラスター遺伝子の重なりと比較した。 二次代謝産物クラスターは、ランダムな遺伝子セットと比較して、リピートリッチ領域に有意に重複していた(P値: 0.0017; Z-score: – 2.7963)。 また、これらのクラスターは、リピートリッチ領域の中でトランスポーザブルエレメントと有意に重複していた(P-value: 0.0087; Z-score: 2.9871)。 このことから、A. brassicae の場合、上記のような新規二次代謝産物ポテンシャルの獲得メカニズムの両方が可能である可能性が示唆された。 また、種や属レベルでの個体群解析により、Alternaria属におけるこれらの機構の普及状況が明らかになる可能性がある。
シンテニー解析により、A. brassicaeによるDestruxin B生産の排他性の遺伝的基盤が明らかとなった。 brassicaeの宿主特異的な毒素であることが報告されている。 Destruxin Bは他のAlternaria属細菌では生産されることが報告されていない。 本論文では、A. brassicaeのDestruxin B生産に関わる生合成遺伝子クラスターを初めて報告する。 このクラスターは、NRPS遺伝子にコードされる主要な生合成酵素(DtxS1)と律速酵素であるDtxS3(アルド・ケト還元酵素)を含む10遺伝子からなる(Additional file 4: Table S4)。 興味深いことに、6種のAlternariaの間でこのクラスターのシンテニー解析を行ったところ、これらのクラスターの全体的なシンテニーは維持されていたものの、これらの遺伝子は他のAlternaria属のいずれにも存在していなかった(図5)。 Destruxin Bクラスターに含まれる酵素DtxS1とDtxS3をコードする重要な遺伝子が他のAlternaria種に存在しないことは、それらの種にDestruxin Bが存在しないことを説明する。
図5 
Alternaria6種におけるDestruxin Bクラスターのシンテニー(Synteny in the six Alternaria species. DtxS1とDtxS3は赤で示され(それぞれの遺伝子IDでラベルされている)、他のすべてのAlternaria種には存在しない

Alternaria6種におけるDestruxin Bクラスターのシンテニー(Synteny in the six Alternaria species. DtxS1とDtxS3は赤で示され(それぞれの遺伝子IDでラベルされている)、他のすべてのAlternaria種には存在しない
Destruxin BはA. brassicae以外に昆虫病原体のMetalhizium robertsii 、およびバーミューダの春枯れの原因であるOphiosphaerella herpotrichaから報告されていた。 Wang らは、M. robertsii の Destruxin 生産に関与する二次代謝産物生合成クラスターを報告した。 Metarhizhium属のDestruxinクラスターは、DtxS2とDtxS4という2つの重要な遺伝子から構成されています。 DtxS2はDestruxin BからDestruxin A, C, D, Eへの変換に関与する。A. brassicaeのDtxS2およびDtxS4に対応するシンテニック遺伝子はクラスター内に見出せなかった。 A. brassicaeはDestruxin Bのみを生産し、他の形態のDestruxinを生産しないことが知られており、これがA. brassicaeにDtxS2が存在しないことの説明となる可能性がある。 DtxS4はアスパラギン酸からβ-アラニンを生成するのに必要なアスパラギン酸脱炭酸酵素で、Destruxinの骨格構造の生成に不可欠である。 さらに、DtxS4の欠失変異体は、培地中にβ-アラニンを補充すると、デストルキシンを生産することができた 。 DtxS4はDestruxinクラスターに存在しないが、A. brassicaeはゲノムの別の場所に存在するアスパラギン酸デカルボキシラーゼの機能コピーを1つ持っており、したがってDestruxin B合成のためのβ-アラニンの供給を確保できる可能性がある。 Metarhizhium属におけるDestruxin B生産の進化の歴史はAlternariaとよく類似していた。 M. acridumのような特殊な病原菌は、2つの重要な酵素を持たないため、Destruxinを生産することはない。 しかし、より広い宿主範囲を持つジェネラリストであるM. robertsiiは、Destruxinを生産する。 そこで、M. roberstiiの宿主域の拡大にはDestruxinsが関与している可能性が考えられた。 A. brassicaeは宿主域が広く、アブラナ科のほぼ全科に感染することから、同様の仮説が成り立つと考えられる。 A. brassicaeのDestruxin欠損変異体の宿主範囲を決定するためのさらなる実験は、A. brassicaeの宿主範囲を拡張するためのこの重要な植物毒の役割を明らかにするのに役立つであろう。
Distinct CAZyme profiles of A. brassicae and A. brassicicola within the Alternaria genus
CAZymes (Carbohydrate-Active enZymes) とはグリコシド結合の分解、再配置、合成に関わるタンパク質である。 植物病原菌は、植物細胞壁の複雑な多糖類を分解する多様なCAZymesを分泌している。 これらの酵素は、グリコシド加水分解酵素(GH)、糖転移酵素(GT)、多糖リアーゼ(PL)、糖質エステラーゼ(CE)、糖質結合モジュール(CBM)の5つのクラスから構成されている。 A. brassicaeを含む6つのAlternaria属に> 500のCAZymesを同定した(Additional file 5: Table S5)。 A. brassicaeとA. brassicicolaのCAZymeの分布は他のAlternaria属のものとは異なっており、別のクラスターを形成していた(図6)。 補助活性酵素や植物細胞壁分解に関与する酵素の数は、比較した異なる種間でかなり異なっていた。 図6

Alternaria属16種におけるCAZymeプロファイルのヒートマップ。 生物のクラスタリングは、アウトグループのStemphylium lycopersiciと別々にクラスタリングするA. brassicaeとA. brassicicolaを除いて、種の系統樹と密接に類似している。 CAZymesはCAZy分類
AA9ファミリー(旧GH61;銅依存性溶菌多糖類モノオキシゲナーゼ(LPMO))はAlternaria属の他のCAZyファミリーと比較して著しく大きく、各種が> 20コピーの遺伝子を含んでいることが示された。 また、Alternaria属のコピー数は、Botrytis属やFusarium属で報告されているコピー数よりはるかに多い。 AA9ファミリーは細胞壁多糖の分解に関与し、デンプン、キシログルカン、セロデキストリン、グルコマンナンなど様々な多糖類に作用することが知られている。 LPMOは、細胞壁多糖を直接切断すると同時に、活性酸素の発生源として働き、植物組織の壊死につながる酸化ストレスの一因となるという二重の役割を担っていると考えられてきた。 A. brassicaeに存在する26個のAA9タンパク質のうち、11個が分泌型エフェクターであると予測されるのは驚くべきことである。 これらの CAZymes の特性や病原体としての役割については、今後さらに研究を進める予定である。
Overlapping effector profiles within the Alternaria genus
A. brassicae を含む6種の Alternaria (Table 2) について、方法のセクションで述べたパイプラインを用いてエフェクターのレパートリーを予測した。 エフェクターと予測された小型分泌タンパク質のほとんどは未特定/予測タンパク質であったため、新規の種または属特異的エフェクターである可能性がある。 A. brassicaeはエフェクター候補の集合の中で未知/予測/仮説タンパク質の割合が最も大きかった。 また、6種間のエフェクターはほとんどが共通で重複していることから、タンパク質性エフェクターが関与する広範な病原性メカニズムが属内で保存されている可能性が示唆された。 しかし、β/γ-クリスタリンフォールドを含むタンパク質の2つのコピーがA. brassicaeとA. brassicicolaにのみ存在し、他の4種のAlternariaには存在しないことを見出した。 NCBIのnrデータベースでさらに検索したところ、このタンパク質はAlternaria属には全く存在せず、他の植物病原菌、すなわちMacrophomina属とFusarium属に最も近いものが見つかった。 また、A. brassicaeのエフェクターは、ゲノムのリピートリッチ領域と有意に関連していた(P値:0.0003、Zスコア:-2.8778)ことから、適応的に進化する可能性を持っていることが確認された。 したがって、集団レベルでの解析により、正選択下にあるエフェクターを同定し、アブラナ科植物の病原性の進化を明らかにすることが必要である。 本研究で同定されたエフェクターは、A. brassicaeの病原性に関与していると考えられる幅広いタンパク質を明らかにしている。 これらのエフェクターのうち39個は、細胞壁や関連多糖の分解に様々な役割を持つCAZymesであると予測された。 A. brassicaeのゲノムには2種類の壊死およびエチレン誘導ペプチドタンパク質が含まれており、これらは菌類や壊死栄養細菌を含む様々な病原体の病因に関与していると考えられている。 A. alternataのHrip1 (Hypersensitive response inducing protein 1)はMdNLR16に古典的な遺伝子対遺伝子で認識され、A. alternataからHrip1を欠損させると病原性が高まることが最近明らかにされた。 A. brassicaeにもHrip1ホモログが存在するが、細胞外に分泌されないと予測される。 このように遺伝子レベルで認識されるエフェクターが存在することは、宿主の相補的なR遺伝子を同定し、抵抗性品種や栽培品種の開発に活用できる可能性があると考えられる
。