Le caratteristiche genomiche di A. brassicae e di altri due isolati di A. alternata coinfettati
Abbiamo sequenziato i genomi di due isolati di A. alternata (PN1 e PN2) che stavano coinfettando B. juncea con A. brassicae. L’assemblaggio di A. brassicae è stato precedentemente descritto. In breve, l’insieme consisteva di nove cromosomi completi e un cromosoma con ripetizioni telomeriche mancanti a una delle estremità. Oltre a questi cromosomi, c’erano sei contigs di cui uno di loro era ~ 1 Mb di dimensioni, che possono insieme costituire un cromosoma dispensabile (Fig. 1). L’N50 dell’insieme di A. brassicae era di 2,98 Mb (Tabella 1). I due isolati che co-infettano B. juncea sono stati identificati come A. alternata sulla base delle loro sequenze ITS e GAPDH. Gli assemblaggi A. alternata Aat_PN1 e Aat_PN2 consistevano in 14 contigs per un totale di 33,77 Mb e 15 contigs per un totale di 33,53 Mb, rispettivamente (Tabella 1). Sei contigs in ciascuno dei due assemblaggi contenevano ripetizioni telomeriche su entrambe le estremità e quindi, molto probabilmente, rappresentano molecole cromosomiche complete. Quattro altri contigs in entrambi gli assemblaggi contenevano ripetizioni telomeriche su un’estremità ma erano di dimensioni simili di molecole cromosomiche complete come descritto in A. solani . Pertanto, gli assemblaggi del genoma per gli isolati di A. alternata rappresentavano dieci cromosomi quasi completi di ciascuno dei due isolati.
L’allineamento del genoma intero con Alternaria spp. correlate ha mostrato una sintonia generale tra i genomi con riarrangiamenti minori (Fig. 2). Inoltre, le sequenze mitocondriali sono state ottenute dai dati di sequenziamento per i due isolati di A. alternata. I genomi mitocondriali dei ceppi di A. alternata erano rispettivamente di circa 49.783 bp e 50.765 bp e hanno mostrato un’elevata somiglianza con il genoma mitocondriale di A. alternata precedentemente pubblicato.
La predizione genica dopo il mascheramento delle ripetizioni ha portato all’identificazione di 11593, 11495 e 11387 geni negli assemblaggi del genoma di A. brassicae, A. alternata PN1 e PN2, rispettivamente. Questo era paragonabile ai numeri di geni stimati in altre Alternaria spp. (Tabella 1). L’analisi BUSCO ha mostrato che i modelli genici previsti nei tre genomi coprivano il 98% dei geni fungini conservati a copia singola, indicando la quasi completezza degli assemblaggi. I geni predetti sono stati annotati in modo completo utilizzando una combinazione di database come descritto nella sezione Metodi (Fig. 1). Oltre ai tre genomi, abbiamo anche predetto geni de novo nel genoma assemblee di altre tre specie di Alternaria che sono stati sequenziati utilizzando tecnologie long-read viz. A. brassicicola (abra43), A. alternata (ATCC34957), e A. solani (altNL03003) (Tabella 1). Questi sei genomi e le loro predizioni geniche sono stati utilizzati per le analisi comparative dei cluster di geni codificanti i metaboliti secondari e i geni codificanti gli effettori.

Sintesi del genoma di A. brassicae, (dalle tracce circolari esterne a quelle interne) a pseudocromosomi/scaffold, b geni codificanti proteine, c elementi ripetitivi, d elementi trasponibili (DNA e LTR), e cluster di metaboliti secondari previsti, f proteine secrete, g effettori previsti
L’analisi filogenomica assegna un clade separato per A. brassicae e A. brassicicola all’interno del genere Alternaria
Al fine di ricostruire accuratamente la divergenza e la relazione tra A. brassicae, i due isolati di A. alternata (PN1 e PN2), e le altre specie di Alternaria, abbiamo condotto analisi filogenomiche utilizzando 29 ortologhi a copia singola che avevano il più alto segnale filogenetico come calcolato dal programma Mirlo. La selezione dei geni con segnali filogenetici più alti porta a filogenesi che sono più congruenti con l’albero delle specie. La filogenesi risultante ha mostrato che le specie di Alternaria a pori grandi e a pori piccoli si sono raggruppate separatamente in due cladi diverse (Fig. 3). È interessante notare che i due principali patogeni del Brassicas viz. A. brassicae e A. brassicicola si sono raggruppati separatamente da tutte le altre specie di Alternaria, forse indicando una diversa traiettoria evolutiva basata sulle preferenze comuni dell’ospite di queste due specie.
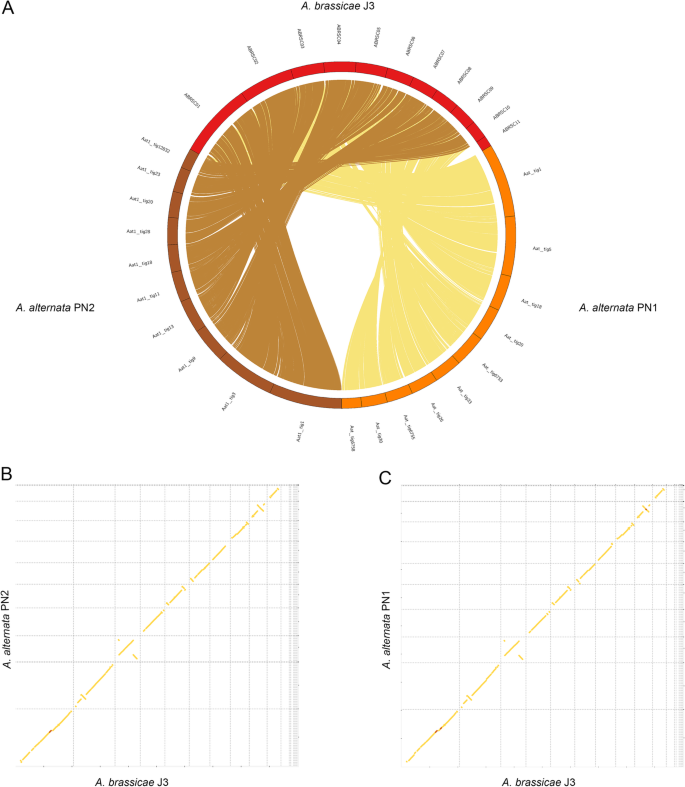
Allineamenti dell’intero genoma di A. alternata PN1 e PN2 con A. brassicae. brassicae attraverso tutti i contigs eccetto i contigs dispensabili (ABRSC11, scaffold13,17,18,19), b e c plot di punti sintetici di A. brassicae con A. alternata PN1 e PN2
Analisi comparative di A. alternata isolati ottenuti da diversi ospiti
Abbiamo confrontato i genomi di A. alternata PN1 e PN2 (isolati da B. juncea) con quello di A. alternata ATCC34957 (isolato dal sorgo) per identificare eventuali differenze nel loro contenuto genomico che potrebbero permettere a questi di infettare due specie molto diverse. Gli allineamenti dell’intero genoma di A. alternata PN1 e PN2 con quello di A. alternata ATCC34957 hanno rivelato livelli molto alti di sintenia e l’assenza di regioni specie-specifiche. Abbiamo identificato 719, 152 e 586 geni specifici dell’isolato tra i tre isolati di A. alternata, rispettivamente (file aggiuntivo 1: Tabella S1). Più di due terzi dei geni isolato-specifici in tutti e tre gli isolati erano proteine non caratterizzate o non avevano annotazioni. In particolare, tutti e tre gli isolati non contenevano cromosomi dispensabili che possono conferire patogenicità, come è stato riportato per gli isolati di A. alternata che infettano molte delle colture di frutta come agrumi, pere e mele. I repertori genici dei tre isolati consistevano anche in un numero simile e un tipo di effettori, CAZymes, e gruppi di metaboliti secondari (Tabella 2). Inoltre, i due isolati PN1 e PN2 non causano sintomi di infezione da soli in B. juncea in condizioni epifite (dati non mostrati). I nostri risultati suggeriscono che questi isolati di A. alternata (PN1 e PN2) possono essere patogeni facoltativi che conducono uno stile di vita saprofita e possono passare a uno stile di vita patogeno in determinate condizioni ambientali.
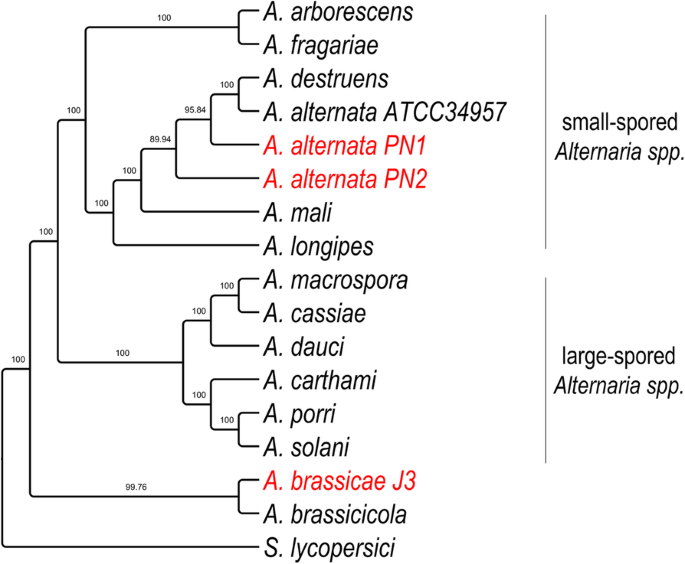
Albero filogenetico delle specie di Alternaria con S. lycopersici come outgroup. L’albero è stato costruito utilizzando 29 ortologhi a copia singola, che avevano il più alto segnale filogenetico come calcolato in Mirlo. I valori di supporto dei rami da 1000 repliche bootstrap sono mostrati
Un’abbondanza di regioni ricche di ripetizioni ed elementi trasponibili in A. brassicae
I patogeni delle piante filamentose tendono ad avere un’architettura del genoma distinta con un maggiore contenuto di ripetizioni. La stima del contenuto di ripetizioni e il mascheramento con RepeatModeler e RepeatMasker hanno rivelato che il genoma di A. brassicae consisteva di ~ 9,33% di ripetizioni rispetto al 2,43 e 2,64% di ripetizioni nei genomi di A. alternata. Il genoma di A. brassicae ospita il più alto contenuto di ripetizioni (~ 9,33%) tra tutte le specie di Alternaria sequenziate fino ad oggi. La nostra analisi ha mostrato che il contenuto di ripetizioni differisce significativamente tra gli isolati di A. alternata e le altre specie patogene di Alternaria. Le specie patogene di Alternaria, in particolare A. brassicae e A. brassicicola avevano un repertorio notevolmente più grande di elementi LTR/Gypsy e LTR/Copia (> 8X) rispetto agli altri isolati di A. alternata (patogeni e non patogeni) (Fig. 4). I genomi di A. brassicae e A. brassicicola avevano anche una sovrarappresentazione dei trasposoni del DNA, che ammontavano a ~ 5% del genoma, rispetto a < 1% nelle altre specie di Alternaria (Fig. 4).

Comparazione del contenuto di ripetizioni in sei specie di Alternaria. La dimensione delle bolle corrisponde alla (a) percentuale di elementi trasponibili (TEs) nel genoma, b numero di copie del TE nel genoma
Questa proliferazione di DNA ripetitivo e la successiva evoluzione dei geni che si sovrappongono a queste regioni può essere la chiave del successo evolutivo in cui questi patogeni sono riusciti a persistere per generazioni di conflitto co-evolutivo con i loro ospiti. La vicinanza ai TE espone potenzialmente i geni a mutazioni puntiformi indotte dalla ripetizione (RIP) e quindi ad un’evoluzione accelerata. La ricombinazione ectopica tra TE simili può anche portare a nuove combinazioni di geni e quindi aumentare la diversità delle proteine o dei metaboliti.
Presenza di un cromosoma dispensabile nell’A. brassicae a pori larghi
Cromosomi specifici del lignaggio (LS) o cromosomi dispensabili (DC) sono stati riportati da diverse specie fitopatogene tra cui A. alternata. I DC in A. alternata sono noti per conferire virulenza e specificità dell’ospite all’isolato. Gli allineamenti del genoma intero di A. brassicae con altre Alternaria spp. hanno rivelato che un contig di circa 1 Mb insieme ad altri contig più piccoli (66-366 kb) era specifico per A. brassicae e non mostrava sintenia con nessuna regione nelle altre Alternaria spp. Tuttavia, una parziale sintenia è stata osservata quando il contig è stato allineato alle sequenze di altri cromosomi dispensabili riportati in Alternaria spp. Questo ci ha portato a ipotizzare che questi contig insieme possano rappresentare un DC di A. brassicae. Per confermare questo, abbiamo cercato i contigs per la presenza di AaMSAS e ALT1genes, che sono noti geni marcatori per cromosomi dispensabili in Alternaria spp. Abbiamo trovato due copie del gene AaMSAS come parte di due cluster biosintetici di metaboliti secondari sul contig di 1 Mb. Tuttavia, non abbiamo trovato alcun omologo del gene ALT1. Inoltre, il contenuto di ripetizioni dei contig (ABRSC11, scaffold 13, 17, 18 e 19) è stato confrontato con l’intero genoma. Il contenuto genico dei contigs specifici del lignaggio era significativamente inferiore a quello dei cromosomi del nucleo (Tabella 3). Al contrario, i contigs DC erano altamente arricchiti nel contenuto di TE rispetto ai cromosomi del nucleo (Tabella 3).
Anche se il DC non era arricchito di geni che codificano proteine secrete, la percentuale di geni effettori secreti era del 30% più alta rispetto ai cromosomi del nucleo. Tutte le prove di cui sopra indicano che A. brassicae può effettivamente ospitare un DC. I DC in Alternaria spp. sono stati riportati finora solo da Alternaria spp. a pori piccoli e nessuna specie di Alternaria a pori grandi è nota per ospitare i DC. Resta da vedere se il DC contribuisce alla virulenza di A. brassicae. Studi futuri dovrebbero comprendere la caratterizzazione del cromosoma dispensabile in A. brassicae e correlare la sua presenza alla patogenicità di diversi isolati.
L’analisi ortologica rivela geni specie-specifici con ruoli putativi nella virulenza
Le differenze nel contenuto genico e la diversità all’interno dei geni contribuiscono all’adattamento, alla crescita e alla patogenicità. Al fine di catalogare le differenze nel contenuto genico all’interno del genere Alternaria e dei Dothideomycetes, abbiamo effettuato un’analisi di ortologia sul set combinato di 3.60.216 proteine da 30 specie diverse (comprese 16 specie di Alternaria) appartenenti ai Dothideomycetes (file aggiuntivo 2: Tabella S2) di cui 3.45.321 proteine potrebbero essere assegnate ad almeno uno degli ortogruppi. Abbiamo identificato 460 geni specifici di A. brassicae che erano presenti in A. brassicae ma assenti in tutte le altre specie di Alternaria (file aggiuntivo 3: tabella S3). Questi geni specie-specifici includevano 35 geni codificanti proteine secrete di cui 11 sono stati predetti essere effettori. Inoltre, 20 di questi geni specie-specifici erano presenti sul DC. Un gran numero di queste proteine apparteneva alla categoria delle proteine non caratterizzate e senza funzione nota. Per verificare se questi geni specie-specifici sono il risultato dell’evoluzione adattativa che ha luogo nelle regioni ricche di ripetizioni del genoma, abbiamo effettuato un test di permutazione per confrontare la sovrapposizione delle regioni ricche di ripetizioni e degli elementi trasponibili con un set di geni casuali con la sovrapposizione di questi geni specie-specifici. Abbiamo trovato che questi geni specie-specifici si sono sovrapposti in modo significativo con regioni ricche di ripetizioni (P-value: 9.99e-05; Z-score: – 4.825) ed elementi trasponibili (P-value: 0.0460; Z-score: 2.539) nel genoma.
Profilo dei metaboliti secondari di A. brassicae e la sua associazione con elementi trasponibili (TEs)
I generi di Alternaria e Cochliobolus sono noti per essere i maggiori produttori di tossine di metaboliti secondari specifici dell’ospite. L’Alternaria spp. in particolare è nota per la produzione di metaboliti secondari chimicamente diversi, che includono le tossine specifiche dell’ospite (HST) e le non-HST. Questi metaboliti secondari sono di solito generati da non-ribosomal peptide synthases (NRPS) e polyketide synthases (PKS). Abbiamo identificato cinque cluster di geni SM di tipo NRPS, 12 cluster di geni di tipo PKS e sette cluster di geni simili a terpeni in A. brassicae (file aggiuntivo 4: tabella S4). Dei cinque cluster NRPS, abbiamo potuto identificare tre cluster che producono metaboliti secondari noti, cioè Destruxin B, HC-toxin e dimetilcoprogeno (sideroforo) con ruoli noti nella virulenza.
Il cluster genico responsabile della produzione di dimetilcoprogeno (sideroforo) in A. brassicae consiste di 22 geni, compresi i principali geni biosintetici, ossidoreduttasi e trasportatori di sideroforo. I siderofori sono composti ferro-chelanti, utilizzati dai funghi per acquisire ferro ferrico extracellulare e sono stati segnalati per essere coinvolti nella virulenza fungina. L’identificazione del cluster genico responsabile della sintesi dei siderofori permetterebbe lo studio dei siderofori e del loro ruolo nella patogenicità in A. brassicae. Inoltre, è stato identificato anche un cluster di tipo PKS composto da 12 geni, responsabile della produzione di melanina (file aggiuntivo 4: tabella S4). Il cluster biosintetico della melanina è stato descritto precedentemente per A. alternata. Inoltre, il fattore di trascrizione Amr1, che induce la produzione di melanina, è stato caratterizzato in A. brassicicola ed è noto per sopprimere la virulenza. Tuttavia, il ruolo della melanina nella virulenza è ambiguo e specie-specifico.
I patogeni delle piante appartenenti al genere Alternaria sembrano avere una capacità dinamica di acquisire nuovo potenziale di metaboliti secondari per colonizzare nuove nicchie ecologiche. La spiegazione più parsimoniosa per questa acquisizione dinamica del potenziale di metaboliti secondari è il trasferimento genico orizzontale all’interno del genere di Alternaria e possibilmente con altri generi. C’è un’ampia evidenza in letteratura che gran parte degli HST di Alternaria sono trasportati sui cromosomi dispensabili e lo scambio di questi cromosomi può ampliare la specificità dell’ospite. Abbiamo anche identificato un cluster NRPS, possibilmente codificante per HC-toxin in uno dei DCs (scaffold 18) (Additional file 4: Tabella S4). HC-toxin è un noto determinante di virulenza del patogeno vegetale Cochliobolus carbonum, che infetta i genotipi di mais che mancano di una copia funzionale di HM1, una carbonil reduttasi che detossifica la tossina. Un recente rapporto ha dimostrato che anche A. jesenskae potrebbe produrre HC-tossina, rendendolo l’unico altro fungo oltre a C. carbonum a produrre la tossina. La presenza del cluster di geni HC-toxin, un determinante di virulenza in C. carbonum, in un DC di A. brassicae indica il fatto che il trasferimento genico orizzontale interspecie può essere più comune del previsto.
Oltre al trasferimento genico orizzontale, la rapida duplicazione, divergenza e perdita dei geni SM può anche contribuire all’evoluzione di nuove capacità metaboliche del patogeno. Questi processi di duplicazione e divergenza possono essere favoriti dalla vicinanza dei cluster di metaboliti secondari agli elementi di ripetizione che li rende inclini alle mutazioni RIP. Pertanto, abbiamo testato se i cluster di metaboliti secondari erano anche associati a regioni ricche di ripetizioni. Un test di permutazione è stato utilizzato per confrontare la sovrapposizione delle regioni ricche di ripetizioni con un set di geni casuali contro la sovrapposizione dei cluster di geni del metabolita secondario. I cluster di metaboliti secondari si sono sovrapposti in modo significativo alle regioni ricche di ripetizioni rispetto al set di geni casuali (P-value: 0.0017; Z-score: – 2.7963). Inoltre, questi cluster si sono sovrapposti significativamente con elementi trasponibili tra le regioni ricche di ripetizioni (P-value: 0.0087; Z-score: 2.9871). Questo dimostra che entrambi i meccanismi descritti sopra per l’acquisizione del potenziale di nuovi metaboliti secondari possono essere possibili nel caso di A. brassicae. Le analisi su scala di popolazione a livello di specie e di genere possono far luce sulla prevalenza di questi meccanismi all’interno del genere Alternaria.
L’analisi della sinteticità rivela la base genetica dell’esclusività della produzione di Destruxin B da parte di A. brassicae all’interno del genere Alternaria
La Destruxina B rappresenta una classe di depsipeptidi ciclici che è nota per essere uno dei fattori chiave di patogenicità di A. brassicae ed è stata riportata come tossina ospite-specifica di A. brassicae. La Destruxina B non è stata segnalata come prodotta da nessuna delle altre specie di Alternaria. Qui riportiamo per la prima volta i cluster di geni biosintetici responsabili della produzione di Destruxin B in A. brassicae. Il cluster è composto da 10 geni, tra cui il principale enzima biosintetico codificato da un gene NRPS (DtxS1) e l’enzima limitante, DtxS3 (aldo-cheto reduttasi) (file aggiuntivo 4: tabella S4). È interessante notare che l’analisi della sintenia di questo cluster tra le sei specie di Alternaria ha mostrato che entrambi questi geni non erano presenti in nessuna delle altre Alternaria spp. sebbene la sintenia complessiva del cluster fosse mantenuta in tutte queste specie (Fig. 5). L’assenza dei geni chiave che codificano per gli enzimi DtxS1 e DtxS3 nel cluster della Destruxina B nelle altre specie di Alternaria spiega l’assenza della Destruxina B in queste specie.

Sintonia del cluster della Destruxina B nelle sei specie di Alternaria. DtxS1 e DtxS3 sono segnati in rosso (etichettati con i rispettivi ID dei geni) e sono assenti da tutte le altre specie di Alternaria
Destruxina B è stata riportata dall’entomopatogeno Metarhizium robertsii , e Ophiosphaerella herpotricha , la causa della macchia morta primaverile di bermudagrass, a parte A. brassicae. Wang et al. hanno descritto il cluster biosintetico del metabolita secondario responsabile della produzione di Destruxin in M. robertsii. Il cluster della Destruxina nelle specie di Metarhizhium consiste di due geni chiave aggiuntivi, cioè DtxS2 e DtxS4. DtxS2 è responsabile della conversione della Destruxina B in Destruxina A, C, D, ed E. Non abbiamo potuto trovare nessun gene sintenico nel cluster che corrisponda a DtxS2 o DtxS4 in A. brassicae. A. brassicae è nota per produrre solo la Destruxina B e non le altre forme di Destruxina, il che potrebbe spiegare l’assenza di DtxS2 in A. brassicae. DtxS4 è una decarbossilasi dell’aspartato che è richiesta per la produzione di β-alanina dall’acido aspartico ed è essenziale per la produzione della struttura portante della Destruxina. Inoltre, i mutanti di delezione di DtxS4 sono stati in grado di produrre Destruxin quando sono stati integrati con β-alanina nel mezzo. Anche se DtxS4 non è presente nel cluster Destruxin, A. brassicae ha una copia funzionale di decarbossilasi aspartato presente altrove nel genoma e quindi può eventualmente garantire la fornitura di β-alanina per la sintesi di Destruxin B. La storia evolutiva della produzione di Destruxin B all’interno del genere Metarhizhium assomiglia molto a quella di Alternaria. I patogeni specializzati come M. acridum mancano dei due enzimi chiave e quindi non producono Destruxine. Tuttavia, M. robertsii, un generalista con una gamma più ampia di ospiti produce Destruxins. È stato quindi ipotizzato che le Destruxine potrebbero essere responsabili dell’instaurazione dell’estesa gamma di ospiti di M. roberstii. Un’ipotesi simile potrebbe essere vera anche nel caso di A. brassicae, che ha un ampio raggio d’azione e può infettare quasi tutte le Brassicaceae. Ulteriori esperimenti per determinare la gamma di ospiti dei mutanti nulli Destruxin di A. brassicae possono aiutare a chiarire il ruolo di questa importante fitotossina nell’estendere la gamma di ospiti di A. brassicae.
Profili distinti di CAZyme di A. brassicae e A. brassicicola nel genere Alternaria
CAZymes (Carbohydrate-Active enZymes) sono proteine coinvolte nella degradazione, riorganizzazione o sintesi di legami glicosidici. I patogeni delle piante secernono una gamma diversificata di CAZimi che scompongono i complessi polisaccaridi della parete cellulare delle piante. Essi consistono in cinque classi distinte: idrolasi glicosidiche (GH), glicosiltransferasi (GT), lisasi polisaccaridiche (PL), esterasi dei carboidrati (CE) e moduli di legame dei carboidrati (CBM). Abbiamo identificato > 500 CAZymes nei sei Alternaria spp. compreso A. brassicae (file aggiuntivo 5: Tabella S5). La distribuzione CAZyme di A. brassicae e A. brassicicola variava da quelle delle altre Alternaria spp. formando così un cluster separato (Fig. 6). Il numero di enzimi ad attività ausiliaria o gli enzimi coinvolti nella degradazione della parete cellulare delle piante variava notevolmente tra le diverse specie confrontate. Quasi il 46% dei CAZymes in A. brassicae erano secreti di cui ~ 17% erano predetti essere effettori.

Heatmap dei profili CAZyme in 16 specie con genere Alternaria. Il raggruppamento degli organismi assomiglia molto a quello della filogenesi delle specie tranne A. brassicae e A. brassicicola, che si raggruppano separatamente con l’outgroup Stemphylium lycopersici. I CAZimi sono stati raggruppati secondo la classificazione CAZy
La famiglia AA9 (ex GH61; monoossigenasi litiche polisaccaridi dipendenti dal rame (LPMO)) è significativamente ingrandita rispetto alle altre famiglie CAZy nel genere Alternaria con ogni specie contenente > 20 copie del gene. I numeri di copie nelle Alternaria spp. sono molto più alti dei numeri di copie riportati per Botrytis e Fusarium spp. La famiglia AA9 è coinvolta nella degradazione dei polisaccaridi della parete cellulare e sono noti per agire su una serie di polisaccaridi tra cui amido, xiloglucano, cellodestrine e glucomannano. Si è ipotizzato che gli LPMO abbiano un duplice ruolo: scindere direttamente i polisaccaridi delle pareti cellulari e agire come generatori di ROS, contribuendo così allo stress ossidativo che porta alla necrosi dei tessuti vegetali. Sorprendentemente, 11 delle 26 proteine AA9 presenti in A. brassicae sono previste come effettori secreti. La caratterizzazione di questi CAZymes e il loro ruolo nella patogenesi potrebbe essere oggetto di ulteriori studi.
Profili effettori sovrapposti all’interno del genere Alternaria
Abbiamo predetto il repertorio effettore di sei specie di Alternaria (Tabella 2) tra cui A. brassicae utilizzando la pipeline descritta nella sezione metodi. La maggior parte delle piccole proteine secrete previste per essere effettori erano proteine non caratterizzate/predette e quindi possono essere nuove specie o effettori specifici del genere. A. brassicae aveva la più grande proporzione di proteine sconosciute/predette/ipotetiche nel set di candidati effettori. Abbiamo trovato che la maggior parte degli effettori tra le sei specie sono comuni e si sovrappongono, suggerendo che gli ampi meccanismi di patogenesi che coinvolgono gli effettori proteici possono essere conservati all’interno del genere. Tuttavia, abbiamo trovato due copie di una proteina contenente la piega beta/gamma-cristallina presenti solo in A. brassicae e A. brassicicola e non nelle altre quattro specie di Alternaria. Un’ulteriore ricerca attraverso il database nr di NCBI ha rivelato che questa proteina era completamente assente nel genere Alternaria e le corrispondenze più vicine sono state trovate in altri patogeni delle piante, cioè Macrophomina e Fusarium. Tuttavia, nessuna informazione è disponibile per quanto riguarda la sua funzione o il ruolo nella patogenicità in nessuna delle specie.
Abbiamo anche potuto stabilire che alcuni degli effettori in A. brassicae hanno il potenziale di evolvere in modo adattivo poiché erano anche significativamente associati con le regioni ricche di ripetizioni del genoma (P-value: 0.0003; Z-score: – 2.8778). Le analisi a livello di popolazione sono quindi necessarie per identificare gli effettori sotto selezione positiva, che potrebbero far luce sull’evoluzione della patogenicità in A. brassicae. Gli effettori identificati in questo studio rivelano la vasta gamma di proteine che possono essere coinvolte nella patogenesi di A. brassicae. 39 di questi effettori sono stati predetti essere CAZymes che hanno vari ruoli nella degradazione della parete cellulare e dei polisaccaridi associati. Il genoma di A. brassicae conteneva due proteine peptidiche che inducono la necrosi e l’etilene, che sono state implicate nella patogenesi di vari patogeni tra cui oomiceti e necrotrofi. Hrip1 (Hypersensitive response inducing protein 1) di A. alternata ha recentemente dimostrato di essere riconosciuto da MdNLR16 in un modo classico gene-per-gene, e la delezione di Hrip1 da A. alternata aumenta la sua virulenza. Un omologo di Hrip1 è presente anche in A. brassicae, ma non è previsto che sia secreto al di fuori della cellula, anche se questo deve essere verificato sperimentalmente. La presenza di effettori che sono riconosciuti in un modo gene-per-gene apre la possibilità di identificazione di R-geni complementari nell’ospite che possono essere utilizzati per sviluppare varietà o cultivar resistenti.