Caracteristici genomice ale A. brassicae și a altor două izolate de A. alternata coinfectante
Am secvențiat genomurile a două izolate de A. alternata (PN1 și PN2) care au coinfectat B. juncea cu A. brassicae. Ansamblul A. brassicae a fost descris anterior . Pe scurt, ansamblul a constat din nouă cromozomi compleți și un cromozom cu repetări telomerice lipsă la unul dintre capete. În afară de acești cromozomi, existau șase contig-uri, dintre care unul dintre ele avea o dimensiune de ~ 1 Mb, care pot constitui împreună un cromozom dispensabil (Fig. 1). N50 a ansamblului A. brassicae a fost de 2,98 Mb (tabelul 1). Cele două izolate care au coinfectat B. juncea au fost identificate ca fiind A. alternata pe baza secvențelor ITS și GAPDH ale acestora. Ansamblurile A. alternata Aat_PN1 și Aat_PN2 au constat din 14 contig-uri care totalizează 33,77 Mb și, respectiv, 15 contig-uri care totalizează 33,53 Mb (tabelul 1). Șase contig-uri din fiecare dintre cele două ansambluri conțineau repetări telomerice la ambele capete și, prin urmare, este foarte probabil să reprezinte molecule cromozomiale complete. Alte patru contig-uri din ambele ansambluri conțineau repetări telomerice la un capăt, dar aveau dimensiuni similare moleculelor cromozomiale complete, așa cum s-a descris la A. solani . Prin urmare, ansamblurile genomice pentru izolatele de A. alternata au reprezentat zece cromozomi aproape compleți ai fiecăruia dintre cele două izolate.
Alinierile genomului întreg cu Alternaria spp. înrudite au arătat o sintenie generală între genomuri, cu rearanjamente minore (Fig. 2). În plus, secvențele mitocondriale au fost obținute, de asemenea, din datele de secvențiere pentru cele două izolate de A. alternata. Genomurile mitocondriale ale tulpinilor de A. alternata au avut o dimensiune de aproximativ 49 783 pb și, respectiv, 50 765 pb și au prezentat o similitudine ridicată cu genomul mitocondrial al A. alternata publicat anterior.
Predicția genelor în urma mascării repetițiilor a dus la identificarea a 11593, 11495 și 11387 gene în ansamblurile genomice A. brassicae, A. alternata PN1 și, respectiv, PN2. Acest lucru a fost comparabil cu numărul de gene estimat la alte specii de Alternaria (tabelul 1). Analiza BUSCO a arătat că modelele de gene prezise în cele trei genomuri au acoperit 98% din genele fungice conservate într-o singură copie, indicând caracterul cvasi-complet al ansamblurilor. Genele prezise au fost adnotate în mod cuprinzător utilizând o combinație de baze de date, așa cum este descris în secțiunea Metode (Fig. 1). În plus față de cele trei genomuri, am prezis, de asemenea, genele de novo în ansamblurile genomice ale altor trei specii de Alternaria care au fost secvențiate cu ajutorul tehnologiilor long-read, și anume A. brassicicola (abra43), A. alternata (ATCC34957) și A. solani (altNL03003) (tabelul 1). Aceste șase genomuri și predicțiile genetice ale acestora au fost utilizate pentru analizele comparative ale clusterelor de gene codificatoare de metaboliți secundari și ale genelor codificatoare de efectori.

Rezumat al genei A. brassicae, (De la pistele circulare exterioare la cele interioare) a pseudocromosomi/scafolduri, b Gene codificatoare de proteine, c Elemente repetate, d Elemente transpozabile (ADN și LTR), e Clustere de metaboliți secundari prezise, f Proteine secretate, g Efectoare prezise
Analiza filogenomică atribuie o cladă separată pentru A. brassicae-infectantă. brassicae și A. brassicicola în cadrul genului Alternaria
Pentru a reconstitui cu exactitate divergența și relația dintre A. brassicae, cele două izolate de A. alternata (PN1 și PN2) și celelalte specii de Alternaria, am efectuat analize filogenomice folosind 29 de ortologi cu o singură copie care au avut cel mai mare semnal filogenetic, așa cum a fost calculat de programul Mirlo. Selectarea genelor cu semnale filogenetice mai mari conduce la filogenii care sunt mai congruente cu arborele speciilor . Filogenia rezultată a arătat că speciile Alternaria cu spori mari și Alternaria cu spori mici s-au grupat separat în două clade diferite (Fig. 3). Este interesant faptul că cei doi agenți patogeni majori ai culturilor de Brassicae, și anume A. brassicae și A. brassicicola, s-au grupat separat de toate celelalte specii de Alternaria, ceea ce poate indica o traiectorie evolutivă diferită bazată pe preferințele comune ale gazdei acestor două specii.
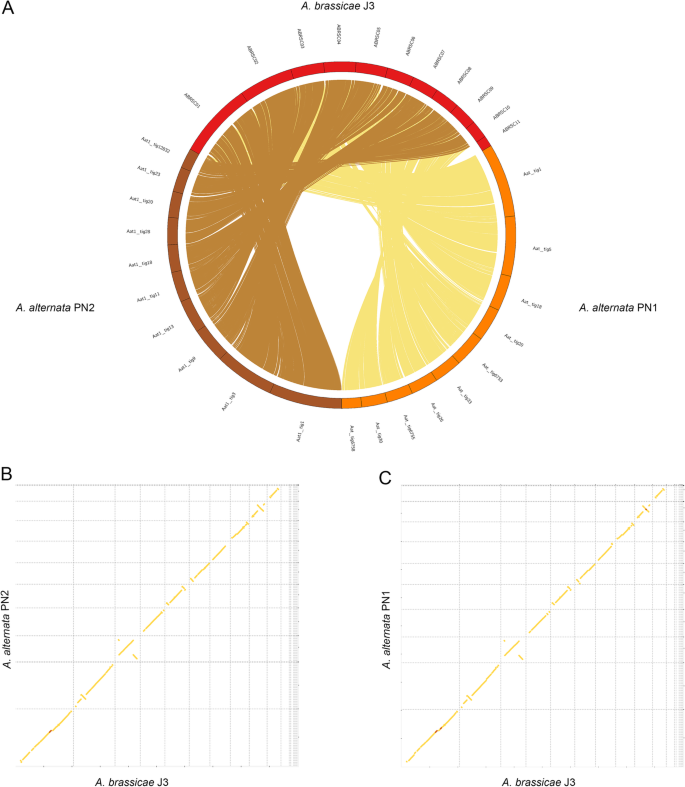
Alinieri ale întregului genom de A. alternata PN1 și PN2 cu A. brassicae. a Circos plot care arată macrosintenia lui A. alternata PN1 și PN2 cu A. brassicae. brassicae în toate contigurile, cu excepția contigurilor dispensabile (ABRSC11, scaffold13,17,18,19), b și c Syntenic dotplots de A. brassicae cu A. alternata PN1 și PN2
Analize comparative ale A. alternata PN1 și PN2 cu A. alternata PN1 și PN2 . alternata izolate obținute din gazde diferite
Am comparat genomurile A. alternata PN1 și PN2 (izolate din B. juncea) cu cel al A. alternata ATCC34957 (izolată din sorg) pentru a identifica orice diferențe în conținutul lor genomic care ar putea permite ca acestea să infecteze două specii foarte diferite. Alinierea genomului integral al A. alternata PN1 și PN2 cu cel al A. alternata ATCC34957 a evidențiat niveluri foarte ridicate de sintenie și absența oricăror regiuni specifice speciilor. Am identificat 719, 152 și, respectiv, 586 de gene specifice izolatelor între cele trei izolate de A. alternata (Fișierul suplimentar 1: tabelul S1). Mai mult de două treimi din genele specifice izolatelor din toate cele trei izolate erau proteine necaracterizate sau nu aveau adnotări. În special, toate cele trei izolate nu conțineau niciun cromozom dispensabil care ar putea conferi patogenitate, așa cum s-a raportat pentru izolatele de A. alternata care infectează multe dintre culturile pomicole, cum ar fi citricele, perele și merele . Repertoriile genetice ale celor trei izolate au constat, de asemenea, într-un număr și tip similar de efectoare, CAZime și grupuri de metaboliți secundari (tabelul 2). În plus, cele două izolate PN1 și PN2 nu provoacă singure simptome de infecție la B. juncea în condiții epifitotice (datele nu sunt prezentate). Rezultatele noastre sugerează că acești izolați de A. alternata (PN1 și PN2) pot fi agenți patogeni facultativi care duc un stil de viață saprofit și pot trece la un stil de viață patogen în anumite condiții de mediu.
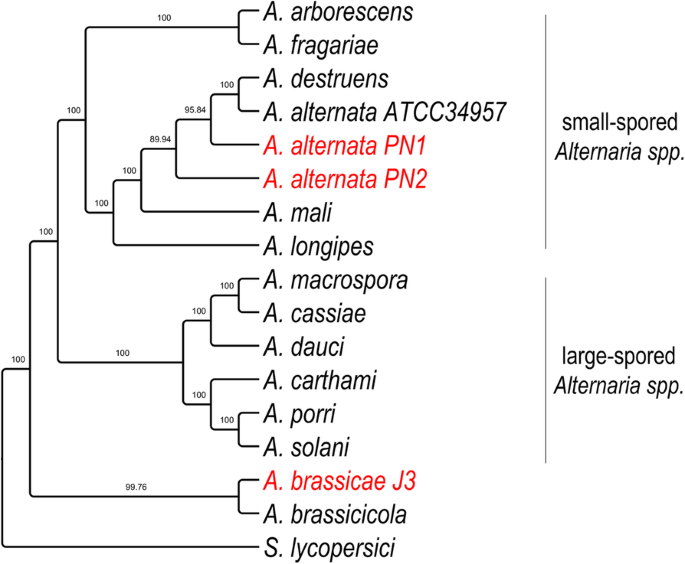
Arbore filogenetic al speciilor de Alternaria cu S. lycopersici ca outgroup. Arborele a fost construit folosind 29 de ortologi cu o singură copie, care au avut cel mai mare semnal filogenetic calculat în Mirlo. Sunt prezentate valorile suportului de ramură din 1000 de replici bootstrap
O abundență de regiuni bogate în repetiții și elemente transpozabile în A. brassicae
Patogenii plantelor filamentoase tind să aibă o arhitectură genomică distinctă cu un conținut mai mare de repetări. Estimarea conținutului de repetări și mascarea cu ajutorul RepeatModeler și RepeatMasker a arătat că genomul A. brassicae a constat din ~ 9,33% de repetări, în comparație cu 2,43 și 2,64% de repetări în genomurile A. alternata. Genomul A. brassicae are cel mai mare conținut de repetări (~ 9,33 %) dintre toate speciile de Alternaria secvențiate până în prezent. Analiza noastră a arătat că conținutul de repetări diferă semnificativ între izolatele de A. alternata și celelalte specii patogene de Alternaria. Speciile patogene de Alternaria, în special A. brassicae și A. brassicicola, au avut un repertoriu considerabil mai mare de elemente LTR/Gypsy și LTR/Copia (> 8X) în comparație cu celelalte izolate de A. alternata (patogene și nepatogene) (Fig. 4) (Fig. 4). Genomurile A. brassicae și A. brassicicola au avut, de asemenea, o suprareprezentare a transpozonilor de ADN, care s-au ridicat la ~ 5% din genom, în comparație cu < 1% la celelalte specii de Alternaria (Fig. 4).

Compararea conținutului de repetiții la șase specii de Alternaria. Mărimea bulelor corespunde (a) procentului de elemente transpozabile (TE) din genom, b numărului de copii ale TE în genom
Această proliferare a ADN-ului repetitiv și evoluția ulterioară a genelor care se suprapun peste aceste regiuni poate fi cheia succesului evolutiv în care acești agenți patogeni au reușit să persiste de-a lungul generațiilor de conflict coevolutiv cu gazdele lor. Proximitatea față de TE-uri expune potențial genele la mutații punctiforme induse de repetiții (RIP) și, prin urmare, la o evoluție accelerată . De asemenea, recombinarea ectopică între TE-uri similare poate duce la noi combinații de gene și, astfel, la creșterea diversității proteinelor sau a metaboliților.
Prezența unui cromozom dispensabil la A. brassicae cu spori mari
Cromozomii specifici de linie (LS) sau cromozomii dispensabili (DC) au fost raportați la mai multe specii fitopatogene, inclusiv la A. alternata. Se știe că DCs la A. alternata conferă izolatului virulență și specificitate de gazdă. Alinierea întregului genom al A. brassicae cu alte Alternaria spp. a arătat că un contig de aproximativ 1 Mb împreună cu alte contig-uri mai mici (66-366 kb) era specific pentru A. brassicae și nu prezenta sintenie cu nicio regiune din alte Alternaria spp. Cu toate acestea, s-a observat o sintenie parțială atunci când contig-ul a fost aliniat cu secvențele altor cromozomi dispensabili raportate la Alternaria spp. Acest lucru ne-a determinat să emitem ipoteza că aceste contig-uri pot reprezenta împreună un DC de A. brassicae. Pentru a confirma acest lucru, am căutat în contig-uri prezența genelor AaMSAS și ALT1, care sunt gene marker cunoscute pentru cromozomii dispensabili la Alternaria spp. Am găsit două copii ale genei AaMSAS ca parte a două clustere de biosinteză a metaboliților secundari pe contig-ul de 1 Mb. Cu toate acestea, nu am găsit niciun omolog al genei ALT1. În plus, conținutul de repetiții al contigurilor (ABRSC11, scheletul 13, 17, 18 și 19) a fost comparat cu cel al întregului genom. Conținutul de gene al contigurilor specifice liniei a fost semnificativ mai mic decât cel al cromozomilor de bază (tabelul 3). În schimb, contigii DC au fost foarte îmbogățiți în conținutul de TE în comparație cu cromozomii nucleu (tabelul 3).
Deși, DC nu a fost îmbogățit cu gene care codifică proteine secretate, proporția de gene efectoare secretate a fost cu 30% mai mare în comparație cu cromozomii nucleu. Toate dovezile de mai sus indică faptul că A. brassicae poate într-adevăr să adăpostească un DC. Până în prezent, au fost raportate DC-uri la Alternaria spp. numai la Alternaria spp. cu spori mici și nu se cunoaște nicio specie de Alternaria cu spori mari care să găzduiască DC-uri. Rămâne de văzut dacă DC contribuie la virulența lui A. brassicae. Studiile viitoare ar implica caracterizarea cromozomului dispensabil la A. brassicae și corelarea prezenței sale cu patogenitatea diferitelor izolate.
Analiza ortologică dezvăluie gene specifice speciilor cu roluri putative în virulență
Diferențele în conținutul genelor și diversitatea în cadrul genelor contribuie la adaptare, creștere și patogenitate. Pentru a cataloga diferențele în ceea ce privește conținutul de gene în cadrul genului Alternaria și al Dothideomycetes, am efectuat o analiză ortologică asupra setului combinat de 3 60 216 proteine din 30 de specii diferite (inclusiv 16 specii de Alternaria) aparținând Dothideomycetes (Fișier suplimentar 2: Tabelul S2), din care 3 45 321 de proteine au putut fi atribuite cel puțin unuia dintre ortogrupuri. Am identificat 460 de gene specifice A. brassicae care erau prezente în A. brassicae, dar absente în toate celelalte specii de Alternaria (Fișier suplimentar 3: Tabelul S3). Aceste gene specifice speciilor au inclus 35 de gene codificatoare de proteine secretate, dintre care 11 au fost prezise ca fiind efectoare. În plus, 20 dintre aceste gene specifice speciilor erau prezente pe DC. Un număr mare dintre aceste proteine au aparținut categoriei de proteine necaracterizate fără funcție cunoscută. Pentru a testa dacă aceste gene specifice speciilor sunt rezultatul unei evoluții adaptative care are loc în regiunile bogate în repetări ale genomului, am efectuat un test de permutare pentru a compara suprapunerea regiunilor bogate în repetări și a elementelor transpozabile cu un set de gene aleatorii cu suprapunerea acestor gene specifice speciilor. Am constatat că aceste gene specifice speciilor se suprapun în mod semnificativ cu regiunile bogate în repetări (valoare P: 9,99e-05; Z-score: – 4,825) și cu elementele transpozabile (valoare P: 0,0460; Z-score: 2,539) din genom.
Profilul de metaboliți secundari al A. brassicae și asocierea sa cu elementele transpozabile (TEs)
Genurile Alternaria și Cochliobolus sunt cunoscute ca fiind principalii producători de toxine de metaboliți secundari specifici gazdei. Alternaria spp. în special sunt cunoscute pentru producția de metaboliți secundari diversificați din punct de vedere chimic, care includ toxine specifice gazdei (HST) și non-HST. Acești metaboliți secundari sunt, de obicei, generați de sintetaze peptidice non-ribosomale (NRPS) și de sintetaze de poliketide (PKS). Am identificat cinci clustere de gene SM de tip NRPS, 12 clustere de gene de tip PKS și șapte clustere de gene asemănătoare terpenelor în A. brassicae (Fișier suplimentar 4: Tabelul S4). Dintre cele cinci clustere NRPS, am putut identifica trei clustere care produc metaboliți secundari cunoscuți, și anume Destruxina B, HC-toxina și dimetilcoprogenul (siderofor) cu roluri cunoscute în virulență.
Clusterul de gene responsabil pentru producția de dimetilcoprogen (siderofor) la A. brassicae este format din 22 de gene, inclusiv principalele gene biosintetice, oxidoreductaze și transportatori de siderofor. Sideroforii sunt compuși de chelare a fierului, utilizați de ciuperci pentru a dobândi fierul feric extracelular și despre care s-a raportat că sunt implicați în virulența fungică. Identificarea grupului de gene responsabile de sinteza sideroforilor ar permite studierea sideroforilor și a rolului lor în patogenitatea la A. brassicae. În plus, a fost identificat, de asemenea, un cluster de tip PKS format din 12 gene, responsabil pentru producția de melanină (Fișier suplimentar 4: Tabelul S4). Clusterul de biosinteză a melaninei a fost descris anterior pentru A. alternata . De asemenea, factorul de transcripție Amr1, care induce producția de melanină, a fost caracterizat la A. brassicicola și se știe că suprimă virulența . Cu toate acestea, rolul melaninei în virulență este ambiguu și specific speciilor .
Patogenii vegetali aparținând genului Alternaria par să aibă o capacitate dinamică de a dobândi un nou potențial de metaboliți secundari pentru a coloniza noi nișe ecologice. Cea mai parcimonioasă explicație pentru această achiziție dinamică a potențialului de metaboliți secundari este transferul orizontal de gene în cadrul genului Alternaria și, posibil, cu alte genuri. Există numeroase dovezi în literatura de specialitate conform cărora o mare parte din HST-urile din Alternaria sunt transportate pe cromozomii dispensabili, iar schimbul acestor cromozomi poate extinde specificitatea gazdei. Am identificat, de asemenea, un grup NRPS, posibil să codifice pentru HC-toxina într-unul dintre DC (scheletul 18) (Fișier suplimentar 4: Tabelul S4). HC-toxina este un determinant de virulență cunoscut al plantei patogene Cochliobolus carbonum, care infectează genotipurile de porumb cărora le lipsește o copie funcțională a HM1, o carbonilreductază care detoxifică toxina . Un raport recent a arătat că A. jesenskae poate produce, de asemenea, HC-toxină, fiind singura ciupercă, în afară de C. carbonum, care produce această toxină. Prezența clusterului de gene HC-toxină, un determinant de virulență la C. carbonum, într-un DC de A. brassicae indică faptul că transferul orizontal de gene interspecii poate fi mai comun decât se așteaptă.
Pe lângă transferul orizontal de gene, duplicarea rapidă, divergența și pierderea genelor SM pot contribui, de asemenea, la dezvoltarea de noi capacități metabolice ale agentului patogen. Aceste procese de duplicare și divergență ar putea fi ajutate de proximitatea clusterelor de metaboliți secundari față de elementele repetate, care le face predispuse la mutații RIP. Prin urmare, am testat dacă grupurile de metaboliți secundari au fost, de asemenea, asociate cu regiunile bogate în repetări. A fost utilizat un test de permutare pentru a compara suprapunerea regiunilor bogate în repetări cu un set de gene aleatorii cu suprapunerea genelor clusterului de metaboliți secundari. Clusterele de metaboliți secundari s-au suprapus în mod semnificativ peste regiunile bogate în repetiții în comparație cu setul de gene aleatorii (valoare P: 0,0017; scor Z: – 2,7963). De asemenea, aceste clustere s-au suprapus în mod semnificativ cu elementele transpozabile din regiunile bogate în repetări (valoare P: 0,0087; Z-score: 2,9871). Acest lucru arată că ambele mecanisme descrise mai sus pentru dobândirea unui nou potențial de metaboliți secundari pot fi posibile în cazul A. brassicae. Analizele la scară de populație la nivel de specie și gen ar putea arunca lumină asupra prevalenței acestor mecanisme în cadrul genului Alternaria.
Analiza de sinteză dezvăluie baza genetică a exclusivității producerii Destruxinei B de către A. brassicae în cadrul genului Alternaria
Destruxina B reprezintă o clasă de depsipeptide ciclice care este cunoscută ca fiind unul dintre factorii cheie de patogenitate ai A. brassicae și a fost raportată ca fiind o toxină specifică gazdei de A. brassicae . Destruxina B nu a fost raportată ca fiind produsă de niciuna dintre celelalte specii de Alternaria. Aici raportăm pentru prima dată clusterele de gene biosintetice responsabile de producerea Destruxinei B la A. brassicae. Clusterul este format din 10 gene, inclusiv principala enzimă biosintetică codificată de o genă NRPS (DtxS1) și enzima de limitare a vitezei, DtxS3 (aldo-cetoreductază) (Fișier suplimentar 4: tabelul S4). În mod interesant, analiza de sintenie a acestui grup între cele șase specii de Alternaria a arătat că ambele gene nu erau prezente în niciuna dintre celelalte specii de Alternaria spp. deși sintenia generală a grupului a fost menținută la toate aceste specii (Fig. 5). Absența genelor cheie care codifică enzimele DtxS1 și DtxS3 din clusterul Destruxina B în celelalte specii de Alternaria explică absența Destruxinei B la aceste specii.

Sinteza clusterului Destruxina B în cele șase specii de Alternaria. DtxS1 și DtxS3 sunt marcate cu roșu (etichetate cu ID-urile genelor respective) și sunt absente la toate celelalte specii de Alternaria
Destruxina B a fost raportată la entomopatogenul Metarhizium robertsii , și la Ophiosphaerella herpotricha , cauza apariției petelor moarte de primăvară la iarba de bermudă, în afară de A. brassicae. Wang și colab. au descris grupul de biosinteză a metaboliților secundari responsabili de producția de destruxină la M. robertsii . Clusterul Destruxin din speciile de Metarhizhium constă în două gene cheie suplimentare, și anume DtxS2 și DtxS4. DtxS2 este responsabilă de conversia Destruxinei B în Destruxina A, C, D și E. Nu am putut găsi nicio genă sintenică în cluster care să corespundă cu DtxS2 sau DtxS4 la A. brassicae. Se știe că A. brassicae produce numai Destruxina B, nu și celelalte forme de Destruxină, ceea ce ar putea explica absența DtxS2 la A. brassicae. DtxS4 este o aspartat decarboxilază care este necesară pentru producerea de β-alanină din acid aspartic și este esențială pentru producerea structurii vertebrale a Destruxinei. Mai mult, mutanții de deleție ai DtxS4 au fost capabili să producă Destruxin atunci când au fost suplimentați cu β-alanină în mediu. Chiar dacă DtxS4 nu este prezent în clusterul Destruxin, A. brassicae are o copie funcțională a aspartat decarboxilazei prezentă în altă parte a genomului și, prin urmare, este posibil să poată asigura aprovizionarea cu β-alanină pentru sinteza Destruxin B. Istoria evolutivă a producției de Destruxin B în cadrul genului Metarhizhium se aseamănă foarte mult cu cea a Alternaria. Agenții patogeni specializați, cum ar fi M. acridum, nu au cele două enzime cheie și, prin urmare, nu produc destruxine. Cu toate acestea, M. robertsii, un generalist cu o gamă mai largă de gazde, produce destruxine. Prin urmare, s-a emis ipoteza că Destruxinele ar putea fi responsabile pentru stabilirea gamei extinse de gazde a lui M. roberstii. O ipoteză similară poate fi valabilă și în cazul lui A. brassicae, care are o gamă largă de gazde și poate infecta aproape toate Brassicaceae. Experimente suplimentare pentru a determina gama de gazde a mutanților de A. brassicae nulă pentru Destruxin ar putea ajuta la clarificarea rolului acestei importante fitotoxine în extinderea gamei de gazde a A. brassicae.
Profile CAZyme distincte ale A. brassicae și A. brassicicola în cadrul genului Alternaria
CAZimele (Carbohydrate-Active enZymes) sunt proteine implicate în degradarea, rearanjarea sau sinteza legăturilor glicozidice. Agenții patogeni ai plantelor secretă o gamă diversă de CAZime care descompun polizaharidele complexe din peretele celular al plantelor. Acestea sunt alcătuite din cinci clase distincte, și anume: glicozid hidrolaze (GH), glicoziltransferaze (GT), liaze polizaharidice (PL), esteraze de carbohidrați (CE) și module de legare a carbohidraților (CBM). Am identificat > 500 de CAZime în cele șase Alternaria spp. inclusiv A. brassicae (Fișier suplimentar 5: Tabelul S5). Distribuția CAZimelor de la A. brassicae și A. brassicicola a variat față de cele de la celelalte Alternaria spp. formând astfel un grup separat (Fig. 6). Numărul de enzime de activitate auxiliară sau de enzime implicate în degradarea peretelui celular vegetal a variat considerabil între diferitele specii comparate. Aproape 46% din CAZimele din A. brassicae au fost secretate din care ~ 17% au fost prezise a fi efectoare.

Harta termică a profilurilor CAZimelor la 16 specii cu genul Alternaria. Gruparea organismelor se aseamănă foarte mult cu cea a filogeniei speciilor, cu excepția A. brassicae și A. brassicicola, care se grupează separat cu outgroupul Stemphylium lycopersici. CAZimele au fost grupate în conformitate cu clasificarea CAZy
Familia AA9 (anterior GH61; monooxigenaze polizaharidice litice dependente de cupru (LPMO)) este semnificativ extinsă în comparație cu celelalte familii CAZy din genul Alternaria, fiecare specie conținând > 20 de copii ale genei. Numărul de copii la Alternaria spp. este mult mai mare decât numărul de copii raportat pentru Botrytis și Fusarium spp. Familia AA9 este implicată în degradarea polizaharidelor din peretele celular și se știe că acționează asupra unei serii de polizaharide, inclusiv amidonul, xiloglucanul, cellodextrinele și glucomananul. S-a emis ipoteza că LPMO au un dublu rol – descompunerea directă a polizaharidelor din peretele celular și rolul de generator de ROS, contribuind astfel la stresul oxidativ care duce la necroza țesuturilor vegetale. În mod surprinzător, 11 dintre cele 26 de proteine AA9 prezente la A. brassicae sunt prezise a fi efectoare secretate. Caracterizarea acestor CAZime și a rolului lor în patogeneză ar putea face obiectul unor studii suplimentare.
Profiluri de efectori care se suprapun în cadrul genului Alternaria
Am prezis repertoriul de efectori a șase specii de Alternaria (tabelul 2), inclusiv A. brassicae, utilizând conducta descrisă în secțiunea metode. Cele mai multe dintre proteinele secretate mici prezise a fi efectoare au fost proteine necaracterizate/proteine prezise și, prin urmare, ar putea fi efectoare noi specifice speciei sau genului. A. brassicae a avut cea mai mare proporție de proteine necunoscute/prevăzute/hipotetice în setul de efectori candidați. Am constatat că cei mai mulți dintre efectorii dintre cele șase specii sunt comuni și se suprapun, sugerând că mecanismele largi de patogeneză care implică efectorii proteici pot fi conservate în cadrul genului. Cu toate acestea, am descoperit că două copii ale unei proteine care conține un pliu de beta/gamma-cristalină sunt prezente numai la A. brassicae și A. brassicicola, dar nu și la celelalte patru specii de Alternaria. O căutare suplimentară în baza de date nr. din NCBI a arătat că această proteină era complet absentă în genul Alternaria, iar cele mai apropiate corespondențe au fost găsite în alte specii de plante patogene, și anume Macrophomina și Fusarium. Cu toate acestea, nu sunt disponibile informații cu privire la funcția sau rolul său în patogenitate la niciuna dintre specii.
Am putut stabili, de asemenea, că unii dintre efectorii din A. brassicae au potențialul de a evolua în mod adaptiv, deoarece au fost, de asemenea, asociate în mod semnificativ cu regiunile bogate în repetări ale genomului (valoare P: 0,0003; Z-score: – 2,8778). Prin urmare, sunt necesare analize la nivel de populație pentru a identifica efectorii supuși selecției pozitive, care ar putea aduce lumină asupra evoluției patogenității la A. brassicae. Efectorii identificați în acest studiu dezvăluie gama largă de proteine care pot fi implicate în patogeneza A. brassicae. 39 dintre acești efectori au fost prezise ca fiind CAZime care au diferite roluri în degradarea peretelui celular și a polizaharidelor asociate. Genomul lui A. brassicae conținea două proteine peptidice inducătoare de necroză și etilenă, care au fost implicate în patogeneza diferiților agenți patogeni, inclusiv a oomocetelor și a necrotrofilor. S-a demonstrat recent că Hrip1 (Hypersensitive response inducing protein 1) din A. alternata este recunoscută de MdNLR16 într-o manieră clasică de gen pentru gen, iar ștergerea Hrip1 din A. alternata îi sporește virulența . Un omolog Hrip1 este, de asemenea, prezent în A. brassicae, dar nu se preconizează că este secretat în afara celulei, deși acest lucru trebuie verificat experimental. Prezența unor efectori care sunt recunoscuți într-o manieră de genă pentru genă deschide posibilitatea identificării unor gene R complementare în gazdă care pot fi utilizate pentru dezvoltarea de soiuri sau cultivare rezistente.
.